+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of PqqU with ligand PQQ | |||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of E. coli PqqU with ligand PQQ at 1.99 A global resolution. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | TonD-dependent / outer membrane / transporter / PQQ uptake / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報siderophore transmembrane transport / intracellular monoatomic cation homeostasis / siderophore uptake transmembrane transporter activity / transmembrane transporter complex / cell outer membrane / signaling receptor activity / membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.99 Å | |||||||||||||||
 データ登録者 データ登録者 | Munder F / Venugopal H / Grinter R | |||||||||||||||
| 資金援助 |  オーストラリア, 4件 オーストラリア, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: High-affinity PQQ import is widespread in Gram-negative bacteria. 著者: Munder F / Voutsinos M / Hantke K / Venugopal H / Grinter R | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_45192.map.gz emd_45192.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-45192-v30.xml emd-45192-v30.xml emd-45192.xml emd-45192.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_45192_fsc.xml emd_45192_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_45192.png emd_45192.png | 189.4 KB | ||
| Filedesc metadata |  emd-45192.cif.gz emd-45192.cif.gz | 6.4 KB | ||
| その他 |  emd_45192_half_map_1.map.gz emd_45192_half_map_1.map.gz emd_45192_half_map_2.map.gz emd_45192_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-45192 http://ftp.pdbj.org/pub/emdb/structures/EMD-45192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-45192 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_45192_validation.pdf.gz emd_45192_validation.pdf.gz | 943.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_45192_full_validation.pdf.gz emd_45192_full_validation.pdf.gz | 942.7 KB | 表示 | |
| XML形式データ |  emd_45192_validation.xml.gz emd_45192_validation.xml.gz | 18.9 KB | 表示 | |
| CIF形式データ |  emd_45192_validation.cif.gz emd_45192_validation.cif.gz | 24.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45192 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-45192 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9c4oMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_45192.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_45192.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of E. coli PqqU with ligand PQQ at 1.99 A global resolution. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map B of PqqU with ligand PQQ.
| ファイル | emd_45192_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B of PqqU with ligand PQQ. | ||||||||||||
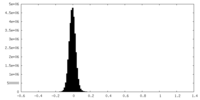
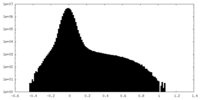
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A of PqqU with ligand PQQ.
| ファイル | emd_45192_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A of PqqU with ligand PQQ. | ||||||||||||
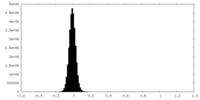
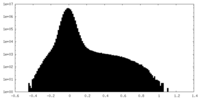
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of PqqU with PQQ
| 全体 | 名称: Complex of PqqU with PQQ |
|---|---|
| 要素 |
|
-超分子 #1: Complex of PqqU with PQQ
| 超分子 | 名称: Complex of PqqU with PQQ / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: TonB-dependent outer membrane transporter PqqU in complex with its substrate PQQ. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 78 KDa |
-分子 #1: Pyrroloquinoline quinone transporter
| 分子 | 名称: Pyrroloquinoline quinone transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 78.401164 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSNHHHHHHH HHHENLYFQG AMDIGINSDP ADEQTMIVSA APQVVSELDT PAAVSVVDGE EMRLATPRIN LSESLTGVPG LQVQNRQNY AQDLQLSIRG FGSRSTYGIR GIRLYVDGIP ATMPDGQGQT SNIDLSSVQN VEVLRGPFSA LYGNASGGVM N VTTQTGQQ ...文字列: MSNHHHHHHH HHHENLYFQG AMDIGINSDP ADEQTMIVSA APQVVSELDT PAAVSVVDGE EMRLATPRIN LSESLTGVPG LQVQNRQNY AQDLQLSIRG FGSRSTYGIR GIRLYVDGIP ATMPDGQGQT SNIDLSSVQN VEVLRGPFSA LYGNASGGVM N VTTQTGQQ PPTIEASSYY GSFGSWRYGL KATGATGDGT QPGDVDYTVS TTRFTTHGYR DHSGAQKNLA NAKLGVRIDE AS KLSLIFN SVDIKADDPG GLTKAEWKAN PQQAPRAEQY DTRKTIKQTQ AGLRYERSLS SRDDMSVMMY AGERETTQYQ SIP MAPQLN PSHAGGVITL QRHYQGIDSR WTHRGELGVP VTFTTGLNYE NMSENRKGYN NFRLNSGMPE YGQKGELRRD ERNL MWNID PYLQTQWQLS EKLSLDAGVR YSSVWFDSND HYVTPGNGDD SGDASYHKWL PAGSLKYAMT DAWNIYLAAG RGFET PTIN ELSYRADGQS GMNLGLKPST NDTIEIGSKT RIGDGLLSLA LFQTDTDDEI VVDSSSGGRT TYKNAGKTRR QGAELA WDQ RFAGDFRVNA SWTWLDATYR SNVCNEQDCN GNRMPGIARN MGFASIGYVP EDGWYAGTEA RYMGDIMADD ENTAKAP SY TLVGLFTGYK YNYHNLTVDL FGRVDNLFDK EYVGSVIVNE SNGRYYEPSP GRNYGVGMNI AWRFE UniProtKB: Pyrroloquinoline quinone transporter |
-分子 #2: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #3: PYRROLOQUINOLINE QUINONE
| 分子 | 名称: PYRROLOQUINOLINE QUINONE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: PQQ |
|---|---|
| 分子量 | 理論値: 330.206 Da |
| Chemical component information | 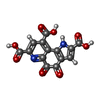 ChemComp-PQQ: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 111 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: LMNG was partly removed by concentration in a 100 kDa cut-off concentrator. | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 30 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: 3 microliters of sample. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOCONTINUUM (6k x 4k) 実像数: 8744 / 平均露光時間: 5.22 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 Z
Z Y
Y X
X