+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CCHFV GP38 bound to ADI-46152 and ADI-58048 Fabs | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CCHFV / GP38 / Antibody / Immunology / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / clathrin-dependent endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Crimean-Congo hemorrhagic fever virus (ウイルス) / Crimean-Congo hemorrhagic fever virus (ウイルス) /  Crimean-Congo hemorrhagic fever virus strain IbAr10200 (ウイルス) Crimean-Congo hemorrhagic fever virus strain IbAr10200 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Hjorth CK / McLellan JS | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2024 ジャーナル: Cell Rep / 年: 2024タイトル: Crimean-Congo hemorrhagic fever survivors elicit protective non-neutralizing antibodies that target 11 overlapping regions on glycoprotein GP38. 著者: Olivia S Shin / Stephanie R Monticelli / Christy K Hjorth / Vladlena Hornet / Michael Doyle / Dafna Abelson / Ana I Kuehne / Albert Wang / Russell R Bakken / Akaash K Mishra / Marissa ...著者: Olivia S Shin / Stephanie R Monticelli / Christy K Hjorth / Vladlena Hornet / Michael Doyle / Dafna Abelson / Ana I Kuehne / Albert Wang / Russell R Bakken / Akaash K Mishra / Marissa Middlecamp / Elizabeth Champney / Lauran Stuart / Daniel P Maurer / Jiannan Li / Jacob Berrigan / Jennifer Barajas / Stephen Balinandi / Julius J Lutwama / Leslie Lobel / Larry Zeitlin / Laura M Walker / John M Dye / Kartik Chandran / Andrew S Herbert / Noel T Pauli / Jason S McLellan /   要旨: Crimean-Congo hemorrhagic fever virus can cause lethal disease in humans yet there are no approved medical countermeasures. Viral glycoprotein GP38, exclusive to Nairoviridae, is a target of ...Crimean-Congo hemorrhagic fever virus can cause lethal disease in humans yet there are no approved medical countermeasures. Viral glycoprotein GP38, exclusive to Nairoviridae, is a target of protective antibodies and is a key antigen in preclinical vaccine candidates. Here, we isolate 188 GP38-specific antibodies from human survivors of infection. Competition experiments show that these antibodies bind across 5 distinct antigenic sites, encompassing 11 overlapping regions. Additionally, we show structures of GP38 bound with 9 of these antibodies targeting different antigenic sites. Although these GP38-specific antibodies are non-neutralizing, several display protective efficacy equal to or better than murine antibody 13G8 in two highly stringent rodent models of infection. Together, these data expand our understanding regarding this important viral protein and may inform the development of broadly effective CCHFV antibody therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43604.map.gz emd_43604.map.gz | 259.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43604-v30.xml emd-43604-v30.xml emd-43604.xml emd-43604.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43604_fsc.xml emd_43604_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43604.png emd_43604.png | 53.4 KB | ||
| Filedesc metadata |  emd-43604.cif.gz emd-43604.cif.gz | 6.6 KB | ||
| その他 |  emd_43604_half_map_1.map.gz emd_43604_half_map_1.map.gz emd_43604_half_map_2.map.gz emd_43604_half_map_2.map.gz | 254.9 MB 254.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43604 http://ftp.pdbj.org/pub/emdb/structures/EMD-43604 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43604 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43604 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43604_validation.pdf.gz emd_43604_validation.pdf.gz | 849.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43604_full_validation.pdf.gz emd_43604_full_validation.pdf.gz | 848.8 KB | 表示 | |
| XML形式データ |  emd_43604_validation.xml.gz emd_43604_validation.xml.gz | 22.8 KB | 表示 | |
| CIF形式データ |  emd_43604_validation.cif.gz emd_43604_validation.cif.gz | 29.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43604 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43604 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43604 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43604 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8vwwMC  8vvkC  8vvlC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43604.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43604.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8332 Å | ||||||||||||||||||||||||||||||||||||
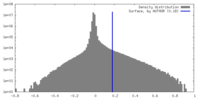
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A
| ファイル | emd_43604_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
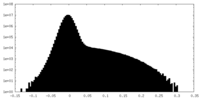
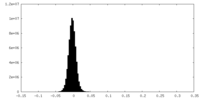
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_43604_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
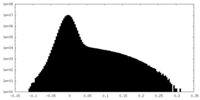
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CCHFV GP38 bound with ADI-46152 and ADI-58048 Fabs
| 全体 | 名称: CCHFV GP38 bound with ADI-46152 and ADI-58048 Fabs |
|---|---|
| 要素 |
|
-超分子 #1: CCHFV GP38 bound with ADI-46152 and ADI-58048 Fabs
| 超分子 | 名称: CCHFV GP38 bound with ADI-46152 and ADI-58048 Fabs / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: ADI-46152 Fab
| 超分子 | 名称: ADI-46152 Fab / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: CCHFV GP38
| 超分子 | 名称: CCHFV GP38 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Crimean-Congo hemorrhagic fever virus (ウイルス) Crimean-Congo hemorrhagic fever virus (ウイルス)株: IbAr10200 |
-超分子 #4: ADI-58048 Fab
| 超分子 | 名称: ADI-58048 Fab / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #4-#5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: GP38
| 分子 | 名称: GP38 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Crimean-Congo hemorrhagic fever virus strain IbAr10200 (ウイルス) Crimean-Congo hemorrhagic fever virus strain IbAr10200 (ウイルス)株: IbAr10200 |
| 分子量 | 理論値: 30.241828 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: NLKMEIILTL SQGLKKYYGK ILRLLQLTLE EDTEGLLEWC KRNLGLDCDD TFFQKRIEEF FITGEGHFNE VLQFRTPGTL STTESTPAG LPTAEPFKSY FAKGFLSIDS GYYSAKCYSG TSNSGLQLIN ITRHSTRIVD TPGPKITNLK TINCINLKAS I FKEHREVE ...文字列: NLKMEIILTL SQGLKKYYGK ILRLLQLTLE EDTEGLLEWC KRNLGLDCDD TFFQKRIEEF FITGEGHFNE VLQFRTPGTL STTESTPAG LPTAEPFKSY FAKGFLSIDS GYYSAKCYSG TSNSGLQLIN ITRHSTRIVD TPGPKITNLK TINCINLKAS I FKEHREVE INVLLPQVAV NLSNCHVVIK SHVCDYSLDI DGAVRLPHIY HEGVFIPGTY KIVIDKKNKL NDRCTLFTDC VI KGREVRK GQSVLRQYKT EIRIGKASTG S UniProtKB: Envelopment polyprotein |
-分子 #2: ADI-46152 Fab Heavy Chain
| 分子 | 名称: ADI-46152 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.370311 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVESGGV LVQPGGSLRL SCAASGFTVN SNYMTWVRQA PGKGLEWVSV IYSGGYTYYA DSVKGRFAIS RDNSKNTVYL QMNSLRVED TAVYYCARLR LSSSWYPEAF DYWGQGTLVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV T VSWNSGAL ...文字列: EVQLVESGGV LVQPGGSLRL SCAASGFTVN SNYMTWVRQA PGKGLEWVSV IYSGGYTYYA DSVKGRFAIS RDNSKNTVYL QMNSLRVED TAVYYCARLR LSSSWYPEAF DYWGQGTLVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV T VSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK KVEPKSCDKG |
-分子 #3: ADI-46152 Fab Light Chain
| 分子 | 名称: ADI-46152 Fab Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.957463 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AILLTQSPSS LSASVGDRVT ITCRASQGIS SALAWYQQKP GRAPKVLIYD ASSLANGVPS RFSGSGSGTD FTLTINSLQP EDFATYYCQ QFNYYPLTFG GGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: AILLTQSPSS LSASVGDRVT ITCRASQGIS SALAWYQQKP GRAPKVLIYD ASSLANGVPS RFSGSGSGTD FTLTINSLQP EDFATYYCQ QFNYYPLTFG GGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTQ GTTSVTKSFN RGEC |
-分子 #4: ADI-58048 Fab Heavy Chain
| 分子 | 名称: ADI-58048 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.790645 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QLQLQESGPG LVKPSETLSL TCTVSGGSIT TSHYYWGWIR QPPGKGLEWV GSMYYSGGIY YNPSLKGRVT ISVDTSKNQF SLKLSSVTA ADTAVYYCAL ADAPDDAFDI WGQGTMVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS ...文字列: QLQLQESGPG LVKPSETLSL TCTVSGGSIT TSHYYWGWIR QPPGKGLEWV GSMYYSGGIY YNPSLKGRVT ISVDTSKNQF SLKLSSVTA ADTAVYYCAL ADAPDDAFDI WGQGTMVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV S WNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EPKSCDKG |
-分子 #5: ADI-58048 Fab Light Chain
| 分子 | 名称: ADI-58048 Fab Light Chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.36591 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSAFVGDRVT ITCRASQSIS SYLNWYQQKP GKAPKLLIYA ASTLQSGVPS RFSGSGSGTD FTLTISSLQS EDFATYYCQ ESYSIPFTFG PGTKVDIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSAFVGDRVT ITCRASQSIS SYLNWYQQKP GKAPKLLIYA ASTLQSGVPS RFSGSGSGTD FTLTISSLQS EDFATYYCQ ESYSIPFTFG PGTKVDIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3647 / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||||||||
| 得られたモデル |  PDB-8vww: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)