+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Herpes simplex virus 1 polymerase holoenzyme bound to mismatched DNA in editing conformation | |||||||||
 マップデータ マップデータ | sharpened map after 3D classification based on DNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | herpes simplex virus / replication / DNA polymerase holoenzyme / editing conformation / TRANSFERASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral DNA genome replication / DNA-templated DNA replication / RNA-DNA hybrid ribonuclease activity / DNA replication / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / nucleotide binding / host cell nucleus / DNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) / Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) /   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) / synthetic construct (人工物) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) / synthetic construct (人工物) | |||||||||
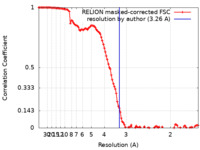
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | |||||||||
 データ登録者 データ登録者 | Pan J / Abraham J / Coen DM / Shankar S / Yang P / Hogle J | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Viral DNA polymerase structures reveal mechanisms of antiviral drug resistance. 著者: Sundaresh Shankar / Junhua Pan / Pan Yang / Yuemin Bian / Gábor Oroszlán / Zishuo Yu / Purba Mukherjee / David J Filman / James M Hogle / Mrinal Shekhar / Donald M Coen / Jonathan Abraham /    要旨: DNA polymerases are important drug targets, and many structural studies have captured them in distinct conformations. However, a detailed understanding of the impact of polymerase conformational ...DNA polymerases are important drug targets, and many structural studies have captured them in distinct conformations. However, a detailed understanding of the impact of polymerase conformational dynamics on drug resistance is lacking. We determined cryoelectron microscopy (cryo-EM) structures of DNA-bound herpes simplex virus polymerase holoenzyme in multiple conformations and interacting with antivirals in clinical use. These structures reveal how the catalytic subunit Pol and the processivity factor UL42 bind DNA to promote processive DNA synthesis. Unexpectedly, in the absence of an incoming nucleotide, we observed Pol in multiple conformations with the closed state sampled by the fingers domain. Drug-bound structures reveal how antivirals may selectively bind enzymes that more readily adopt the closed conformation. Molecular dynamics simulations and the cryo-EM structure of a drug-resistant mutant indicate that some resistance mutations modulate conformational dynamics rather than directly impacting drug binding, thus clarifying mechanisms that drive drug selectivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42889.map.gz emd_42889.map.gz | 117.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42889-v30.xml emd-42889-v30.xml emd-42889.xml emd-42889.xml | 31 KB 31 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42889_fsc.xml emd_42889_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42889.png emd_42889.png | 74.5 KB | ||
| Filedesc metadata |  emd-42889.cif.gz emd-42889.cif.gz | 8.4 KB | ||
| その他 |  emd_42889_additional_1.map.gz emd_42889_additional_1.map.gz emd_42889_additional_2.map.gz emd_42889_additional_2.map.gz emd_42889_half_map_1.map.gz emd_42889_half_map_1.map.gz emd_42889_half_map_2.map.gz emd_42889_half_map_2.map.gz | 98.7 MB 116.6 MB 98.6 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42889 http://ftp.pdbj.org/pub/emdb/structures/EMD-42889 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42889 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42889 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42889_validation.pdf.gz emd_42889_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42889_full_validation.pdf.gz emd_42889_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_42889_validation.xml.gz emd_42889_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_42889_validation.cif.gz emd_42889_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42889 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42889 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42889 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42889 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42889.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42889.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map after 3D classification based on DNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HSV-1 DNA polymerase holoenzyme editing complex
| 全体 | 名称: HSV-1 DNA polymerase holoenzyme editing complex |
|---|---|
| 要素 |
|
-超分子 #1: HSV-1 DNA polymerase holoenzyme editing complex
| 超分子 | 名称: HSV-1 DNA polymerase holoenzyme editing complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Herpes simplex virus type 1 polymerase holoenzyme UL30:UL42 in complex with primer DNA that has a 3'-end mismatch with template DNA |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) Human herpesvirus 1 (strain KOS) (ヘルペスウイルス) |
| 分子量 | 理論値: 197 KDa |
-分子 #1: DNA polymerase
| 分子 | 名称: DNA polymerase / タイプ: protein_or_peptide / ID: 1 詳細: Herpes simplex virus type 1 (KOS strain) DNA polymerase catalytic subunit UL30 with its N-terminal 42 residues deleted and replaced by an N-terminal poly-histidine tag in the expression construct コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) |
| 分子量 | 理論値: 133.614344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHNFYN PYLAPVGTQQ KPTGPTQRHT YYSECDEFRF IAPRVLDEDA PPEKRAGVHD GHLKRAPKVY CGGDERDVLR VGSGGFWPR RSRLWGGVDH APAGFNPTVT VFHVYDILEN VEHAYGMRAA QFHARFMDAI TPTGTVITLL GLTPEGHRVA V HVYGTRQY ...文字列: HHHHHHNFYN PYLAPVGTQQ KPTGPTQRHT YYSECDEFRF IAPRVLDEDA PPEKRAGVHD GHLKRAPKVY CGGDERDVLR VGSGGFWPR RSRLWGGVDH APAGFNPTVT VFHVYDILEN VEHAYGMRAA QFHARFMDAI TPTGTVITLL GLTPEGHRVA V HVYGTRQY FYMNKEEVDR HLQCRAPRDL CERMAAALRE SPGASFRGIS ADHFEAEVVE RTDVYYYETR PALFYRVYVR SG RVLSYLC DNFCPAIKKY EGGVDATTRF ILDNPGFVTF GWYRLKPGRN NTLAQPRAPM AFGTSSDVEF NCTADNLAIE GGM SDLPAY KLMCFDIECK AGGEDELAFP VAGHPEDLVI QISCLLYDLS TTALEHVLLF SLGSCDLPES HLNELAARGL PTPV VLEFD SEFEMLLAFM TLVKQYGPEF VTGYNIINFD WPFLLAKLTD IYKVPLDGYG RMNGRGVFRV WDIGQSHFQK RSKIK VNGM VNIDMYGIIT DKIKLSSYKL NAVAEAVLKD KKKDLSYRDI PAYYATGPAQ RGVIGEYCIQ DSLLVGQLFF KFLPHL ELS AVARLAGINI TRTIYDGQQI RVFTCLLRLA DQKGFILPDT QGRFRGAGGE APKRPAAARE DEERPEEEGE DEDEREE GG GEREPEGARE TAGRHVGYQG ARVLDPTSGF HVNPVVVFDF ASLYPSIIQA HNLCFSTLSL RADAVAHLEA GKDYLEIE V GGRRLFFVKA HVRESLLSIL LRDWLAMRKQ IRSRIPQSSP EEAVLLDKQQ AAIKVVCNSV YGFTGVQHGL LPCLHVAAT VTTIGREMLL ATREYVHARW AAFEQLLADF PEAADMRAPG PYSMRIIYGD TDSIFVLCRG LTAAGLTAMG DKMASHISRA LFLPPIKLE CEKTFTKLLL IAKKKYIGVI YGGKMLIKGV DLVRKNNCAF INRTSRALVD LLFYDDTVSG AAAALAERPA E EWLARPLP EGLQAFGAVL VDAHRRITDP ERDIQDFVLT AELSRHPRAY TNKRLAHLTV YYKLMARRAQ VPSIKDRIPY VI VAQTREV EETVARLAAL RELDAAAPGD EPAPPAALPS PAKRPRETPS HADPPGGASK PRKLLVSELA EDPAYAIAHG VAL NTDYYF SHLLGAACVT FKALFGNNAK ITESLLKRFI PEVWHPPDDV AARLRAAGFG AVGAGATAEE TRRMLHRAFD TLA UniProtKB: DNA polymerase |
-分子 #2: DNA polymerase processivity factor
| 分子 | 名称: DNA polymerase processivity factor / タイプ: protein_or_peptide / ID: 2 詳細: Herpes simplex virus 1 (KOS strain) DNA polymerase processivity factor UL42 residues 1-340 tagged with a prescission protease cleavage site followed by a maltose binding proten (MBP) tag in ...詳細: Herpes simplex virus 1 (KOS strain) DNA polymerase processivity factor UL42 residues 1-340 tagged with a prescission protease cleavage site followed by a maltose binding proten (MBP) tag in the expression construct コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) Human alphaherpesvirus 1 strain KOS (ヘルペスウイルス) |
| 分子量 | 理論値: 36.34618 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDSPGGVAP ASPVEDASDA SLGQPEEGAP CQVVLQGAEL NGILQAFAPL RTSLLDSLLV MGDRGILIHN TIFGEQVFLP LEHSQFSRY RWRGPTAAFL SLVDQKRSLL SVFRANQYPD LRRVELAITG QAPFRTLVQR IWTTTSDGEA VELASETLMK R ELTSFVVL ...文字列: MTDSPGGVAP ASPVEDASDA SLGQPEEGAP CQVVLQGAEL NGILQAFAPL RTSLLDSLLV MGDRGILIHN TIFGEQVFLP LEHSQFSRY RWRGPTAAFL SLVDQKRSLL SVFRANQYPD LRRVELAITG QAPFRTLVQR IWTTTSDGEA VELASETLMK R ELTSFVVL VPQGTPDVQL RLTRPQLTKV LNATGADSAT PTTFELGVNG KFSVFTTSTC VTFAAREEGV SSSTSTQVQI LS NALTKAG QAAANAKTVY GENTHRTFSV VVDDCSMRAV LRRLQVAGGT LKFFLTTPVP SLCVTATGPN AVSAVFLLKP QKI CLDWLG HSQGSPSAGS SASR UniProtKB: DNA polymerase processivity factor |
-分子 #3: PRIMER DNA (33-MER)
| 分子 | 名称: PRIMER DNA (33-MER) / タイプ: dna / ID: 3 詳細: Contains phosphorothioated 3' mismatch with template DNA コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.227623 KDa |
| 配列 | 文字列: (DG)(DA)(DT)(DT)(DA)(DC)(DG)(DA)(DA)(DT) (DT)(DC)(DG)(DA)(DG)(DC)(DT)(DC)(DG)(DG) (DT)(DA)(DC)(DC)(DC)(DG)(DG)(DG)(DG) (DA)(DT)(DC)(GS) |
-分子 #4: TEMPLATE DNA (50-MER)
| 分子 | 名称: TEMPLATE DNA (50-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.223799 KDa |
| 配列 | 文字列: (DC)(DA)(DC)(DA)(DC)(DA)(DC)(DA)(DC)(DA) (DC)(DA)(DC)(DA)(DC)(DA)(DC)(DA)(DG)(DA) (DT)(DC)(DC)(DC)(DC)(DG)(DG)(DG)(DT) (DA)(DC)(DC)(DG)(DA)(DG)(DC)(DT)(DC)(DG) (DA) (DA)(DT)(DT)(DC)(DG)(DT)(DA)(DA) (DT)(DC) |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.38 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 25 mM HEPES, pH 7.5, 150 mM NaCl, 2 mM tris(2-carboxyethyl)phosphine (TCEP) |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR 詳細: GRIDS WERE GLOW DISCHARGED IN A PELCO EASIGLOW AT 15 MA FOR 30 SECONDS UNDER 0.39 MBAR PRESSURE. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: 3 microliters of sample were blotted for 3 seconds with filter paper saturated under 100% humidity prior to plunging.. |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 77.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 5076 / 平均露光時間: 1.5 sec. / 平均電子線量: 58.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 60606 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | rigid body, minimization_global, local_grid_search, adp refinement |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 54.86 / 当てはまり具合の基準: correlation coefficient |
| 得られたモデル |  PDB-8v1s: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)