+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacillus niacini flavin monooxygenase | |||||||||
 マップデータ マップデータ | Homologous refinement map filtered by estimated local resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Monooxygenase / flavin binding / CYTOSOLIC PROTEIN | |||||||||
| 生物種 |  Neobacillus niacini (バクテリア) Neobacillus niacini (バクテリア) | |||||||||
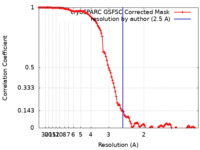
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Richardson BC / French JB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2024 ジャーナル: Biochemistry / 年: 2024タイトル: Structural and Functional Characterization of a Novel Class A Flavin Monooxygenase from . 著者: Brian C Richardson / Zachary R Turlington / Sofia Vaz Ferreira de Macedo / Sara K Phillips / Kay Perry / Savannah G Brancato / Emmalee W Cooke / Jonathan R Gwilt / Morgan A Dasovich / Andrew ...著者: Brian C Richardson / Zachary R Turlington / Sofia Vaz Ferreira de Macedo / Sara K Phillips / Kay Perry / Savannah G Brancato / Emmalee W Cooke / Jonathan R Gwilt / Morgan A Dasovich / Andrew J Roering / Francis M Rossi / Mark J Snider / Jarrod B French / Katherine A Hicks /  要旨: A gene cluster responsible for the degradation of nicotinic acid (NA) in has recently been identified, and the structures and functions of the resulting enzymes are currently being evaluated to ...A gene cluster responsible for the degradation of nicotinic acid (NA) in has recently been identified, and the structures and functions of the resulting enzymes are currently being evaluated to establish pathway intermediates. One of the genes within this cluster encodes a flavin monooxygenase (BnFMO) that is hypothesized to catalyze a hydroxylation reaction. Kinetic analyses of the recombinantly purified BnFMO suggest that this enzyme catalyzes the hydroxylation of 2,6-dihydroxynicotinic acid (2,6-DHNA) or 2,6-dihydroxypyridine (2,6-DHP), which is formed spontaneously by the decarboxylation of 2,6-DHNA. To understand the details of this hydroxylation reaction, we determined the structure of BnFMO using a multimodel approach combining protein X-ray crystallography and cryo-electron microscopy (cryo-EM). A liganded BnFMO cryo-EM structure was obtained in the presence of 2,6-DHP, allowing us to make predictions about potential catalytic residues. The structural data demonstrate that BnFMO is trimeric, which is unusual for Class A flavin monooxygenases. In both the electron density and coulomb potential maps, a region at the trimeric interface was observed that was consistent with and modeled as lipid molecules. High-resolution mass spectral analysis suggests that there is a mixture of phosphatidylethanolamine and phosphatidylglycerol lipids present. Together, these data provide insights into the molecular details of the central hydroxylation reaction unique to the aerobic degradation of NA in . | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42489.map.gz emd_42489.map.gz | 8.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42489-v30.xml emd-42489-v30.xml emd-42489.xml emd-42489.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42489_fsc.xml emd_42489_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42489.png emd_42489.png | 112.8 KB | ||
| Filedesc metadata |  emd-42489.cif.gz emd-42489.cif.gz | 6.7 KB | ||
| その他 |  emd_42489_additional_1.map.gz emd_42489_additional_1.map.gz emd_42489_half_map_1.map.gz emd_42489_half_map_1.map.gz emd_42489_half_map_2.map.gz emd_42489_half_map_2.map.gz | 82.8 MB 154.6 MB 154.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42489 http://ftp.pdbj.org/pub/emdb/structures/EMD-42489 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42489 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42489 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42489_validation.pdf.gz emd_42489_validation.pdf.gz | 900.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42489_full_validation.pdf.gz emd_42489_full_validation.pdf.gz | 900.5 KB | 表示 | |
| XML形式データ |  emd_42489_validation.xml.gz emd_42489_validation.xml.gz | 21.1 KB | 表示 | |
| CIF形式データ |  emd_42489_validation.cif.gz emd_42489_validation.cif.gz | 26.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42489 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42489 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42489 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42489 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42489.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42489.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Homologous refinement map filtered by estimated local resolution | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65119 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Flavin monooxygenase trimer
| 全体 | 名称: Flavin monooxygenase trimer |
|---|---|
| 要素 |
|
-超分子 #1: Flavin monooxygenase trimer
| 超分子 | 名称: Flavin monooxygenase trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Neobacillus niacini (バクテリア) Neobacillus niacini (バクテリア) |
| 分子量 | 理論値: 165 KDa |
-分子 #1: Flavin monooxygenase
| 分子 | 名称: Flavin monooxygenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neobacillus niacini (バクテリア) Neobacillus niacini (バクテリア) |
| 分子量 | 理論値: 52.534094 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (MSE)GSSHHHHHH SSGENLYFQG H(MSE)EELIKEVQ SDVCIVGAGP AG(MSE)LLGLLLA KQGLEVIVLE QNGDFHRE Y RGEITQPRFV QL(MSE)KQLNLLD YIESNSHVKI PEVNVFHNNV KI(MSE)QLAFNTL IDEESYCARL TQPTLLSALL D KAKKYPNF ...文字列: (MSE)GSSHHHHHH SSGENLYFQG H(MSE)EELIKEVQ SDVCIVGAGP AG(MSE)LLGLLLA KQGLEVIVLE QNGDFHRE Y RGEITQPRFV QL(MSE)KQLNLLD YIESNSHVKI PEVNVFHNNV KI(MSE)QLAFNTL IDEESYCARL TQPTLLSALL D KAKKYPNF KLLFNTKVRD LLREDGKVTG VYAVAKPGEQ INFTEDEVFE GNLNIKSRVT VGVDGRNST(MSE) EKLGNFEL E LDYYDNDLLW FSFEKPESWD YNIYHFYFQK NYNYLFLPKL GGYIQCGISL TKGEYQKIKK EGIESFKEKI LED(MSE)P ILKQ HFDTVTDFKS FVQLLCR(MSE)RY IKDWAKEEGC (MSE)LIGDAAHCV TPWGAVGSTL A(MSE)GTAVIAAD VIYK GFKNN DLSLETLKQV QSRRKEEVK(MSE) IQNLQLTIEK FLTREPIKKE IAPL(MSE)FSIAT K(MSE)PDITNLYK KLF TREFPL DIDESFIFHD ELVEAN |
-分子 #2: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 3 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #3: 1-CIS-9-OCTADECANOYL-2-CIS-9-HEXADECANOYL PHOSPHATIDYL GLYCEROL
| 分子 | 名称: 1-CIS-9-OCTADECANOYL-2-CIS-9-HEXADECANOYL PHOSPHATIDYL GLYCEROL タイプ: ligand / ID: 3 / コピー数: 3 / 式: DR9 |
|---|---|
| 分子量 | 理論値: 746.991 Da |
| Chemical component information |  ChemComp-DR9: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 4176 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 165000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8urc: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)