+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The mTORC1 cholesterol sensor LYCHOS (GPR155) - dimer with auxin | ||||||||||||
 マップデータ マップデータ | Final combined map, filtered to Gold-standard resolution. Not sharpened. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | PIN-FORMED / GPCR / Cholesterol / auxin / transporter / cell-growth / mTORC1 / cancer / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to cholesterol / cholesterol binding / negative regulation of BMP signaling pathway / positive regulation of TORC1 signaling / cellular response to amino acid starvation / transmembrane transport / cognition / intracellular signal transduction / lysosomal membrane / extracellular exosome 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.4 Å | ||||||||||||
 データ登録者 データ登録者 | Bayly-Jones C / Lupton CJ / Ellisdon AM | ||||||||||||
| 資金援助 |  オーストラリア, オーストラリア,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 | ジャーナル: Science / 年: 2022 タイトル: Lysosomal GPCR-like protein LYCHOS signals cholesterol sufficiency to mTORC1. 著者: Hijai R Shin / Y Rose Citron / Lei Wang / Laura Tribouillard / Claire S Goul / Robin Stipp / Yusuke Sugasawa / Aakriti Jain / Nolwenn Samson / Chun-Yan Lim / Oliver B Davis / David Castaneda- ...著者: Hijai R Shin / Y Rose Citron / Lei Wang / Laura Tribouillard / Claire S Goul / Robin Stipp / Yusuke Sugasawa / Aakriti Jain / Nolwenn Samson / Chun-Yan Lim / Oliver B Davis / David Castaneda-Carpio / Mingxing Qian / Daniel K Nomura / Rushika M Perera / Eunyong Park / Douglas F Covey / Mathieu Laplante / Alex S Evers / Roberto Zoncu /    要旨: Lysosomes coordinate cellular metabolism and growth upon sensing of essential nutrients, including cholesterol. Through bioinformatic analysis of lysosomal proteomes, we identified lysosomal ...Lysosomes coordinate cellular metabolism and growth upon sensing of essential nutrients, including cholesterol. Through bioinformatic analysis of lysosomal proteomes, we identified lysosomal cholesterol signaling (LYCHOS, previously annotated as G protein-coupled receptor 155), a multidomain transmembrane protein that enables cholesterol-dependent activation of the master growth regulator, the protein kinase mechanistic target of rapamycin complex 1 (mTORC1). Cholesterol bound to the amino-terminal permease-like region of LYCHOS, and mutating this site impaired mTORC1 activation. At high cholesterol concentrations, LYCHOS bound to the GATOR1 complex, a guanosine triphosphatase (GTPase)-activating protein for the Rag GTPases, through a conserved cytoplasm-facing loop. By sequestering GATOR1, LYCHOS promotes cholesterol- and Rag-dependent recruitment of mTORC1 to lysosomes. Thus, LYCHOS functions in a lysosomal pathway for cholesterol sensing and couples cholesterol concentrations to mTORC1-dependent anabolic signaling. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41930.map.gz emd_41930.map.gz | 49.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41930-v30.xml emd-41930-v30.xml emd-41930.xml emd-41930.xml | 26.4 KB 26.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41930_fsc.xml emd_41930_fsc.xml | 11 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41930.png emd_41930.png | 99.4 KB | ||
| マスクデータ |  emd_41930_msk_1.map emd_41930_msk_1.map | 98.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41930.cif.gz emd-41930.cif.gz | 7.3 KB | ||
| その他 |  emd_41930_additional_1.map.gz emd_41930_additional_1.map.gz emd_41930_additional_2.map.gz emd_41930_additional_2.map.gz emd_41930_half_map_1.map.gz emd_41930_half_map_1.map.gz emd_41930_half_map_2.map.gz emd_41930_half_map_2.map.gz | 112.1 MB 93 MB 91.6 MB 91.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41930 http://ftp.pdbj.org/pub/emdb/structures/EMD-41930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41930 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41930_validation.pdf.gz emd_41930_validation.pdf.gz | 753 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41930_full_validation.pdf.gz emd_41930_full_validation.pdf.gz | 752.6 KB | 表示 | |
| XML形式データ |  emd_41930_validation.xml.gz emd_41930_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_41930_validation.cif.gz emd_41930_validation.cif.gz | 23.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41930 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41930 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8u5qMC  8u54C  8u56C  8u58C  8u5cC  8u5nC  8u5vC  8u5xC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41930.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41930.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final combined map, filtered to Gold-standard resolution. Not sharpened. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1081 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41930_msk_1.map emd_41930_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Monomer of LYCHOS (GPR155)
| 全体 | 名称: Monomer of LYCHOS (GPR155) |
|---|---|
| 要素 |
|
-超分子 #1: Monomer of LYCHOS (GPR155)
| 超分子 | 名称: Monomer of LYCHOS (GPR155) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 99 KDa |
-分子 #1: Integral membrane protein GPR155
| 分子 | 名称: Integral membrane protein GPR155 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 99.276891 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MNSNLPAENL TIAVNMTKTL PTAVTHGFNS TNDPPSMSIT RLFPALLECF GIVLCGYIAG RANVITSTQA KGLGNFVSRF ALPALLFKN MVVLNFSNVD WSFLYSILIA KASVFFIVCV LTLLVASPDS RFSKAGLFPI FATQSNDFAL GYPIVEALYQ T TYPEYLQY ...文字列: MNSNLPAENL TIAVNMTKTL PTAVTHGFNS TNDPPSMSIT RLFPALLECF GIVLCGYIAG RANVITSTQA KGLGNFVSRF ALPALLFKN MVVLNFSNVD WSFLYSILIA KASVFFIVCV LTLLVASPDS RFSKAGLFPI FATQSNDFAL GYPIVEALYQ T TYPEYLQY IYLVAPISLM MLNPIGFIFC EIQKWKDTQN ASQNKIKIVG LGLLRVLQNP IVFMVFIGIA FNFILDRKVP VY VENFLDG LGNSFSGSAL FYLGLTMVGK IKRLKKSAFV VLILLITAKL LVLPLLCREM VELLDKGDSV VNHTSLSNYA FLY GVFPVA PGVAIFATQF NMEVEIITSG MVISTFVSAP IMYVSAWLLT FPTMDPKPLA YAIQNVSFDI SIVSLISLIW SLAI LLLSK KYKQLPHMLT TNLLIAQSIV CAGMMIWNFV KEKNFVGQIL VFVLLYSSLY STYLWTGLLA ISLFLLKKRE RVQIP VGII IISGWGIPAL LVGVLLITGK HNGDSIDSAF FYGKEQMITT AVTLFCSILI AGISLMCMNQ TAQAGSYEGF DQSQSH KVV EPGNTAFEES PAPVNEPELF TSSIPETSCC SCSMGNGELH CPSIEPIANT STSEPVIPSF EKNNHCVSRC NSQSCIL AQ EEEQYLQSGD QQLTRHVLLC LLLIIGLFAN LSSCLWWLFN QEPGRLYVEL QFFCAVFNFG QGFISFGIFG LDKHLIIL P FKRRLEFLWN NKDTAENRDS PVSEEIKMTC QQFIHYHRDL CIRNIVKERR CGAKTSAGTF CGCDLVSWLI EVGLASDRG EAVIYGDRLV QGGVIQHITN EYEFRDEYLF YRFLQKSPEQ SPPAINANTL QQERYKEIEH SSPPSHSPKT GSGSDYKDDD DKDYKDDDD K UniProtKB: Lysosomal cholesterol signaling protein |
-分子 #2: 1H-INDOL-3-YLACETIC ACID
| 分子 | 名称: 1H-INDOL-3-YLACETIC ACID / タイプ: ligand / ID: 2 / コピー数: 2 / 式: IAC |
|---|---|
| 分子量 | 理論値: 175.184 Da |
| Chemical component information | 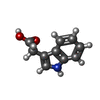 ChemComp-IAC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 8067 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: AlphaFold / Chain - Initial model type: in silico model 詳細: A monomeric assembly was predicted with AlphaFold and then rigid body fit into the map. Subsequent refinements were performed in ISOLDE. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8u5q: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






























