+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human DNMT3A UDR bound to H2AK119ub1-modified nucleosome | |||||||||
 マップデータ マップデータ | main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA cytosine methyltransferase / STRUCTURAL PROTEIN-TRANSFERASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of cellular response to hypoxia / epigenetic programming of gene expression / cellular response to bisphenol A / protein-cysteine methyltransferase activity / DNA (cytosine-5-)-methyltransferase / unmethylated CpG binding / DNA (cytosine-5-)-methyltransferase activity / autosome genomic imprinting / S-adenosylmethionine metabolic process / SUMOylation of DNA methylation proteins ...positive regulation of cellular response to hypoxia / epigenetic programming of gene expression / cellular response to bisphenol A / protein-cysteine methyltransferase activity / DNA (cytosine-5-)-methyltransferase / unmethylated CpG binding / DNA (cytosine-5-)-methyltransferase activity / autosome genomic imprinting / S-adenosylmethionine metabolic process / SUMOylation of DNA methylation proteins / DNA methylation-dependent heterochromatin formation / XY body / cellular response to ethanol / response to vitamin A / negative regulation of gene expression via chromosomal CpG island methylation / response to ionizing radiation / hepatocyte apoptotic process / chromosome, centromeric region / catalytic complex / heterochromatin / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / Glycogen synthesis / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Endosomal Sorting Complex Required For Transport (ESCRT) / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Constitutive Signaling by NOTCH1 HD Domain Mutants / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / NOTCH2 Activation and Transmission of Signal to the Nucleus / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / p75NTR recruits signalling complexes / Downregulation of ERBB4 signaling / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / Pexophagy / Regulation of innate immune responses to cytosolic DNA / InlA-mediated entry of Listeria monocytogenes into host cells / VLDLR internalisation and degradation / Downregulation of ERBB2:ERBB3 signaling / NF-kB is activated and signals survival / NRIF signals cell death from the nucleus / Regulation of PTEN localization / Activated NOTCH1 Transmits Signal to the Nucleus / Regulation of BACH1 activity / Translesion synthesis by REV1 / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / Translesion synthesis by POLK / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / Downregulation of TGF-beta receptor signaling / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / DNA methylation / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Translesion synthesis by POLI / Gap-filling DNA repair synthesis and ligation in GG-NER / Josephin domain DUBs / Regulation of activated PAK-2p34 by proteasome mediated degradation / InlB-mediated entry of Listeria monocytogenes into host cell / IKK complex recruitment mediated by RIP1 / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / PRC2 methylates histones and DNA / response to cocaine / Autodegradation of Cdh1 by Cdh1:APC/C / TNFR1-induced NF-kappa-B signaling pathway / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / Defective pyroptosis / TCF dependent signaling in response to WNT / SCF-beta-TrCP mediated degradation of Emi1 / AUF1 (hnRNP D0) binds and destabilizes mRNA / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D / Regulation of NF-kappa B signaling / TNFR2 non-canonical NF-kB pathway / activated TAK1 mediates p38 MAPK activation / Assembly of the pre-replicative complex / Vpu mediated degradation of CD4 / NOTCH3 Activation and Transmission of Signal to the Nucleus / Negative regulators of DDX58/IFIH1 signaling / Deactivation of the beta-catenin transactivating complex / Degradation of DVL / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Regulation of signaling by CBL / Dectin-1 mediated noncanonical NF-kB signaling 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) / | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.23 Å | |||||||||
 データ登録者 データ登録者 | Chen X / Song J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of human DNMT3A N-terminal domain bound to H2AK119Ub nucleosome 著者: Chen X / Song J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41922.map.gz emd_41922.map.gz | 25.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41922-v30.xml emd-41922-v30.xml emd-41922.xml emd-41922.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41922.png emd_41922.png | 102.3 KB | ||
| Filedesc metadata |  emd-41922.cif.gz emd-41922.cif.gz | 6.2 KB | ||
| その他 |  emd_41922_additional_1.map.gz emd_41922_additional_1.map.gz | 12.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41922 http://ftp.pdbj.org/pub/emdb/structures/EMD-41922 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41922 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41922 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41922_validation.pdf.gz emd_41922_validation.pdf.gz | 511.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41922_full_validation.pdf.gz emd_41922_full_validation.pdf.gz | 511.1 KB | 表示 | |
| XML形式データ |  emd_41922_validation.xml.gz emd_41922_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_41922_validation.cif.gz emd_41922_validation.cif.gz | 7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41922 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41922 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41922 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41922 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8u5hMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41922.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41922.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2347 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: additional map
| ファイル | emd_41922_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | additional map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
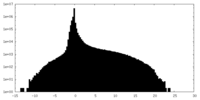
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DNMT3A N-terminal domain and H2AK119Ub nucleosome complex
| 全体 | 名称: DNMT3A N-terminal domain and H2AK119Ub nucleosome complex |
|---|---|
| 要素 |
|
-超分子 #1: DNMT3A N-terminal domain and H2AK119Ub nucleosome complex
| 超分子 | 名称: DNMT3A N-terminal domain and H2AK119Ub nucleosome complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#8 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DNA (cytosine-5)-methyltransferase 3A
| 分子 | 名称: DNA (cytosine-5)-methyltransferase 3A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.070631 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PEASRAVENG CCTPKEGRGA PAEAGKEQKE TNIESMKMEG SRGRLRGGLG WESSLRQRPM PRLTFQAGDP YYISKRKRDE WLARWKREA EKKAKVIAG UniProtKB: DNA (cytosine-5)-methyltransferase 3A |
-分子 #3: Ubiquitin
| 分子 | 名称: Ubiquitin / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.576831 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQIFVKTLTG KTITLEVEPS DTIENVKAKI QDKEGIPPDQ QRLIFAGKQL EDGRTLSDYN IQKESTLHLV LRLRGG UniProtKB: Polyubiquitin-C |
-分子 #5: Histone H3.3C
| 分子 | 名称: Histone H3.3C / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.521349 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LVTKAAKKCA PATGGVKKPH RYRPGTVALR EIRRYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQRSA VMALQEASEA YLVALFEDTN LCAIHAKRVT IMPKDIQLAR RIRGERA UniProtKB: Histone H3.3C |
-分子 #6: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.394426 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRDA VTYTEHAKRK TVTAMDVVY ALKRQGRTLY GFGG UniProtKB: Histone H4 |
-分子 #7: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 14.083398 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKQGGK TRAKAKTRSS RAGLQFPVGR VHRLLRKGNY AERVGAGAPV YLAAVLEYLT AEILELAGNA ARDNKKTRII PRHLQLAVR NDEELNKLLG RVTIAQGGVL PNIQSVLLPK CTESSKSAKS K UniProtKB: Histone H2A |
-分子 #8: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 8 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.655948 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKSAPAPKK GSKKAVTKTQ KKDGKKRRKT RKESYAIYVY KVLKQVHPDT GISSKAMSIM NSFVNDVFER IAGEASRLAH YNKRSTITS REIQTAVRLL LPGELAKHAV SEGTKAVTKY TSAK UniProtKB: Histone H2B 1.1 |
-分子 #2: nucleosome DNA
| 分子 | 名称: nucleosome DNA / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 55.176137 KDa |
| 配列 | 文字列: (DG)(DC)(DT)(DC)(DT)(DC)(DT)(DA)(DC)(DG) (DT)(DA)(DA)(DA)(DC)(DA)(DT)(DC)(DC)(DT) (DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG) (DC)(DC)(DG)(DC)(DT) ...文字列: (DG)(DC)(DT)(DC)(DT)(DC)(DT)(DA)(DC)(DG) (DT)(DA)(DA)(DA)(DC)(DA)(DT)(DC)(DC)(DT) (DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG) (DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA)(DC) (DA)(DG)(DC)(DT)(DC)(DT)(DA) (DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT)(DA) (DA)(DA)(DC) (DG)(DC)(DA)(DC)(DG)(DT) (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT)(DC) (DC)(DC)(DC)(DC) (DG)(DC)(DG)(DT)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DA) (DA)(DG)(DG)(DG)(DG) (DA)(DT)(DT)(DA) (DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT)(DC) (DT)(DC)(DC)(DA)(DG)(DG) (DC)(DA)(DC) (DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT)(DA) (DT)(DA)(DT)(DA)(DC)(DA)(DT) (DC)(DC) (DT)(DG)(DT)(DG)(DA)(DC)(DT)(DT)(DA)(DC) (DC)(DC)(DA)(DC)(DT)(DT)(DC)(DG) |
-分子 #4: nucleosome DNA
| 分子 | 名称: nucleosome DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 55.963656 KDa |
| 配列 | 文字列: (DC)(DG)(DA)(DA)(DG)(DT)(DG)(DG)(DG)(DT) (DA)(DA)(DG)(DT)(DC)(DA)(DC)(DA)(DG)(DG) (DA)(DT)(DG)(DT)(DA)(DT)(DA)(DT)(DA) (DT)(DC)(DT)(DG)(DA)(DC)(DA)(DC)(DG)(DT) (DG) (DC)(DC)(DT)(DG)(DG) ...文字列: (DC)(DG)(DA)(DA)(DG)(DT)(DG)(DG)(DG)(DT) (DA)(DA)(DG)(DT)(DC)(DA)(DC)(DA)(DG)(DG) (DA)(DT)(DG)(DT)(DA)(DT)(DA)(DT)(DA) (DT)(DC)(DT)(DG)(DA)(DC)(DA)(DC)(DG)(DT) (DG) (DC)(DC)(DT)(DG)(DG)(DA)(DG)(DA) (DC)(DT)(DA)(DG)(DG)(DG)(DA)(DG)(DT)(DA) (DA)(DT) (DC)(DC)(DC)(DC)(DT)(DT)(DG) (DG)(DC)(DG)(DG)(DT)(DT)(DA)(DA)(DA)(DA) (DC)(DG)(DC) (DG)(DG)(DG)(DG)(DG)(DA) (DC)(DA)(DG)(DC)(DG)(DC)(DG)(DT)(DA)(DC) (DG)(DT)(DG)(DC) (DG)(DT)(DT)(DT)(DA) (DA)(DG)(DC)(DG)(DG)(DT)(DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT) (DA)(DC)(DG)(DA)(DC)(DC)(DA)(DA)(DT)(DT) (DG)(DA)(DG)(DC)(DG)(DG) (DC)(DC)(DT) (DC)(DG)(DG)(DC)(DA)(DC)(DC)(DG)(DG)(DG) (DA)(DT)(DT)(DC)(DT)(DC)(DC) (DA)(DG) (DG)(DA)(DT)(DG)(DT)(DT)(DT)(DA)(DC)(DG) (DT)(DA)(DG)(DA)(DG)(DA)(DG)(DC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.23 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 86685 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X