| 登録情報 | データベース: EMDB / ID: EMD-41574
|
|---|
| タイトル | Cryo-EM structure of transglutaminase 2 bound to GDP |
|---|
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: Transglutaminase 2 bound to GDP
- タンパク質・ペプチド: Protein-glutamine gamma-glutamyltransferase 2
- リガンド: GUANOSINE-5'-DIPHOSPHATE
|
|---|
 キーワード キーワード | GDP / Complex / Signaling / G-protein / Cancer / cryo-EM / TRANSFERASE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
histone serotonyltransferase activity / histone dopaminyltransferase activity / peptide noradrenalinyltransferase activity / peptide histaminyltransferase activity / cellular response to serotonin / regulation of apoptotic cell clearance / protein deamination / protein-glutamine glutaminase activity / protein-glutamine glutaminase / protein-glutamine gamma-glutamyltransferase ...histone serotonyltransferase activity / histone dopaminyltransferase activity / peptide noradrenalinyltransferase activity / peptide histaminyltransferase activity / cellular response to serotonin / regulation of apoptotic cell clearance / protein deamination / protein-glutamine glutaminase activity / protein-glutamine glutaminase / protein-glutamine gamma-glutamyltransferase / positive regulation of mitochondrial calcium ion concentration / salivary gland cavitation / protein-glutamine gamma-glutamyltransferase activity / negative regulation of endoplasmic reticulum calcium ion concentration / dopamine secretion / peptide cross-linking / branching involved in salivary gland morphogenesis / cellular response to dopamine / positive regulation of small GTPase mediated signal transduction / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / apoptotic cell clearance / cellular response to cocaine / positive regulation of neurogenesis / positive regulation of sprouting angiogenesis / positive regulation of cell adhesion / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / extracellular matrix / positive regulation of GTPase activity / bone development / protein homooligomerization / nucleosome / peptidase activity / phospholipase C-activating G protein-coupled receptor signaling pathway / gene expression / collagen-containing extracellular matrix / regulation of apoptotic process / positive regulation of apoptotic process / focal adhesion / calcium ion binding / chromatin / GTP binding / perinuclear region of cytoplasm / endoplasmic reticulum / mitochondrion / proteolysis / extracellular exosome / nucleus / plasma membrane / cytosol類似検索 - 分子機能 : / Transglutaminase, N-terminal / Transglutaminase, C-terminal / Transglutaminase, active site / Protein-glutamine gamma-glutamyltransferase, animal / Transglutaminase, C-terminal domain superfamily / Transglutaminase family / Transglutaminase family, C-terminal ig like domain / Transglutaminases active site. / Transglutaminase-like superfamily ...: / Transglutaminase, N-terminal / Transglutaminase, C-terminal / Transglutaminase, active site / Protein-glutamine gamma-glutamyltransferase, animal / Transglutaminase, C-terminal domain superfamily / Transglutaminase family / Transglutaminase family, C-terminal ig like domain / Transglutaminases active site. / Transglutaminase-like superfamily / Transglutaminase/protease-like homologues / Transglutaminase-like / Transglutaminase-like superfamily / Papain-like cysteine peptidase superfamily / Immunoglobulin E-set / Immunoglobulin-like fold類似検索 - ドメイン・相同性 Protein-glutamine gamma-glutamyltransferase 2類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
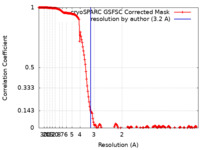
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å |
|---|
 データ登録者 データ登録者 | Aplin C / Cerione RA |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | Organization | Grant number | 国 |
|---|
| National Institutes of Health/National Cancer Institute (NIH/NCI) | R01CA201402-09 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Conformational activation and inhibition of transglutaminase 2 determined by static and time resolved small-angle X-ray scattering and cryoelectron microscopy
著者: Aplin C / Zielinski KA / Pabit S / Ogunribido D / Katt WP / Pollack L / Cerione RA / Milano SK |
|---|
| 履歴 | | 登録 | 2023年8月9日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2024年7月31日 | - |
|---|
| マップ公開 | 2024年7月31日 | - |
|---|
| 更新 | 2024年7月31日 | - |
|---|
| 現状 | 2024年7月31日 | 処理サイト: RCSB / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_41574.map.gz
emd_41574.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-41574-v30.xml
emd-41574-v30.xml emd-41574.xml
emd-41574.xml EMDBヘッダ
EMDBヘッダ emd_41574_fsc.xml
emd_41574_fsc.xml FSCデータファイル
FSCデータファイル emd_41574.png
emd_41574.png emd-41574.cif.gz
emd-41574.cif.gz emd_41574_half_map_1.map.gz
emd_41574_half_map_1.map.gz emd_41574_half_map_2.map.gz
emd_41574_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-41574
http://ftp.pdbj.org/pub/emdb/structures/EMD-41574 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41574
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41574 emd_41574_validation.pdf.gz
emd_41574_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_41574_full_validation.pdf.gz
emd_41574_full_validation.pdf.gz emd_41574_validation.xml.gz
emd_41574_validation.xml.gz emd_41574_validation.cif.gz
emd_41574_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41574
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41574 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41574
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41574
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_41574.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_41574.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X