+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of GPR61- | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GPCR / inverse agonist / membrane protein / BRIL / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ligand-independent adenylate cyclase-activating G protein-coupled receptor signaling pathway / arrestin family protein binding / G protein-coupled receptor activity / electron transport chain / periplasmic space / electron transfer activity / receptor complex / endosome membrane / endosome / iron ion binding ...ligand-independent adenylate cyclase-activating G protein-coupled receptor signaling pathway / arrestin family protein binding / G protein-coupled receptor activity / electron transport chain / periplasmic space / electron transfer activity / receptor complex / endosome membrane / endosome / iron ion binding / G protein-coupled receptor signaling pathway / heme binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
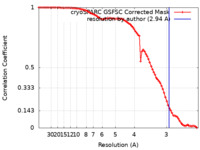
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.94 Å | |||||||||
 データ登録者 データ登録者 | Lees JA / Dias JM / Han S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: An inverse agonist of orphan receptor GPR61 acts by a G protein-competitive allosteric mechanism. 著者: Joshua A Lees / João M Dias / Francis Rajamohan / Jean-Philippe Fortin / Rebecca O'Connor / Jimmy X Kong / Emily A G Hughes / Ethan L Fisher / Jamison B Tuttle / Gabrielle Lovett / Bethany L ...著者: Joshua A Lees / João M Dias / Francis Rajamohan / Jean-Philippe Fortin / Rebecca O'Connor / Jimmy X Kong / Emily A G Hughes / Ethan L Fisher / Jamison B Tuttle / Gabrielle Lovett / Bethany L Kormos / Rayomand J Unwalla / Lei Zhang / Anne-Marie Dechert Schmitt / Dahui Zhou / Michael Moran / Kimberly A Stevens / Kimberly F Fennell / Alison E Varghese / Andrew Maxwell / Emmaline E Cote / Yuan Zhang / Seungil Han /  要旨: GPR61 is an orphan GPCR related to biogenic amine receptors. Its association with phenotypes relating to appetite makes it of interest as a druggable target to treat disorders of metabolism and body ...GPR61 is an orphan GPCR related to biogenic amine receptors. Its association with phenotypes relating to appetite makes it of interest as a druggable target to treat disorders of metabolism and body weight, such as obesity and cachexia. To date, the lack of structural information or a known biological ligand or tool compound has hindered comprehensive efforts to study GPR61 structure and function. Here, we report a structural characterization of GPR61, in both its active-like complex with heterotrimeric G protein and in its inactive state. Moreover, we report the discovery of a potent and selective small-molecule inverse agonist against GPR61 and structural elucidation of its allosteric binding site and mode of action. These findings offer mechanistic insights into an orphan GPCR while providing both a structural framework and tool compound to support further studies of GPR61 function and modulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41145.map.gz emd_41145.map.gz | 56.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41145-v30.xml emd-41145-v30.xml emd-41145.xml emd-41145.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41145_fsc.xml emd_41145_fsc.xml | 8.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41145.png emd_41145.png | 96.2 KB | ||
| Filedesc metadata |  emd-41145.cif.gz emd-41145.cif.gz | 6.6 KB | ||
| その他 |  emd_41145_half_map_1.map.gz emd_41145_half_map_1.map.gz emd_41145_half_map_2.map.gz emd_41145_half_map_2.map.gz | 55.4 MB 55.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41145 http://ftp.pdbj.org/pub/emdb/structures/EMD-41145 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41145 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41145 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41145_validation.pdf.gz emd_41145_validation.pdf.gz | 812.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41145_full_validation.pdf.gz emd_41145_full_validation.pdf.gz | 812 KB | 表示 | |
| XML形式データ |  emd_41145_validation.xml.gz emd_41145_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_41145_validation.cif.gz emd_41145_validation.cif.gz | 20.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41145 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41145 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41145 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41145 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tb7MC  8tb0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41145.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41145.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.18 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_41145_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
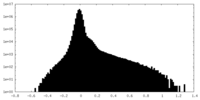
| 投影像・断面図 |
| ||||||||||||
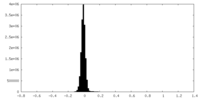
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41145_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
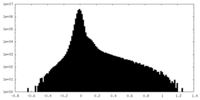
| 投影像・断面図 |
| ||||||||||||
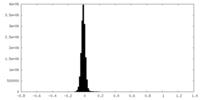
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GPR61-ICL3-BRIL chimera in complex with anti-BRIL Fab24 BAK5 and ...
| 全体 | 名称: GPR61-ICL3-BRIL chimera in complex with anti-BRIL Fab24 BAK5 and hinge-binding nanobody bound to inverse agonist Compound 1 |
|---|---|
| 要素 |
|
-超分子 #1: GPR61-ICL3-BRIL chimera in complex with anti-BRIL Fab24 BAK5 and ...
| 超分子 | 名称: GPR61-ICL3-BRIL chimera in complex with anti-BRIL Fab24 BAK5 and hinge-binding nanobody bound to inverse agonist Compound 1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: G-protein coupled receptor 61,Soluble cytochrome b562
| 分子 | 名称: G-protein coupled receptor 61,Soluble cytochrome b562 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 59.991902 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYPYDVPDYA ESSPIPQSSG NSSTLGRVPQ TPGPSTASGV PEVGLRDVAS ESVALFFMLL LDLTAVAGNA AVMAVIAKTP ALRKFVFVF HLCLVDLLAA LTLMPLAMLS SSALFDHALF GEVACRLYLF LSVCFVSLAI LSVSAINVER YYYVVHPMRY E VRMTLGLV ...文字列: MYPYDVPDYA ESSPIPQSSG NSSTLGRVPQ TPGPSTASGV PEVGLRDVAS ESVALFFMLL LDLTAVAGNA AVMAVIAKTP ALRKFVFVF HLCLVDLLAA LTLMPLAMLS SSALFDHALF GEVACRLYLF LSVCFVSLAI LSVSAINVER YYYVVHPMRY E VRMTLGLV ASVLVGVWVK ALAMASVPVL GRVSWEEGAP SVPPGCSLQW SHSAYCQLFV VVFAVLYFLL PLLLILVVYC SM FRARRQL ADLEDNWETL NDNLKVIEKA DNAAQVKDAL TKMRAAALDA QKATPPKLED KSPDSPEMKD FRHGFDILVG QID DALKLA NEGKVKEAQA AAEQLKTTRN AYIQKYLERA RSTLQKEVKA AVVLLAVGGQ FLLCWLPYFS FHLYVALSAQ PIST GQVES VVTWIGYFCF TSNPFFYGCL NRQIRGELSK QFVCFFKPAP EEELRLPSRE GSIEENFLQF LQGTGCPSES WVSRP LPSP KQEPPAVDFR IPGQIAEETS EFLEQQLTSD IIMSDSYLRP AASPRLESGS DYKDDDDAK UniProtKB: G-protein coupled receptor 61, Soluble cytochrome b562, G-protein coupled receptor 61 |
-分子 #2: Fab24 BAK5 heavy chain
| 分子 | 名称: Fab24 BAK5 heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 52.888203 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKKNIAFLLA SMFVFSIATN AYASDIQMTQ SPSSLSASVG DRVTITCRAS QSVSSAVAWY QQKPGKAPKL LIYSASSLYS GVPSRFSGS RSGTDFTLTI SSLQPEDFAT YYCQQYLYYS LVTFGQGTKV EIKRTVAAPS VFIFPPSDSQ LKSGTASVVC L LNNFYPRE ...文字列: MKKNIAFLLA SMFVFSIATN AYASDIQMTQ SPSSLSASVG DRVTITCRAS QSVSSAVAWY QQKPGKAPKL LIYSASSLYS GVPSRFSGS RSGTDFTLTI SSLQPEDFAT YYCQQYLYYS LVTFGQGTKV EIKRTVAAPS VFIFPPSDSQ LKSGTASVVC L LNNFYPRE AKVQWKVDNA LQSGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE MK KNIAFLL ASMFVFSIAT NAYAEISEVQ LVESGGGLVQ PGGSLRLSCA ASGFNVRSFS IHWVRQAPGK GLEWVAYISS SSG STSYAD SVKGRFTISA DTSKNTAYLQ MNSLRAEDTA VYYCARWGYW PGEPWWKAFD YWGQGTLVTV SSASTKGPSV FPLA PSSKS TSGGTAALGC LVKDYFPEPV TVSWNSGALT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNT KVDK KVEPKS |
-分子 #3: Fab hinge-binding nanobody
| 分子 | 名称: Fab hinge-binding nanobody / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.276541 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSQVQLQESG GGLVQPGGSL RLSCAASGRT ISRYAMSWFR QAPGKEREFV AVARRSGDGA FYADSVQGRF TVSRDDAKTV YLQMNSLKP EDTAVYYCAI DSDTFYSGSY DYWGQGTQVT VSS |
-分子 #4: Fab24 BAK5 light chain
| 分子 | 名称: Fab24 BAK5 light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.398066 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYLYYSLVT FGQGTKVEIK RTVAAPSVFI FPPSDSQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ ...文字列: MDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYLYYSLVT FGQGTKVEIK RTVAAPSVFI FPPSDSQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ DSKDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRG |
-分子 #5: 6-{[(3,5-difluoropyridin-4-yl)methyl]amino}-N-(4-ethoxy-6-methylp...
| 分子 | 名称: 6-{[(3,5-difluoropyridin-4-yl)methyl]amino}-N-(4-ethoxy-6-methylpyrimidin-2-yl)-2-methoxy-N-(2-methoxyethyl)pyridine-3-sulfonamide タイプ: ligand / ID: 5 / コピー数: 1 / 式: ZOB |
|---|---|
| 分子量 | 理論値: 524.541 Da |
| Chemical component information |  ChemComp-ZOB: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X