+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of rat cardiac sodium channel NaV1.5 with batrachotoxin analog BTX-B | |||||||||||||||
 マップデータ マップデータ | Sharpened map CryoSPARC | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Ion channel / Sodium channel / Voltage-gated channel / Sodium transport / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報voltage-gated sodium channel activity involved in AV node cell action potential / voltage-gated sodium channel activity involved in bundle of His cell action potential / voltage-gated sodium channel activity involved in SA node cell action potential / bundle of His cell action potential / AV node cell action potential / sodium channel complex / SA node cell action potential / AV node cell to bundle of His cell communication / membrane depolarization during SA node cell action potential / response to denervation involved in regulation of muscle adaptation ...voltage-gated sodium channel activity involved in AV node cell action potential / voltage-gated sodium channel activity involved in bundle of His cell action potential / voltage-gated sodium channel activity involved in SA node cell action potential / bundle of His cell action potential / AV node cell action potential / sodium channel complex / SA node cell action potential / AV node cell to bundle of His cell communication / membrane depolarization during SA node cell action potential / response to denervation involved in regulation of muscle adaptation / cardiac ventricle development / regulation of ventricular cardiac muscle cell membrane depolarization / regulation of atrial cardiac muscle cell membrane repolarization / membrane depolarization during atrial cardiac muscle cell action potential / voltage-gated sodium channel activity involved in Purkinje myocyte action potential / regulation of sodium ion transmembrane transport / brainstem development / membrane depolarization during AV node cell action potential / voltage-gated sodium channel activity involved in cardiac muscle cell action potential / membrane depolarization during bundle of His cell action potential / positive regulation of action potential / atrial cardiac muscle cell action potential / membrane depolarization during Purkinje myocyte cell action potential / telencephalon development / regulation of atrial cardiac muscle cell membrane depolarization / membrane depolarization during cardiac muscle cell action potential / positive regulation of sodium ion transport / sodium ion import across plasma membrane / ventricular cardiac muscle cell action potential / cardiac muscle cell action potential involved in contraction / regulation of ventricular cardiac muscle cell membrane repolarization / regulation of cardiac muscle cell contraction / voltage-gated sodium channel complex / membrane depolarization during action potential / voltage-gated sodium channel activity / ankyrin binding / sodium ion transport / positive regulation of heart rate / nitric-oxide synthase binding / fibroblast growth factor binding / regulation of heart rate by cardiac conduction / odontogenesis of dentin-containing tooth / membrane depolarization / intercalated disc / sodium ion transmembrane transport / lateral plasma membrane / neuronal action potential / cardiac muscle contraction / T-tubule / cellular response to calcium ion / regulation of heart rate / bioluminescence / cerebellum development / generation of precursor metabolites and energy / caveola / positive regulation of epithelial cell proliferation / response to organic cyclic compound / sarcolemma / Z disc / scaffold protein binding / transmembrane transporter binding / calmodulin binding / protein domain specific binding / axon / ubiquitin protein ligase binding / protein kinase binding / perinuclear region of cytoplasm / enzyme binding / cell surface / endoplasmic reticulum / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   | |||||||||||||||
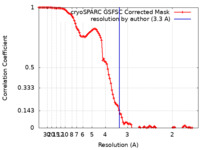
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||||||||
 データ登録者 データ登録者 | Tonggu L / Wisedchaisri G / Gamal El-Din TM / Zheng N / Catterall WA | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Dual receptor-sites reveal the structural basis for hyperactivation of sodium channels by poison-dart toxin batrachotoxin. 著者: Lige Tonggu / Goragot Wisedchaisri / Tamer M Gamal El-Din / Michael J Lenaeus / Matthew M Logan / Tatsuya Toma / Justin Du Bois / Ning Zheng / William A Catterall /   要旨: The poison dart toxin batrachotoxin is exceptional for its high potency and toxicity, and for its multifaceted modification of the function of voltage-gated sodium channels. By using cryogenic ...The poison dart toxin batrachotoxin is exceptional for its high potency and toxicity, and for its multifaceted modification of the function of voltage-gated sodium channels. By using cryogenic electron microscopy, we identify two homologous, but nonidentical receptor sites that simultaneously bind two molecules of toxin, one at the interface between Domains I and IV, and the other at the interface between Domains III and IV of the cardiac sodium channel. Together, these two bound toxin molecules stabilize α/π helical conformation in the S6 segments that gate the pore, and one of the bound BTX-B molecules interacts with the crucial Lys1421 residue that is essential for sodium conductance and selectivity via an apparent water-bridged hydrogen bond. Overall, our structure provides insight into batrachotoxin's potency, efficacy, and multifaceted functional effects on voltage-gated sodium channels via a dual receptor site mechanism. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41071.map.gz emd_41071.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41071-v30.xml emd-41071-v30.xml emd-41071.xml emd-41071.xml | 23.8 KB 23.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41071_fsc.xml emd_41071_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41071.png emd_41071.png | 143.9 KB | ||
| Filedesc metadata |  emd-41071.cif.gz emd-41071.cif.gz | 8.5 KB | ||
| その他 |  emd_41071_half_map_1.map.gz emd_41071_half_map_1.map.gz emd_41071_half_map_2.map.gz emd_41071_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41071 http://ftp.pdbj.org/pub/emdb/structures/EMD-41071 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41071 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41071 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41071_validation.pdf.gz emd_41071_validation.pdf.gz | 996.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41071_full_validation.pdf.gz emd_41071_full_validation.pdf.gz | 996.4 KB | 表示 | |
| XML形式データ |  emd_41071_validation.xml.gz emd_41071_validation.xml.gz | 19.2 KB | 表示 | |
| CIF形式データ |  emd_41071_validation.cif.gz emd_41071_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41071 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41071 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41071 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41071 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8t6lMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41071.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41071.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map CryoSPARC | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8255 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A
| ファイル | emd_41071_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
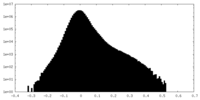
| 投影像・断面図 |
| ||||||||||||
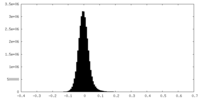
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_41071_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
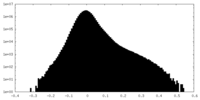
| 投影像・断面図 |
| ||||||||||||
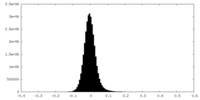
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sodium channel NaV1.5 and batrachotoxinin-A 20-alpha-benzoate
| 全体 | 名称: Sodium channel NaV1.5 and batrachotoxinin-A 20-alpha-benzoate |
|---|---|
| 要素 |
|
-超分子 #1: Sodium channel NaV1.5 and batrachotoxinin-A 20-alpha-benzoate
| 超分子 | 名称: Sodium channel NaV1.5 and batrachotoxinin-A 20-alpha-benzoate タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Sodium channel protein type 5 subunit alpha,Green fluorescent protein
| 分子 | 名称: Sodium channel protein type 5 subunit alpha,Green fluorescent protein タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 213.523875 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MANLLLPRGT SSFRRFTRES LAAIEKRMAE KQTRGGSATS QESREGLQEE EAPRPQLDLQ ASKKLPDLYG NPPRELIGEP LEDLDPFYS TQKTFIVLNK GKTIFRFSAT NALYVLSPFH PVRRAAVKIL VHSLFSMLIM CTILTNCVFM AQHDPPPWTK Y VEYTFTAI ...文字列: MANLLLPRGT SSFRRFTRES LAAIEKRMAE KQTRGGSATS QESREGLQEE EAPRPQLDLQ ASKKLPDLYG NPPRELIGEP LEDLDPFYS TQKTFIVLNK GKTIFRFSAT NALYVLSPFH PVRRAAVKIL VHSLFSMLIM CTILTNCVFM AQHDPPPWTK Y VEYTFTAI YTFESLVKIL ARGFCLHAFT FLRDPWNWLD FSVIVMAYTT EFVDLDNVSA LRTFRVLRAL KTISVISGLK TI VGALIQS VKKLADVMVL TVFCLSVFAL IGLQLFMGNL RHKCVRNFTE LNGTNGSVEA DGLVWNSLDV YLNDPANYLL KNG TTDVLL CGNSSDAGTC PEGYRCLKAG ENPDHGYTSF DSFAWAFLAL FRLMTQDCWE RLYQQTLRSA GKIYMIFFML VIFL GSFYL VNLILAVVAM AYEEQNQATI AETEEKEKRF QEAMEMLKKE HEALTIRGVD TVSRSSRQRA LSAVSVLTSA LEELE ESHR KCPPCWNRFA QHYLIWECCP LWMSIKQKVK FVVMDPFADL TITMCIVLNT LFMALEHYNM TAEFEEMLQV GNLVFT GIF TAEMTFKIIA LDPYYYFQQG WNIFDSIIVI LSLMELGLSR MGNLSVLRSF RLLRVFKLAK SWPTLNTLIK IIGNSVG AL GNLTLVLAII VFIFAVVGMQ LFGKNYSELR HRISDSGLLP RWHMMDFFHA FLIIFRILCG EWIETMWDCM EVSGQSLC L LVFLLVMVIG NLVVLNLFLA LLLSSFSADN LTAPDEDGEM NNLQLALARI QRGLRFVKRT TWDFCCGILR RRPKKPAAL ATHSQLPSCI TAPRSPPPPE VEKVPPARKE TRFEEDKRPG QGTPGDSEPV CVPIAVAESD TEDQEEDEEN SGKVWWRLRK TCYRIVEHS WFETFIIFMI LLSSGALAFE DIYLEERKTI KVLLEYADKM FTYVFVLEML LKWVAYGFKK YFTNAWCWLD F LIVDVSLV SLVANTLGFA EMGPIKSLRT LRALRPLRAL SRFEGMRVVV NALVGAIPSI MNVLLVCLIF WLIFSIMGVN LF AGKFGRC INQTEGDLPL NYTIVNNKSE CESFNVTGEL YWTKVKVNFD NVGAGYLALL QVATFKGWMD IMYAAVDSRG YEE QPQWED NLYMYIYFVV FIIFGSFFTL NLFIGVIIDN FNQQKKKLGG QDIFMTEEQK KYYNAMKKLG SKKPQKPIPR PLNK YQGFI FDIVTKQAFD VTIMFLICLN MVTMMVETDD QSPEKVNILA KINLLFVAIF TGECIVKMAA LRHYYFTNSW NIFDF VVVI LSIVGTVLSD IIQKYFFSPT LFRVIRLARI GRILRLIRGA KGIRTLLFAL MMSLPALFNI GLLLFLVMFI YSIFGM ANF AYVKWEAGID DMFNFQTFAN SMLCLFQITT SAGWDGLLSP ILNTGPPYCD PNLPNSNGSR GNCGSPAVGI LFFTTYI II SFLIVVNMYI AIILENFSVA TEESTEPLSE DDFDMFYEIW EKFDPEATQF IEYLALSDFA DALSEPLRIA KPNQISLI N MDLPMVSGDR IHCMDILFAF TKRVLGESGE MDALKIQMEE KFMAANPSKI SYEPITTTLR RKHEEVSATV IQRAFRRHL LQRSVKHASF LFRVDLEVLF QGPGSMVSKG EELFTGVVPI LVELDGDVNG HKFSVSGEGE GDATYGKLTL KFICTTGKLP VPWPTLVTT LTYGVQCFSR YPDHMKQHDF FKSAMPEGYV QERTIFFKDD GNYKTRAEVK FEGDTLVNRI ELKGIDFKED G NILGHKLE YNYNSHNVYI MADKQKNGIK VNFKIRHNIE DGSVQLADHY QQNTPIGDGP VLLPDNHYLS TQSALSKDPN EK RDHMVLL EFVTAAGITL GMDELYKGSD YKDDDDK UniProtKB: Sodium channel protein type 5 subunit alpha, Green fluorescent protein |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #6: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 6 / コピー数: 10 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #7: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 7 / コピー数: 11 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #8: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]sp...
| 分子 | 名称: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]spirost-5-en タイプ: ligand / ID: 8 / コピー数: 2 / 式: 9Z9 |
|---|---|
| 分子量 | 理論値: 544.805 Da |
| Chemical component information | 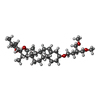 ChemComp-9Z9: |
-分子 #9: (1R)-1-[(5aR,7aR,9R,11aS,11bS,12R,13aR)-9,12-dihydroxy-2,11a-dime...
| 分子 | 名称: (1R)-1-[(5aR,7aR,9R,11aS,11bS,12R,13aR)-9,12-dihydroxy-2,11a-dimethyl-1,2,3,4,7a,8,9,10,11,11a,12,13-dodecahydro-7H-9,11b-epoxy-13a,5a-prop[1]enophenanthro[2,1-f][1,4]oxazepin-14-yl]ethyl benzoate タイプ: ligand / ID: 9 / コピー数: 2 / 式: YIJ |
|---|---|
| 分子量 | 理論値: 521.644 Da |
-分子 #10: water
| 分子 | 名称: water / タイプ: ligand / ID: 10 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 実像数: 7542 / 平均露光時間: 2.04 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X