+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Bre1-nucleosome complex - state3 | |||||||||
 マップデータ マップデータ | Structure of Bre1-nucleosome complex - state3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | H2B ubiquitin E3 ligase / dimer / nucleosome acidic patch binding protein / DNA BINDING PROTEIN-Transferase-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HULC complex / meiotic DNA double-strand break formation / telomere maintenance via recombination / mitotic intra-S DNA damage checkpoint signaling / regulation of DNA-templated DNA replication initiation / DNA replication origin binding / subtelomeric heterochromatin formation / mitotic G1 DNA damage checkpoint signaling / double-strand break repair via homologous recombination / RING-type E3 ubiquitin transferase ...HULC complex / meiotic DNA double-strand break formation / telomere maintenance via recombination / mitotic intra-S DNA damage checkpoint signaling / regulation of DNA-templated DNA replication initiation / DNA replication origin binding / subtelomeric heterochromatin formation / mitotic G1 DNA damage checkpoint signaling / double-strand break repair via homologous recombination / RING-type E3 ubiquitin transferase / structural constituent of chromatin / ubiquitin protein ligase activity / nucleosome / nucleosome assembly / scaffold protein binding / transcription by RNA polymerase II / chromosome, telomeric region / protein ubiquitination / protein heterodimerization activity / chromatin / DNA binding / nucleoplasm / identical protein binding / nucleus / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.21 Å | |||||||||
 データ登録者 データ登録者 | Zhao F / Hicks CW / Wolberger C | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Mechanism of histone H2B monoubiquitination by Bre1. 著者: Fan Zhao / Chad W Hicks / Cynthia Wolberger /  要旨: Monoubiquitination of histone H2B-K120/123 plays several roles in regulating transcription, DNA replication and the DNA damage response. The structure of a nucleosome in complex with the dimeric RING ...Monoubiquitination of histone H2B-K120/123 plays several roles in regulating transcription, DNA replication and the DNA damage response. The structure of a nucleosome in complex with the dimeric RING E3 ligase Bre1 reveals that one RING domain binds to the nucleosome acidic patch, where it can position the E2 ubiquitin conjugating enzyme Rad6, while the other RING domain contacts the DNA. Comparisons with H2A-specific E3 ligases suggest a general mechanism of tuning histone specificity via the non-E2-binding RING domain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41011.map.gz emd_41011.map.gz | 91.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41011-v30.xml emd-41011-v30.xml emd-41011.xml emd-41011.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41011_fsc.xml emd_41011_fsc.xml | 13.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41011.png emd_41011.png | 141.5 KB | ||
| Filedesc metadata |  emd-41011.cif.gz emd-41011.cif.gz | 6.4 KB | ||
| その他 |  emd_41011_half_map_1.map.gz emd_41011_half_map_1.map.gz emd_41011_half_map_2.map.gz emd_41011_half_map_2.map.gz | 165.1 MB 165.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41011 http://ftp.pdbj.org/pub/emdb/structures/EMD-41011 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41011 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41011 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41011_validation.pdf.gz emd_41011_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41011_full_validation.pdf.gz emd_41011_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_41011_validation.xml.gz emd_41011_validation.xml.gz | 20.9 KB | 表示 | |
| CIF形式データ |  emd_41011_validation.cif.gz emd_41011_validation.cif.gz | 26.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41011 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41011 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41011 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41011 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41011.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41011.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Bre1-nucleosome complex - state3 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_41011_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
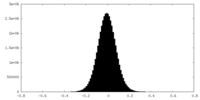
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_41011_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bre1-nucleosome complex
| 全体 | 名称: Bre1-nucleosome complex |
|---|---|
| 要素 |
|
-超分子 #1: Bre1-nucleosome complex
| 超分子 | 名称: Bre1-nucleosome complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Histone H3.2
| 分子 | 名称: Histone H3.2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.30393 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVALFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA UniProtKB: Histone H3.2 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.263231 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG UniProtKB: Histone H4 |
-分子 #3: Histone H2A type 1
| 分子 | 名称: Histone H2A type 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.978241 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKT RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAVRN DEELNKLLGR VTIAQGGVLP NIQSVLLPKK TESSKSAKSK UniProtKB: Histone H2A type 1 |
-分子 #4: Histone H2B
| 分子 | 名称: Histone H2B / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.524752 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AKSAPAPKKG SKKAVTKTQK KDGKKRRKTR KESYAIYVYK VLKQVHPDTG ISSKAMSIMN SFVNDVFERI AGEASRLAHY NKRSTITSR EIQTAVRLLL PGELAKHAVS EGTKAVTKYT SAK UniProtKB: Histone H2B 1.1 |
-分子 #7: E3 ubiquitin-protein ligase BRE1
| 分子 | 名称: E3 ubiquitin-protein ligase BRE1 / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.633649 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KLNDTEIELK HFKQKASHLE SKCEKLHDTL FRGNNKNKGS SDEALVEELA NFRTLVYCSL CSKNWKNMAI KTCGHVFCEN CCKERLAAR MRKCPTCNKA FSSNDLLTVH L UniProtKB: E3 ubiquitin-protein ligase BRE1 |
-分子 #5: 601 DNA strand 1
| 分子 | 名称: 601 DNA strand 1 / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 44.825559 KDa |
| 配列 | 文字列: (DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT) ...文字列: (DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT)(DC)(DT)(DA) (DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT)(DA) (DA)(DA) (DC)(DG)(DC)(DA)(DC)(DG)(DT) (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT)(DC) (DC)(DC)(DC) (DC)(DG)(DC)(DG)(DT)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DA) (DA)(DG)(DG)(DG) (DG)(DA)(DT)(DT)(DA) (DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT)(DC) (DT)(DC)(DC)(DA)(DG) (DG)(DC)(DA)(DC) (DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT)(DA) (DT)(DA)(DT)(DA)(DC)(DA) (DT)(DC)(DC) (DG)(DA)(DT) |
-分子 #6: 601 DNA strand 2
| 分子 | 名称: 601 DNA strand 2 / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.305852 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT) (DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC)(DT) (DC)(DG)(DA) |
-分子 #8: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 8 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20 mM HEPES pH 7.5, 50 mM NaCl, 1 mM DTT |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Crosslinked with glutaraldehyde |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.02 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.25 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| ソフトウェア | 名称: UCSF ChimeraX (ver. 1.3) |
|---|---|
| 得られたモデル |  PDB-8t3t: |
-原子モデル構築 2
| ソフトウェア | 名称: UCSF ChimeraX (ver. 1.3) |
|---|---|
| 得られたモデル |  PDB-8t3t: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)