+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli dodecamer SIR2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SIR2 / NADase / Nuclease / Anti-phage system / IMMUNE SYSTEM | |||||||||
| 機能・相同性 | SIR2-like domain / SIR2-like domain / SIR2-like domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Shen ZF / Lin QP / Fu TM | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Assembly-mediated activation of the SIR2-HerA supramolecular complex for anti-phage defense. 著者: Zhangfei Shen / Qingpeng Lin / Xiao-Yuan Yang / Elizabeth Fosuah / Tian-Min Fu /  要旨: SIR2-HerA, a bacterial two-protein anti-phage defense system, induces bacterial death by depleting NAD upon phage infection. Biochemical reconstitution of SIR2, HerA, and the SIR2-HerA complex ...SIR2-HerA, a bacterial two-protein anti-phage defense system, induces bacterial death by depleting NAD upon phage infection. Biochemical reconstitution of SIR2, HerA, and the SIR2-HerA complex reveals a dynamic assembly process. Unlike other ATPases, HerA can form various oligomers, ranging from dimers to nonamers. When assembled with SIR2, HerA forms a hexamer and converts SIR2 from a nuclease to an NAD hydrolase, representing an unexpected regulatory mechanism mediated by protein assembly. Furthermore, high concentrations of ATP can inhibit NAD hydrolysis by the SIR2-HerA complex. Cryo-EM structures of the SIR2-HerA complex reveal a giant supramolecular assembly up to 1 MDa, with SIR2 as a dodecamer and HerA as a hexamer, crucial for anti-phage defense. Unexpectedly, the HerA hexamer resembles a spiral staircase and exhibits helicase activities toward dual-forked DNA. Together, we reveal the supramolecular assembly of SIR2-HerA as a unique mechanism for switching enzymatic activities and bolstering anti-phage defense strategies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40860.map.gz emd_40860.map.gz | 1.3 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40860-v30.xml emd-40860-v30.xml emd-40860.xml emd-40860.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40860_fsc.xml emd_40860_fsc.xml | 24.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40860.png emd_40860.png | 71.9 KB | ||
| Filedesc metadata |  emd-40860.cif.gz emd-40860.cif.gz | 5.4 KB | ||
| その他 |  emd_40860_additional_1.map.gz emd_40860_additional_1.map.gz emd_40860_half_map_1.map.gz emd_40860_half_map_1.map.gz emd_40860_half_map_2.map.gz emd_40860_half_map_2.map.gz | 1.2 GB 1.3 GB 1.3 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40860 http://ftp.pdbj.org/pub/emdb/structures/EMD-40860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40860 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40860_validation.pdf.gz emd_40860_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40860_full_validation.pdf.gz emd_40860_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_40860_validation.xml.gz emd_40860_validation.xml.gz | 34.2 KB | 表示 | |
| CIF形式データ |  emd_40860_validation.cif.gz emd_40860_validation.cif.gz | 44.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40860 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40860 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40860.map.gz / 形式: CCP4 / 大きさ: 1.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40860.map.gz / 形式: CCP4 / 大きさ: 1.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.4495 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
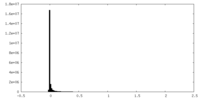
-追加マップ: #1
| ファイル | emd_40860_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
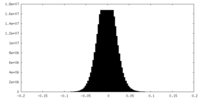
-ハーフマップ: #2
| ファイル | emd_40860_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
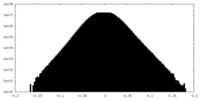
-ハーフマップ: #1
| ファイル | emd_40860_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E. coli dodecamer SIR2
| 全体 | 名称: E. coli dodecamer SIR2 |
|---|---|
| 要素 |
|
-超分子 #1: E. coli dodecamer SIR2
| 超分子 | 名称: E. coli dodecamer SIR2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: Six dimeric SIR2 form a dodecamer |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: SIR2-like domain-containing protein
| 分子 | 名称: SIR2-like domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46.817664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSIYQGGNKL NEDDFRSHVY SLCQLDNVGV LLGAGASVGC GGKTMKDVWK SFKQNYPELL GALIDKYLLV SQIDSDNNLV NVELLIDEA TKFLSVAKTR RCEDEEEEFR KILSSLYKEV TKAALLTGEQ FREKNQGKKD AFKYHKELIS KLISNRQPGQ S APAIFTTN ...文字列: MSIYQGGNKL NEDDFRSHVY SLCQLDNVGV LLGAGASVGC GGKTMKDVWK SFKQNYPELL GALIDKYLLV SQIDSDNNLV NVELLIDEA TKFLSVAKTR RCEDEEEEFR KILSSLYKEV TKAALLTGEQ FREKNQGKKD AFKYHKELIS KLISNRQPGQ S APAIFTTN YDLALEWAAE DLGIQLFNGF SGLHTRQFYP QNFDLAFRNV NAKGEARFGH YHAYLYKLHG SLTWYQNDSL TV NEVSASQ AYDEYINDII NKDDFYRGQH LIYPGANKYS HTIGFVYGEM FRRFGEFISK PQTALFINGF GFGDYHINRI ILG ALLNPS FHVVIYYPEL KEAITKVSKG GGSEAEKAIV TLKNMAFNQV TVVGGGSKAY FNSFVEHLPY PVLFPRDNIV DELV EAIAN LSKGEGNVPF UniProtKB: SIR2-like domain-containing protein |
-分子 #2: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 12 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X