+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a bacterial gasdermin slinky-like oligomer | |||||||||
 マップデータ マップデータ | EM map of Vitiosangium bGSDM slinky oligomer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | viral mimicry / gasdermin / caspase / autoinhibition / pyroptosis / bats / immunity / cell death / IMMUNE SYSTEM | |||||||||
| 機能・相同性 | defense response to virus / plasma membrane / cytoplasm / Gasdermin bGSDM 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Vitiosangium sp. GDMCC 1.1324 (バクテリア) Vitiosangium sp. GDMCC 1.1324 (バクテリア) | |||||||||
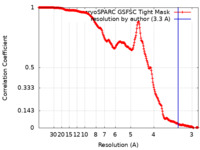
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Johnson AG / Mayer ML / Kranzusch PJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2023 タイトル: Structure and assembly of a bacterial gasdermin pore. 著者: Alex G Johnson / Megan L Mayer / Stefan L Schaefer / Nora K McNamara-Bordewick / Gerhard Hummer / Philip J Kranzusch /   要旨: In response to pathogen infection, gasdermin (GSDM) proteins form membrane pores that induce a host cell death process called pyroptosis. Studies of human and mouse GSDM pores reveal the functions ...In response to pathogen infection, gasdermin (GSDM) proteins form membrane pores that induce a host cell death process called pyroptosis. Studies of human and mouse GSDM pores reveal the functions and architectures of 24-33 protomers assemblies, but the mechanism and evolutionary origin of membrane targeting and GSDM pore formation remain unknown. Here we determine a structure of a bacterial GSDM (bGSDM) pore and define a conserved mechanism of pore assembly. Engineering a panel of bGSDMs for site-specific proteolytic activation, we demonstrate that diverse bGSDMs form distinct pore sizes that range from smaller mammalian-like assemblies to exceptionally large pores containing >50 protomers. We determine a 3.3 Å cryo-EM structure of a bGSDM in an active slinky-like oligomeric conformation and analyze bGSDM pores in a native lipid environment to create an atomic-level model of a full 52-mer bGSDM pore. Combining our structural analysis with molecular dynamics simulations and cellular assays, our results support a stepwise model of GSDM pore assembly and suggest that a covalently bound palmitoyl can leave a hydrophobic sheath and insert into the membrane before formation of the membrane-spanning β-strand regions. These results reveal the diversity of GSDM pores found in nature and explain the function of an ancient post-translational modification in enabling programmed host cell death. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40570.map.gz emd_40570.map.gz | 881.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40570-v30.xml emd-40570-v30.xml emd-40570.xml emd-40570.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40570_fsc.xml emd_40570_fsc.xml | 21.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40570.png emd_40570.png | 46 KB | ||
| Filedesc metadata |  emd-40570.cif.gz emd-40570.cif.gz | 6.4 KB | ||
| その他 |  emd_40570_half_map_1.map.gz emd_40570_half_map_1.map.gz emd_40570_half_map_2.map.gz emd_40570_half_map_2.map.gz | 929.2 MB 929.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40570 http://ftp.pdbj.org/pub/emdb/structures/EMD-40570 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40570 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40570 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40570_validation.pdf.gz emd_40570_validation.pdf.gz | 938.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40570_full_validation.pdf.gz emd_40570_full_validation.pdf.gz | 938.4 KB | 表示 | |
| XML形式データ |  emd_40570_validation.xml.gz emd_40570_validation.xml.gz | 31.3 KB | 表示 | |
| CIF形式データ |  emd_40570_validation.cif.gz emd_40570_validation.cif.gz | 41.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40570 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40570 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40570 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40570 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8sl0MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40570.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40570.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM map of Vitiosangium bGSDM slinky oligomer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.431 Å | ||||||||||||||||||||||||||||||||||||
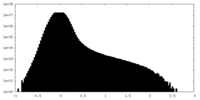
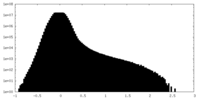
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: EM half map A of Vitiosangium bGSDM slinky oligomer
| ファイル | emd_40570_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map A of Vitiosangium bGSDM slinky oligomer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
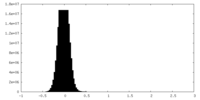
| 密度ヒストグラム |
-ハーフマップ: EM half map B of Vitiosangium bGSDM slinky oligomer
| ファイル | emd_40570_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half map B of Vitiosangium bGSDM slinky oligomer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
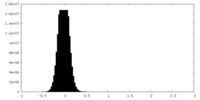
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vitiosangium bGSDM in an active slinky-like oligomeric conformation
| 全体 | 名称: Vitiosangium bGSDM in an active slinky-like oligomeric conformation |
|---|---|
| 要素 |
|
-超分子 #1: Vitiosangium bGSDM in an active slinky-like oligomeric conformation
| 超分子 | 名称: Vitiosangium bGSDM in an active slinky-like oligomeric conformation タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Vitiosangium sp. GDMCC 1.1324 (バクテリア) Vitiosangium sp. GDMCC 1.1324 (バクテリア) |
-分子 #1: Gasdermin bGSDM
| 分子 | 名称: Gasdermin bGSDM / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vitiosangium sp. GDMCC 1.1324 (バクテリア) Vitiosangium sp. GDMCC 1.1324 (バクテリア) |
| 分子量 | 理論値: 25.566246 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGLCSDPAIT YLKRLGYNVV RLPREGIQPL HLLGQQRGTV EYLGSLEKLI TQPPSEPPAI TRDQAAAGIN GQKTENLSFS IGINILKSV LAQFGAGAGI EAQYNQARKV RFEFSNVLAD SVEPLAVGQF LKMAEVDADN PVLKQYVLGN GRLYVITQVI K SNEFTVAA ...文字列: SGLCSDPAIT YLKRLGYNVV RLPREGIQPL HLLGQQRGTV EYLGSLEKLI TQPPSEPPAI TRDQAAAGIN GQKTENLSFS IGINILKSV LAQFGAGAGI EAQYNQARKV RFEFSNVLAD SVEPLAVGQF LKMAEVDADN PVLKQYVLGN GRLYVITQVI K SNEFTVAA EKSGGGSIQL DVPEIQKVVG GKLKVEASVS SQSTVTYKGE KQLVFGFKCF EIGVKNGEIT LFASQLVPR UniProtKB: Gasdermin bGSDM |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 150 mM NaCl, 20 mM HEPES-HOH (pH 7.5), 5.15 mM DDMAB | ||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 27.0 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | The sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 8156 / 平均電子線量: 51.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)