+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Spizellomyces punctatus Fanzor (SpuFz) in complex with omega RNA and target DNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Fanzor / Eukaryotic / RNA-guided / nuclease / Gene editing / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報carbohydrate transmembrane transporter activity / maltose binding / maltose transport / maltodextrin transmembrane transport / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing 類似検索 - 分子機能 | |||||||||
| 生物種 |  Spizellomyces punctatus (菌類) / synthetic construct (人工物) Spizellomyces punctatus (菌類) / synthetic construct (人工物) | |||||||||
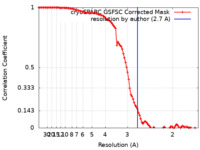
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Xu P / Saito M / Zhang F | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Fanzor is a eukaryotic programmable RNA-guided endonuclease. 著者: Makoto Saito / Peiyu Xu / Guilhem Faure / Samantha Maguire / Soumya Kannan / Han Altae-Tran / Sam Vo / AnAn Desimone / Rhiannon K Macrae / Feng Zhang /  要旨: RNA-guided systems, which use complementarity between a guide RNA and target nucleic acid sequences for recognition of genetic elements, have a central role in biological processes in both ...RNA-guided systems, which use complementarity between a guide RNA and target nucleic acid sequences for recognition of genetic elements, have a central role in biological processes in both prokaryotes and eukaryotes. For example, the prokaryotic CRISPR-Cas systems provide adaptive immunity for bacteria and archaea against foreign genetic elements. Cas effectors such as Cas9 and Cas12 perform guide-RNA-dependent DNA cleavage. Although a few eukaryotic RNA-guided systems have been studied, including RNA interference and ribosomal RNA modification, it remains unclear whether eukaryotes have RNA-guided endonucleases. Recently, a new class of prokaryotic RNA-guided systems (termed OMEGA) was reported. The OMEGA effector TnpB is the putative ancestor of Cas12 and has RNA-guided endonuclease activity. TnpB may also be the ancestor of the eukaryotic transposon-encoded Fanzor (Fz) proteins, raising the possibility that eukaryotes are also equipped with CRISPR-Cas or OMEGA-like programmable RNA-guided endonucleases. Here we report the biochemical characterization of Fz, showing that it is an RNA-guided DNA endonuclease. We also show that Fz can be reprogrammed for human genome engineering applications. Finally, we resolve the structure of Spizellomyces punctatus Fz at 2.7 Å using cryogenic electron microscopy, showing the conservation of core regions among Fz, TnpB and Cas12, despite diverse cognate RNA structures. Our results show that Fz is a eukaryotic OMEGA system, demonstrating that RNA-guided endonucleases are present in all three domains of life. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40184.map.gz emd_40184.map.gz | 90.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40184-v30.xml emd-40184-v30.xml emd-40184.xml emd-40184.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40184_fsc.xml emd_40184_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40184.png emd_40184.png | 85 KB | ||
| その他 |  emd_40184_additional_1.map.gz emd_40184_additional_1.map.gz emd_40184_half_map_1.map.gz emd_40184_half_map_1.map.gz emd_40184_half_map_2.map.gz emd_40184_half_map_2.map.gz | 97.1 MB 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40184 http://ftp.pdbj.org/pub/emdb/structures/EMD-40184 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40184 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40184 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40184_validation.pdf.gz emd_40184_validation.pdf.gz | 703.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40184_full_validation.pdf.gz emd_40184_full_validation.pdf.gz | 703.5 KB | 表示 | |
| XML形式データ |  emd_40184_validation.xml.gz emd_40184_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_40184_validation.cif.gz emd_40184_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40184 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40184 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40184 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40184 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gkhMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40184.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40184.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.825 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_40184_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
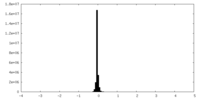
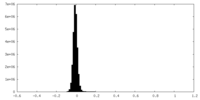
| 投影像・断面図 |
| ||||||||||||
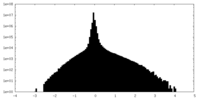
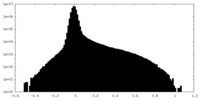
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40184_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
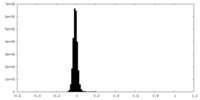
| 投影像・断面図 |
| ||||||||||||
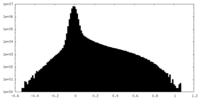
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40184_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SpuFz-wRNA-tgDNA complex
| 全体 | 名称: SpuFz-wRNA-tgDNA complex |
|---|---|
| 要素 |
|
-超分子 #1: SpuFz-wRNA-tgDNA complex
| 超分子 | 名称: SpuFz-wRNA-tgDNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Spizellomyces punctatus (菌類) Spizellomyces punctatus (菌類) |
-分子 #1: DNA (5'-D(P*CP*GP*GP*TP*AP*CP*CP*CP*GP*GP*GP*CP*AP*TP*A)-3')
| 分子 | 名称: DNA (5'-D(P*CP*GP*GP*TP*AP*CP*CP*CP*GP*GP*GP*CP*AP*TP*A)-3') タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.594986 KDa |
| 配列 | 文字列: (DC)(DG)(DG)(DT)(DA)(DC)(DC)(DC)(DG)(DG) (DG)(DC)(DA)(DT)(DA) |
-分子 #3: DNA (35-MER)
| 分子 | 名称: DNA (35-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 10.737919 KDa |
| 配列 | 文字列: (DT)(DT)(DG)(DA)(DT)(DT)(DT)(DC)(DA)(DT) (DA)(DA)(DC)(DC)(DT)(DA)(DT)(DA)(DG)(DA) (DT)(DA)(DT)(DG)(DC)(DC)(DC)(DG)(DG) (DG)(DT)(DA)(DC)(DC)(DG) |
-分子 #2: Maltodextrin-binding protein (Fragment),OrfB_Zn_ribbon domain-con...
| 分子 | 名称: Maltodextrin-binding protein (Fragment),OrfB_Zn_ribbon domain-containing protein タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Spizellomyces punctatus (菌類) Spizellomyces punctatus (菌類) |
| 分子量 | 理論値: 118.029922 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSSHHHHHH HHHHGSSMKI EEGKLVIWIN GDKGYNGLAE VGKKFEKDTG IKVTVEHPDK LEEKFPQVAA TGDGPDIIFW AHDRFGGYA QSGLLAEITP DKAFQDKLYP FTWDAVRYNG KLIAYPIAVE ALSLIYNKDL LPNPPKTWEE IPALDKELKA K GKSALMFN ...文字列: MKSSHHHHHH HHHHGSSMKI EEGKLVIWIN GDKGYNGLAE VGKKFEKDTG IKVTVEHPDK LEEKFPQVAA TGDGPDIIFW AHDRFGGYA QSGLLAEITP DKAFQDKLYP FTWDAVRYNG KLIAYPIAVE ALSLIYNKDL LPNPPKTWEE IPALDKELKA K GKSALMFN LQEPYFTWPL IAADGGYAFK YENGKYDIKD VGVDNAGAKA GLTFLVDLIK NKHMNADTDY SIAEAAFNKG ET AMTINGP WAWSNIDTSK VNYGVTVLPT FKGQPSKPFV GVLSAGINAA SPNKELAKEF LENYLLTDEG LEAVNKDKPL GAV ALKSYE EELAKDPRIA ATMENAQKGE IMPNIPQMSA FWYAVRTAVI NAASGRQTVD EALKDAQTGS ENLYFQSNAP PKKK QKLER LKKLDKPTLH TCNKTSFAKA FLPNETYRQR LLDYIAIIHQ LADHASHALK FYILSTSTSS FPVVHEDTIE AILYL LNKG EAWHPRKEAK KAWRDCLLPY VQRYCQIVGF IHPNLRGEQQ SINYLTVSMM TNLKVNVQEH FMQMLLRYIN LRFDVK GQK QRLPPKSDAR KAFFTRLRYL KSVFLFDVVP ELEFLDDLTP LESEVLEEIW SLDLPFLPND PLAYAIVADP MSFFPAY CK LSGLYEQYGF QRFSAIPLRR SLIQSHVRID TIILYQHILC ITRRDAETVE KDDLWMRVCN LCTKAFRSRC GMHFEGSI T TDGASVSVYL KHPEADKYGK RGARKSANTV AAEVKALYVE NNLPACRAAE NVVVIDPNKR DILYCQDSNG TTFRYTANQ RAVETGSRRF AKRREAMKEE AGVDLIESRI PSHKTMNLMD FTRYLLVRRA DWDRRKEFYS HPAHTRWKWH SFINRQKSES DLISNMRNK YGENFTVVMG DWSDAGRTAR FQTSSKTKGW RTLFKRNRID CFLLDEYKTS SVCPRCSSSE FVEKKFKTRP H SRPWRRRE GKIEKVHGLL GCTNPNCLQQ AWTSGMRYWN RDMLSTCNML LIVRSMLDGH GRPEVFSRSV PAVA UniProtKB: Maltodextrin-binding protein, Uncharacterized protein |
-分子 #4: RNA (78-MER)
| 分子 | 名称: RNA (78-MER) / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Spizellomyces punctatus (菌類) Spizellomyces punctatus (菌類) |
| 分子量 | 理論値: 25.050807 KDa |
| 配列 | 文字列: UCCGAGCCGG UAGUCGCGCG GUUCAAUCCC UGGUGCGGGU GCUAGUGCAC CGGCUCCGCA CUAUCUAUAG GUUAUGAA |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 3 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 51.41 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X