+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo EM structure of Komagataella phaffii Rat1-Rai1-Rtt103 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transcription termination / RNA polymerase II / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA 5'-diphosphatase activity / NAD-cap decapping / 5'-3' RNA exonuclease activity / nuclease activity / mRNA 3'-end processing / exonuclease activity / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / nuclear-transcribed mRNA catabolic process / RNA polymerase II C-terminal domain binding / DNA-templated transcription termination ...mRNA 5'-diphosphatase activity / NAD-cap decapping / 5'-3' RNA exonuclease activity / nuclease activity / mRNA 3'-end processing / exonuclease activity / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / nuclear-transcribed mRNA catabolic process / RNA polymerase II C-terminal domain binding / DNA-templated transcription termination / mRNA processing / rRNA processing / 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ / nucleic acid binding / nucleotide binding / RNA binding / nucleus / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Komagataella phaffii (菌類) Komagataella phaffii (菌類) | |||||||||
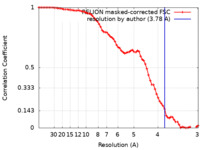
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.78 Å | |||||||||
 データ登録者 データ登録者 | Yanagisawa T / Murayama Y / Ehara H / Sekine SI | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis of eukaryotic transcription termination by the Rat1 exonuclease complex. 著者: Tatsuo Yanagisawa / Yuko Murayama / Haruhiko Ehara / Mie Goto / Mari Aoki / Shun-Ichi Sekine /  要旨: The 5´-3´ exoribonuclease Rat1/Xrn2 is responsible for the termination of eukaryotic mRNA transcription by RNAPII. Rat1 forms a complex with its partner proteins, Rai1 and Rtt103, and acts as a ...The 5´-3´ exoribonuclease Rat1/Xrn2 is responsible for the termination of eukaryotic mRNA transcription by RNAPII. Rat1 forms a complex with its partner proteins, Rai1 and Rtt103, and acts as a "torpedo" to bind transcribing RNAPII and dissociate DNA/RNA from it. Here we report the cryo-electron microscopy structures of the Rat1-Rai1-Rtt103 complex and three Rat1-Rai1-associated RNAPII complexes (type-1, type-1b, and type-2) from the yeast, Komagataella phaffii. The Rat1-Rai1-Rtt103 structure revealed that Rat1 and Rai1 form a heterotetramer with a single Rtt103 bound between two Rai1 molecules. In the type-1 complex, Rat1-Rai1 forms a heterodimer and binds to the RNA exit site of RNAPII to extract RNA into the Rat1 exonuclease active site. This interaction changes the RNA path in favor of termination (the "pre-termination" state). The type-1b and type-2 complexes have no bound DNA/RNA, likely representing the "post-termination" states. These structures illustrate the termination mechanism of eukaryotic mRNA transcription. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39211.map.gz emd_39211.map.gz | 23.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39211-v30.xml emd-39211-v30.xml emd-39211.xml emd-39211.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39211_fsc.xml emd_39211_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39211.png emd_39211.png | 87.6 KB | ||
| Filedesc metadata |  emd-39211.cif.gz emd-39211.cif.gz | 6.7 KB | ||
| その他 |  emd_39211_half_map_1.map.gz emd_39211_half_map_1.map.gz emd_39211_half_map_2.map.gz emd_39211_half_map_2.map.gz | 23.4 MB 23.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39211 http://ftp.pdbj.org/pub/emdb/structures/EMD-39211 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39211 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39211 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39211_validation.pdf.gz emd_39211_validation.pdf.gz | 878.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39211_full_validation.pdf.gz emd_39211_full_validation.pdf.gz | 877.7 KB | 表示 | |
| XML形式データ |  emd_39211_validation.xml.gz emd_39211_validation.xml.gz | 12.7 KB | 表示 | |
| CIF形式データ |  emd_39211_validation.cif.gz emd_39211_validation.cif.gz | 17.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39211 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39211 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39211 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39211 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39211.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39211.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.494 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_39211_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_39211_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Komagataella phaffii Rat1-Rai1-Rtt103 complex
| 全体 | 名称: Komagataella phaffii Rat1-Rai1-Rtt103 complex |
|---|---|
| 要素 |
|
-超分子 #1: Komagataella phaffii Rat1-Rai1-Rtt103 complex
| 超分子 | 名称: Komagataella phaffii Rat1-Rai1-Rtt103 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii (菌類) Komagataella phaffii (菌類) |
-分子 #1: 5'-3' exoribonuclease
| 分子 | 名称: 5'-3' exoribonuclease / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii (菌類) Komagataella phaffii (菌類) |
| 分子量 | 理論値: 115.314461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGVPALFRWL SRKYPKIISP VIQDEDVDID GESRPTRYED PNPNGELDNL YLDMNGIVHP CSHPEHKPVP ETEDEMMLDV FAYTENVIM MARPRKVIYI AVDGVAPRAK MNQQRSRRFR SAQDAKDANE KKAAELKEME KKGEIIDDAI KNKKTWDSNA I TPGTPFMH ...文字列: MGVPALFRWL SRKYPKIISP VIQDEDVDID GESRPTRYED PNPNGELDNL YLDMNGIVHP CSHPEHKPVP ETEDEMMLDV FAYTENVIM MARPRKVIYI AVDGVAPRAK MNQQRSRRFR SAQDAKDANE KKAAELKEME KKGEIIDDAI KNKKTWDSNA I TPGTPFMH RLADSLRYWA AYKLTTDPGW SGIEVIISDA SVPGEGEHKI MSYVRSLRSS PKHDPNTTHC IYGLAAALIF LG LATHEPH FKILREDVFA QDKKSYSLQD QLRMTDIERQ ELKDKKTPFL WLHLNILREY LQIELNVPGL SFPFDLEKSI DDW VFICFF CGNDFLPHLP SLDVRDNSIT TLVTIWKQIL PTMKGYLTTD GYLNLPAVER LLAELAKKED YIFRKRYEDE KRSL ENQKR RKLAQEQSSA RSQNAPNIST GKDKAPLTPN QNIPLYTTSG ESVGIKMTDS EMVNNSALIT KANEANKSIA ELLKQ NLQN EINKKRKISN EEQEVVKESV EEVVEEEDDV LVTSDPEDSS TEILIPKNEE IRLWEPGYRK RYYETKFHTK DPQKVK KIA RNMVQKYIEG VSWVLLYYYQ GCPSWNWYYP YHYAPFAADF VNLSELKIEF VEGTPFRPYE QLMSVLPAAS SHNLPDV FR SLMSDANSEI IDFYPEEFPL DMNGKKVIWQ AIPLLPFIDE NRLLKAVQSK YDQLTEDEKF RNTNRSEILV LGRSHSHY P TLVKELYEEG KDSYEFQVDS SGVSGVAIKL QSFDRSGVLR LPVKQLEGYR HYPDISNRDF LMVEFKQLPK SHAKSMILS GLIPHLRRLT QEDKDSILYG GTNFYGRNRF SPEENADFKQ YIGPHGKSQY LPRQGGYKAF IQIHSDEAKG HRHGIYHGGS HTETEFRRG GGYHQHGNRG GRGGYQGNQG YQANSGGYQN SYQGSYQGGY RGGYQGGSQG RYQAGYQSGY QGGYQGEYKN G YQGGYQGN QGNQGYNRQT YNASKSGTLP MKRRHNSGPS SGLEVLFQ UniProtKB: 5'-3' exoribonuclease |
-分子 #2: Decapping nuclease
| 分子 | 名称: Decapping nuclease / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii (菌類) Komagataella phaffii (菌類) |
| 分子量 | 理論値: 44.595996 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGMSKEKIL PLAARSKKAM LRQPKQVAYF SRDLNYKTHP DRSNLSYYYL PDGDIDNSID LSVGSKHFLL GDSVELSKLD PILLALKEI EKESGAKTKD RIITWRGIMR KLLTLPYDSE EDFVLDVVSF DGQLFIQFNV PYLKSKDVQK QGDTEFHKKL Q FSGYKFEK ...文字列: GPGMSKEKIL PLAARSKKAM LRQPKQVAYF SRDLNYKTHP DRSNLSYYYL PDGDIDNSID LSVGSKHFLL GDSVELSKLD PILLALKEI EKESGAKTKD RIITWRGIMR KLLTLPYDSE EDFVLDVVSF DGQLFIQFNV PYLKSKDVQK QGDTEFHKKL Q FSGYKFEK MATLPKPWPE CTRKEIDSRA KSKCNNIEQY GAIVRTGISR IKILIGGAVA CTADYYDEND PLSRYIELKT TR TINQYKD MIAFEKKLFR TWAQCFLLGI PKIIYGFRDD NCILRTVEEF STNDIPLMVK NNPLNEQPKK ENCYMSSINF YGA VVEWLN ESVKDDQVWK LSYAKRNRQY LVLKEVTDEN EKQQIVDSAI PAWFKEWRSE LRNSEGNI UniProtKB: Decapping nuclease |
-分子 #3: Exonuclease Rat1p and Rai1p interacting protein
| 分子 | 名称: Exonuclease Rat1p and Rai1p interacting protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii (菌類) Komagataella phaffii (菌類) |
| 分子量 | 理論値: 42.27293 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYSKDIVLE KLASLEETQI SIQSIGQWCL FHHRHAPETV AIWSEFVGST QTKKLAGLYL ANEIIQQSRA KRKTTFLDEF AKVLPSTLE QIYPSMISTH QAKLKRIIDV WSQRKIFDSN LIHRLYQSIQ DQKAYNGLSS SSSNSPPINS GDLAPEISSL S TLFNKIST ...文字列: MSYSKDIVLE KLASLEETQI SIQSIGQWCL FHHRHAPETV AIWSEFVGST QTKKLAGLYL ANEIIQQSRA KRKTTFLDEF AKVLPSTLE QIYPSMISTH QAKLKRIIDV WSQRKIFDSN LIHRLYQSIQ DQKAYNGLSS SSSNSPPINS GDLAPEISSL S TLFNKIST LKSSTSLVVN QINEQYSSLF DSETLPGTDI YLKQLGDLST LITSARSKSQ ETQELRESII NELKKLIQVQ ES WITKDAE SSGSLDEKLA TVQQKESELK EFINDVEEED GVPQYAASSD EEGDENVSKK RKMESPPTET VDSGEQPTNP VHP QLSSIL ESLSRTTGLT PEPASTSDES ATATQKQDET PVSSSINPAL ASLLSKLNGL EVLFQ UniProtKB: Exonuclease Rat1p and Rai1p interacting protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 75 % / チャンバー内温度: 283 K / 装置: LEICA EM GP / 詳細: EMGP2. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 61.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)