+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of HCoV-HKU1A spike in the functionally anchored-3up conformation with 3TMPRSS2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HKU1A / spike / TMPRSS2 / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transmembrane protease serine 2 / protein autoprocessing / Attachment and Entry / serine-type peptidase activity / endocytosis involved in viral entry into host cell / viral translation / Induction of Cell-Cell Fusion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / Attachment and Entry / positive regulation of viral entry into host cell ...transmembrane protease serine 2 / protein autoprocessing / Attachment and Entry / serine-type peptidase activity / endocytosis involved in viral entry into host cell / viral translation / Induction of Cell-Cell Fusion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / Attachment and Entry / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / fusion of virus membrane with host plasma membrane / serine-type endopeptidase activity / fusion of virus membrane with host endosome membrane / viral envelope / host cell plasma membrane / virion membrane / proteolysis / extracellular exosome / extracellular region / nucleoplasm / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human coronavirus HKU1 (ウイルス) / Human coronavirus HKU1 (ウイルス) /  Human coronavirus HKU1 (isolate N1) (ウイルス) / Human coronavirus HKU1 (isolate N1) (ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.09 Å | |||||||||
 データ登録者 データ登録者 | Lu YC / Zhang X / Wang HF / Liu XC / Sun L / Yang HT | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: TMPRSS2 and glycan receptors synergistically facilitate coronavirus entry. 著者: Haofeng Wang / Xiaoce Liu / Xiang Zhang / Zhuoqian Zhao / Yuchi Lu / Dingzhe Pu / Zeyang Zhang / Jie Chen / Yajie Wang / Mengfei Li / Xuxue Dong / Yinkai Duan / Yujia He / Qiyu Mao / Hangtian ...著者: Haofeng Wang / Xiaoce Liu / Xiang Zhang / Zhuoqian Zhao / Yuchi Lu / Dingzhe Pu / Zeyang Zhang / Jie Chen / Yajie Wang / Mengfei Li / Xuxue Dong / Yinkai Duan / Yujia He / Qiyu Mao / Hangtian Guo / Haoran Sun / Yihan Zhou / Qi Yang / Yan Gao / Xiuna Yang / Hongzhi Cao / Luke Guddat / Lei Sun / Zihe Rao / Haitao Yang /   要旨: The entry of coronaviruses is initiated by spike recognition of host cellular receptors, involving proteinaceous and/or glycan receptors. Recently, TMPRSS2 was identified as the proteinaceous ...The entry of coronaviruses is initiated by spike recognition of host cellular receptors, involving proteinaceous and/or glycan receptors. Recently, TMPRSS2 was identified as the proteinaceous receptor for HCoV-HKU1 alongside sialoglycan as a glycan receptor. However, the underlying mechanisms for viral entry remain unknown. Here, we investigated the HCoV-HKU1C spike in the inactive, glycan-activated, and functionally anchored states, revealing that sialoglycan binding induces a conformational change of the NTD and promotes the neighboring RBD of the spike to open for TMPRSS2 recognition, exhibiting a synergistic mechanism for the entry of HCoV-HKU1. The RBD of HCoV-HKU1 features an insertion subdomain that recognizes TMPRSS2 through three previously undiscovered interfaces. Furthermore, structural investigation of HCoV-HKU1A in combination with mutagenesis and binding assays confirms a conserved receptor recognition pattern adopted by HCoV-HKU1. These studies advance our understanding of the complex viral-host interactions during entry, laying the groundwork for developing new therapeutics against coronavirus-associated diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39025.map.gz emd_39025.map.gz | 373 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39025-v30.xml emd-39025-v30.xml emd-39025.xml emd-39025.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_39025.png emd_39025.png | 90.6 KB | ||
| Filedesc metadata |  emd-39025.cif.gz emd-39025.cif.gz | 6.7 KB | ||
| その他 |  emd_39025_half_map_1.map.gz emd_39025_half_map_1.map.gz emd_39025_half_map_2.map.gz emd_39025_half_map_2.map.gz | 391.4 MB 391.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39025 http://ftp.pdbj.org/pub/emdb/structures/EMD-39025 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39025 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39025 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39025_validation.pdf.gz emd_39025_validation.pdf.gz | 663.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39025_full_validation.pdf.gz emd_39025_full_validation.pdf.gz | 663.2 KB | 表示 | |
| XML形式データ |  emd_39025_validation.xml.gz emd_39025_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_39025_validation.cif.gz emd_39025_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39025 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39025 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39025 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39025 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8y7xMC  8y7yC  8y87C  8y88C  8y89C  8y8aC  8y8bC  8y8cC  8y8dC  8y8eC  8y8fC  8y8gC  8y8hC  8y8iC  8y8jC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39025.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39025.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_39025_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
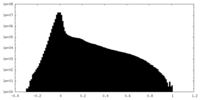
| 投影像・断面図 |
| ||||||||||||
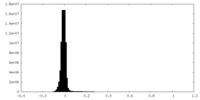
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_39025_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
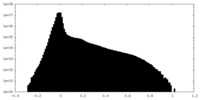
| 投影像・断面図 |
| ||||||||||||
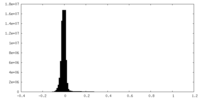
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HKU1A-TMPRSS2 complex
| 全体 | 名称: HKU1A-TMPRSS2 complex |
|---|---|
| 要素 |
|
-超分子 #1: HKU1A-TMPRSS2 complex
| 超分子 | 名称: HKU1A-TMPRSS2 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (ウイルス) Human coronavirus HKU1 (ウイルス) |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human coronavirus HKU1 (isolate N1) (ウイルス) Human coronavirus HKU1 (isolate N1) (ウイルス)株: isolate N1 |
| 分子量 | 理論値: 141.235328 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VIGDFNCTNF AINDLNTTVP RISEYVVDVS YGLGTYYILD RVYLNTTILF TGYFPKSGAN FRDLSLKGTT YLSTLWYQKP FLSDFNNGI FSRVKNTKLY VNKTLYSEFS TIVIGSVFIN NSYTIVVQPH NGVLEITACQ YTMCEYPHTI CKSKGSSRNE S WHFDKSEP ...文字列: VIGDFNCTNF AINDLNTTVP RISEYVVDVS YGLGTYYILD RVYLNTTILF TGYFPKSGAN FRDLSLKGTT YLSTLWYQKP FLSDFNNGI FSRVKNTKLY VNKTLYSEFS TIVIGSVFIN NSYTIVVQPH NGVLEITACQ YTMCEYPHTI CKSKGSSRNE S WHFDKSEP LCLFKKNFTY NVSTDWLYFH FYQERGTFYA YYADSGMPTT FLFSLYLGTL LSHYYVLPLT CNAISSNTDN ET LQYWVTP LSKRQYLLKF DNRGVITNAV DCSSSFFSEI QCKTKSLLPN TGVYDLSGFT VKPVATVHRR IPDLPDCDID KWL NNFNVP SPLNWERKIF SNCNFNLSTL LRLVHTDSFS CNNFDESKIY GSCFKSIVLD KFAIPNSRRS DLQLGSSGFL QSSN YKIDT TSSSCQLYYS LPAINVTINN YNPSSWNRRY GFNNFNLSSH SVVYSRYCFS VNNTFCPCAK PSFASSCKSH KPPSA SCPI GTNYRSCEST TVLDHTDWCR CSCLPDPITA YDPRSCSQKK SLVGVGEHCA GFGVDEEKCG VLDGSYNVSC LCSTDA FLG WSYDTCVSNN RCNIFSNFIL NGINSGTTCS NDLLQPNTEV FTDVCVDYDL YGITGQGIFK EVSAVYYNSW QNLLYDS NG NIIGFKDFVT NKTYNIFPCY AGRVSAAFHQ NASSLALLYR NLKCSYVLNN ISLTTQPYFD SYLGCVFNAD NLTDYSVS S CALRMGSGFC VDYNSPSSSS SGGSGSSISA SYRFVTFEPF NVSFVNDSIE SVGGLYEIKI PTNFTIVGQE EFIQTNSPK VTIDCSLFVC SNYAACHDLL SEYGTFCDNI NSILDEVNGL LDTTQLHVAD TLMQGVTLSS NLNTNLHFDV DNINFKSLVG CLGPHCGSS SRSFFEDLLF DKVKLSDVGF VEAYNNCTGG SEIRDLLCVQ SFNGIKVLPP ILSESQISGY TTAATVAAMF P PWSAAAGI PFSLNVQYRI NGLGVTMDVL NKNQKLIATA FNNALLSIQN GFSATNSALA KIQSVVNSNA QALNSLLQQL FN KFGAISS SLQEILSRLD PPEAQVQIDR LINGRLTALN AYVSQQLSDI SLVKFGAALA MEKVNECVKS QSPRINFCGN GNH ILSLVQ NAPYGLLFMH FSYKPISFKT VLVSPGLCIS GDVGIAPKQG YFIKHNDHWM FTGSSYYYPE PISDKNVVFM NTCS VNFTK APLVYLNHSV PKLSDFESEL SHWFKNQTSI APNLTLNLHT INATFLDLYY EMNLIQESIK SLN UniProtKB: Spike glycoprotein |
-分子 #2: Transmembrane protease serine 2
| 分子 | 名称: Transmembrane protease serine 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号: transmembrane protease serine 2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 42.283629 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSKCSNSGI ECDSSGTCIN PSNWCDGVSH CPGGEDENRC VRLYGPNFIL QVYSSQRKSW HPVCQDDWNE NYGRAACRDM GYKNNFYSS QGIVDDSGST SFMKLNTSAG NVDIYKKLYH SDACSSKAVV SLRCIACGVN LNDDDDKIVG GESALPGAWP W QVSLHVQN ...文字列: MGSKCSNSGI ECDSSGTCIN PSNWCDGVSH CPGGEDENRC VRLYGPNFIL QVYSSQRKSW HPVCQDDWNE NYGRAACRDM GYKNNFYSS QGIVDDSGST SFMKLNTSAG NVDIYKKLYH SDACSSKAVV SLRCIACGVN LNDDDDKIVG GESALPGAWP W QVSLHVQN VHVCGGSIIT PEWIVTAAHC VEKPLNNPWH WTAFAGILRQ SFMFYGAGYQ VEKVISHPNY DSKTKNNDIA LM KLQKPLT FNDLVKPVCL PNPGMMLQPE QLCWISGWGA TEEKGKTSEV LNAAKVLLIE TQRCNSRYVY DNLITPAMIC AGF LQGNVD SCQGDSGGPL VTSKNNIWWL IGDTSWGSGC AKAYRPGVYG NVMVFTDWIY RQMRADG UniProtKB: Transmembrane protease serine 2 |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 23 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.09 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 935674 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)