+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | structure of AtHKT1;1 in KCl at 2.8 Angstroms resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HKT / ion selectivity / salt tolerance / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報potassium ion transmembrane transporter activity / response to osmotic stress / sodium ion transport / response to salt stress / potassium ion transport / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Wang J / Su N / Guo J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Plant / 年: 2024 ジャーナル: Mol Plant / 年: 2024タイトル: Structures and ion transport mechanisms of plant high-affinity potassium transporters. 著者: Jiangqin Wang / Yanping Luo / Fan Ye / Zhong Jie Ding / Shao Jian Zheng / Shuai Qiao / Yong Wang / Jiangtao Guo / Wei Yang / Nannan Su /  要旨: Plant high-affinity K transporters (HKTs) mediate Na and K uptake, maintain Na/K homeostasis, and therefore play crucial roles in plant salt tolerance. In this study, we present cryoelectron ...Plant high-affinity K transporters (HKTs) mediate Na and K uptake, maintain Na/K homeostasis, and therefore play crucial roles in plant salt tolerance. In this study, we present cryoelectron microscopy structures of HKTs from two classes, class I HKT1;1 from Arabidopsis thaliana (AtHKT1;1) and class II HKT2;1 from Triticum aestivum (TaHKT2;1), in both Na- and K-bound states at 2.6- to 3.0-Å resolutions. Both AtHKT1;1 and TaHKT2;1 function as homodimers. Each HKT subunit consists of four tandem domain units (D1-D4) with a repeated K-channel-like M-P-M topology. In each subunit, D1-D4 assemble into an ion conduction pore with a pseudo-four-fold symmetry. Although both TaHKT2;1 and AtHKT1;1 have only one putative Na ion bound in the selectivity filter with a similar coordination pattern, the two HKTs display different K binding modes in the filter. TaHKT2;1 has three K ions bound in the selectivity filter, but AtHKT1;1 has only two K ions bound in the filter, which has a narrowed external entrance due to the presence of a Ser residue in the first filter motif. These structures, along with computational, mutational, and electrophysiological analyses, enable us to pinpoint key residues that are critical for the ion selectivity of HKTs. The findings provide new insights into the ion selectivity and ion transport mechanisms of plant HKTs and improve our understanding about how HKTs mediate plant salt tolerance and enhance crop growth. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37377.map.gz emd_37377.map.gz | 28.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37377-v30.xml emd-37377-v30.xml emd-37377.xml emd-37377.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_37377.png emd_37377.png | 65.3 KB | ||
| Filedesc metadata |  emd-37377.cif.gz emd-37377.cif.gz | 5.4 KB | ||
| その他 |  emd_37377_half_map_1.map.gz emd_37377_half_map_1.map.gz emd_37377_half_map_2.map.gz emd_37377_half_map_2.map.gz | 28 MB 28.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37377 http://ftp.pdbj.org/pub/emdb/structures/EMD-37377 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37377 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37377 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37377_validation.pdf.gz emd_37377_validation.pdf.gz | 899.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37377_full_validation.pdf.gz emd_37377_full_validation.pdf.gz | 899 KB | 表示 | |
| XML形式データ |  emd_37377_validation.xml.gz emd_37377_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_37377_validation.cif.gz emd_37377_validation.cif.gz | 12.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37377 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37377 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37377 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37377 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37377.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37377.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_37377_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
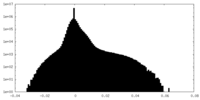
| 投影像・断面図 |
| ||||||||||||
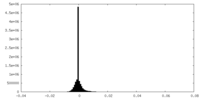
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37377_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
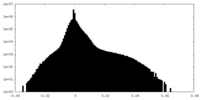
| 投影像・断面図 |
| ||||||||||||
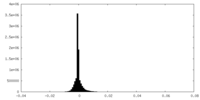
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : structure of AtHKT1;1 in KCl at 2.8 Angstroms resolution
| 全体 | 名称: structure of AtHKT1;1 in KCl at 2.8 Angstroms resolution |
|---|---|
| 要素 |
|
-超分子 #1: structure of AtHKT1;1 in KCl at 2.8 Angstroms resolution
| 超分子 | 名称: structure of AtHKT1;1 in KCl at 2.8 Angstroms resolution タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Sodium transporter HKT1
| 分子 | 名称: Sodium transporter HKT1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.504965 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDRVVAKIAK IRSQLTKLRS LFFLYFIYFL FFSFLGFLAL KITKPRTTSR PHDFDLFFTS VSAITVSSMS TVDMEVFSNT QLIFLTILM FLGGEIFTSF LNLYVSYFTK FVFPHNKIRH ILGSYNSDSS IEDRCDVETV TDYREGLIKI DERASKCLYS V VLSYHLVT ...文字列: MDRVVAKIAK IRSQLTKLRS LFFLYFIYFL FFSFLGFLAL KITKPRTTSR PHDFDLFFTS VSAITVSSMS TVDMEVFSNT QLIFLTILM FLGGEIFTSF LNLYVSYFTK FVFPHNKIRH ILGSYNSDSS IEDRCDVETV TDYREGLIKI DERASKCLYS V VLSYHLVT NLVGSVLLLV YVNFVKTARD VLSSKEISPL TFSVFTTVST FANCGFVPTN ENMIIFRKNS GLIWLLIPQV LM GNTLFPC FLVLLIWGLY KITKRDEYGY ILKNHNKMGY SHLLSVRLCV LLGVTVLGFL IIQLLFFCAF EWTSESLEGM SSY EKLVGS LFQVVNSRHT GETIVDLSTL SPAILVLFIL MMYLPPYTLF MPLTEQKTIE KEGGDDDSEN GKKVKKSGLI VSQL SFLTI CIFLISITER QNLQRDPINF NVLNITLEVI SAYGNVGFTT GYSCERRVDI SDGGCKDASY GFAGRWSPMG KFVLI IVMF YGRFKQFTAK SGRAWILYPS SS UniProtKB: Sodium transporter HKT1 |
-分子 #2: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 2 / コピー数: 4 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均露光時間: 8.0 sec. / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 54018 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)