+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Tomato spotted wilt virus L protein contained endoH domain binding to 3'5'vRNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Tomato spotted wilt virus / L protein / VIRAL PROTEIN / VIRAL PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-directed RNA polymerase / viral RNA genome replication / RNA-dependent RNA polymerase activity / DNA-templated transcription 類似検索 - 分子機能 | |||||||||
| 生物種 |  Tomato spotted wilt virus (strain Bulgarian L3) (ウイルス) / synthetic construct (人工物) Tomato spotted wilt virus (strain Bulgarian L3) (ウイルス) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Cao L / Wang X | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of Tomato spotted wilt virus L protein 著者: Cao L / Wang X | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37253.map.gz emd_37253.map.gz | 38.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37253-v30.xml emd-37253-v30.xml emd-37253.xml emd-37253.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_37253.png emd_37253.png | 105.5 KB | ||
| Filedesc metadata |  emd-37253.cif.gz emd-37253.cif.gz | 6.7 KB | ||
| その他 |  emd_37253_half_map_1.map.gz emd_37253_half_map_1.map.gz emd_37253_half_map_2.map.gz emd_37253_half_map_2.map.gz | 37.8 MB 37.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37253 http://ftp.pdbj.org/pub/emdb/structures/EMD-37253 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37253 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37253 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37253_validation.pdf.gz emd_37253_validation.pdf.gz | 902.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37253_full_validation.pdf.gz emd_37253_full_validation.pdf.gz | 901.6 KB | 表示 | |
| XML形式データ |  emd_37253_validation.xml.gz emd_37253_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_37253_validation.cif.gz emd_37253_validation.cif.gz | 13.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37253 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37253 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37253 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37253 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ki7MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37253.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37253.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
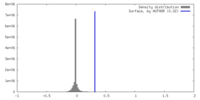
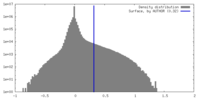
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Structure of Tomato spotted wilt virus L protein contained endoH ...
| 全体 | 名称: Structure of Tomato spotted wilt virus L protein contained endoH domain binding to 3'5'vRNA |
|---|---|
| 要素 |
|
-超分子 #1: Structure of Tomato spotted wilt virus L protein contained endoH ...
| 超分子 | 名称: Structure of Tomato spotted wilt virus L protein contained endoH domain binding to 3'5'vRNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Tomato spotted wilt virus (strain Bulgarian L3) (ウイルス) Tomato spotted wilt virus (strain Bulgarian L3) (ウイルス) |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Tomato spotted wilt virus (strain Bulgarian L3) (ウイルス) Tomato spotted wilt virus (strain Bulgarian L3) (ウイルス)株: Bulgarian L3 |
| 分子量 | 理論値: 240.378375 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNIQKIQKLI ENGTTLLLSI EDCVGSNYDL ALDLHKRNSD EIPEDVIINN NAKNYETMRE LIVKITADGE GLNKGMATVD VKKLSEMVS LFEQKYLETE LARHDIFGEL ISRHLRIKPK QRSEVEIEHA LREYLDELNK KSCINKLSDD EFERINKEYV A TNATPDNY ...文字列: MNIQKIQKLI ENGTTLLLSI EDCVGSNYDL ALDLHKRNSD EIPEDVIINN NAKNYETMRE LIVKITADGE GLNKGMATVD VKKLSEMVS LFEQKYLETE LARHDIFGEL ISRHLRIKPK QRSEVEIEHA LREYLDELNK KSCINKLSDD EFERINKEYV A TNATPDNY VIYKESKNSE LCLIIYDWKI SVDARTETKT MEKYYKNIWK SFKDIKVNGK PFLEDHPVFV SIVILKPIAG MP ITVTSSR VLEKFEDSPS ALHGERIKHA RNAKLLNISH VGQIVGTTPT VVRNYYANTQ KIKSEVRGIL GDDFGSKDVF FSH WTSKYK ERNPTEIAYS EDIERIIDSL VTDEITKEEI IHFLFGNFCF HIETMNDQHI ADKFKGYQSS CINLKIEPKV DLAD LKDHL IQKQQIWESL YGKHLEKIML RIREKKKKEK EIPDITTAFN QNAAEYEEKY PNCFTNDLSE TKTNFSMTWS PSFEK IELS SEVDYNNAII NKFRESFKSS SRVIYNSPYS SINNQTNKAR DITNLVRLCL TELSCDTTKM EKQELEDEID INTGSI KVE RTKKSKEWNK QGSCLTRNKN EFCMKETGRE NKTIYFKGLA VMNIGMSSKK RILKKEEIKE RISKGLEYDT SERQADP ND DYSSIDMSSL THMKKLIRHD NEDSLSWCER IKDSLFVLHN GDIREEGKIT SVYNNYAKNP ECLYIQDSVL KTELETCK K INKLCNDLAI YHYSEDMMQF SKGLMVADRY MTKESFKILT TANTSMMLLA FKGDGMNTGG SGVPYIALHI VDEDMSDQF NICYTKEIYS YFRNGSNYIY IMRPQRLNQV RLLSLFKTPS KVPVCFAQFS KKANEMEKWL KNKDIEKVNV FSMTMTVKQI LINIVFSSV MIGTVTKLSR MGIFDFMRYA GFLPLSDYSN IKEYIRDKFD PDITNVADIY FVNGIKKLLF RMEDLNLSTN A KPVVVDHE NDIIGGITDL NIKCPITGST LLTLEDLYNN VYLAIYMMPK SLHNHVHNLT SLLNVPAEWE LKFRKELGFN IF EDIYPKK AMFDDKDLFS INGALNVKAL SDYYLGNIEN VGLMRSEIEN KEDFLSPCYK ISTLKSSKKC SQSNIISTDE IIE CLQNAK IQDIENWKGN NLAIIKGLIR TYNEEKNRLV EFFEDNCVNS LYLVEKLKEI INSGSITVGK SVTSKFIRNN HPLT VETYL KTKLYYRNNV TVLKSKKVSE ELYDLVKQFH NMMEIDLDSV MNLGKGTEGK KHTFLQMLEF VMSKAKNVTG SVDFL VSVF EKMQRTKTDR EIYLMSMKVK MMLYFIEHTF KHVAQSDPSE AISISGDNKI RALSTLSLDT ITSYNDILNK NSKKSR LAF LSADQSKWSA SDLTYKYVLA IILNPILTTG EASLMIECIL MYVKLKKVCI PTDIFLNLRK AQGTFGQNET AIGLLTK GL TTNTYPVSMN WLQGNLNYLS SVYHSCAMKA YHKTLECYKD CDFQTRWIVH SDDNATSLIA SGEVDKMLTD FSSSSLPE M LFRSIEAHFK SFCITLNPKK SYASSSEVEF ISERIVNGAI IPLYCRHLAN CCTESSHISY FDDLMSLSIH VTMLLRKGC PNEVIPFAYG AVQVQALSIY SMLPGEVNDS IRIFKKLGVS LKSNEIPTNM GGWLTSPIEP LSILGPSSND QIIYYNVIRD FLNKKSLEE VKDSVSSSSY LQMRFRELKG KYEKGTLEEK DKKMIFLINL FEKASVSEDS DVLTIGMKFQ TMLTQIIKLP N FINENALN KMSSYKDFSK LYPNLKKNED LYKSTKNLKI DEDAILEEDE LYEKIASSLE MESVHDIMIK NPETILIAPL ND RDFLLSQ LFMYTSPSKR NQLSNQSTEK LALDRVLRSK ARTFVDISST VKMTYEENME KKILEMLKFD LDSYCSFKTC VNL VIKDVN FSMLIPILDS AYPCESRKRD NYNFRWFQTE KWIPVVEGSP GLVVMHAVYG SNYIENLGLK NIPLTDDSIN VLTS TFGTG LIMEDVKSLV KGKDSFETEA FSNSNECQRL VKACNYMIAA QNRLLAINTC FTRKSFPFYS KFNLGRGFIS NTLAL LSTI YSKEES UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: RNA (5'-R(P*AP*GP*AP*GP*CP*AP*AP*UP*CP*A)-3')
| 分子 | 名称: RNA (5'-R(P*AP*GP*AP*GP*CP*AP*AP*UP*CP*A)-3') / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 3.208012 KDa |
| 配列 | 文字列: AGAGCAAUCA |
-分子 #3: RNA (5'-R(P*GP*CP*AP*AP*UP*CP*AP*GP*G)-3')
| 分子 | 名称: RNA (5'-R(P*GP*CP*AP*AP*UP*CP*AP*GP*G)-3') / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 2.894807 KDa |
| 配列 | 文字列: GCAAUCAGG |
-分子 #4: RNA (5'-R(P*AP*CP*CP*UP*GP*AP*UP*UP*GP*CP*UP*CP*U)-3')
| 分子 | 名称: RNA (5'-R(P*AP*CP*CP*UP*GP*AP*UP*UP*GP*CP*UP*CP*U)-3') タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.055421 KDa |
| 配列 | 文字列: ACCUGAUUGC UCU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 156980 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)