+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a synthetic circadian clock protein KaiC mutant of cyanobacteria Synechococcus elongatus PCC 7942 | |||||||||
 マップデータ マップデータ | Cryo-EM map of anKaiC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | circadian clock protein / Synthetic protein / KaiC / BIOSYNTHETIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of phosphorelay signal transduction system / negative regulation of circadian rhythm / entrainment of circadian clock / protein serine/threonine/tyrosine kinase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / circadian rhythm / non-specific serine/threonine protein kinase / phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity ...regulation of phosphorelay signal transduction system / negative regulation of circadian rhythm / entrainment of circadian clock / protein serine/threonine/tyrosine kinase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / circadian rhythm / non-specific serine/threonine protein kinase / phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / regulation of DNA-templated transcription / magnesium ion binding / ATP hydrolysis activity / DNA binding / ATP binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) | |||||||||
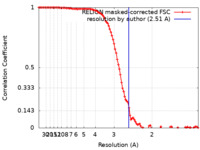
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.51 Å | |||||||||
 データ登録者 データ登録者 | Jia X / Zhang Q / Li S / Guo J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Adaptation of ancient cyanobacterial clock to the day length ~ 0.95 Ga ago 著者: Li S / Wan Y | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36461.map.gz emd_36461.map.gz | 96.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36461-v30.xml emd-36461-v30.xml emd-36461.xml emd-36461.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36461_fsc.xml emd_36461_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36461.png emd_36461.png | 210.6 KB | ||
| Filedesc metadata |  emd-36461.cif.gz emd-36461.cif.gz | 5.5 KB | ||
| その他 |  emd_36461_half_map_1.map.gz emd_36461_half_map_1.map.gz emd_36461_half_map_2.map.gz emd_36461_half_map_2.map.gz | 79.5 MB 79.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36461 http://ftp.pdbj.org/pub/emdb/structures/EMD-36461 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36461 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36461 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36461_validation.pdf.gz emd_36461_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36461_full_validation.pdf.gz emd_36461_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_36461_validation.xml.gz emd_36461_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_36461_validation.cif.gz emd_36461_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36461 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36461 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36461 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36461 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jonMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36461.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36461.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of anKaiC | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.71 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 1
| ファイル | emd_36461_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
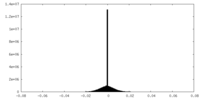
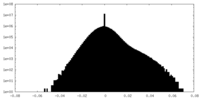
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_36461_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
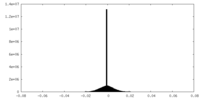
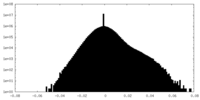
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Synthetic KaiC protein
| 全体 | 名称: Synthetic KaiC protein |
|---|---|
| 要素 |
|
-超分子 #1: Synthetic KaiC protein
| 超分子 | 名称: Synthetic KaiC protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) |
-分子 #1: Circadian clock oscillator protein KaiC
| 分子 | 名称: Circadian clock oscillator protein KaiC / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) |
| 分子量 | 理論値: 58.58909 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSLAEHTSP NNESSHQRLS EVQKMPTMIE GFDDISHGGL PTGRSTLVSG TSGTGKTLFS IQFLYNGITE FDEPGIFVTF EESPQDIIK NARSFGWNLQ KLVDQGKLFI LDASPDPDGQ EVAGGFDLSA LIERINYAIQ KYKARRVSID SVTAVFQQYD A ASVVRREI ...文字列: MTSLAEHTSP NNESSHQRLS EVQKMPTMIE GFDDISHGGL PTGRSTLVSG TSGTGKTLFS IQFLYNGITE FDEPGIFVTF EESPQDIIK NARSFGWNLQ KLVDQGKLFI LDASPDPDGQ EVAGGFDLSA LIERINYAIQ KYKARRVSID SVTAVFQQYD A ASVVRREI FRLVARLKQI GVTTVMTTER IDEYGPIARY GVEEFVSDNV VILRNVLEGE RRRRTVEILK LRGTTHMKGE YP FTINDHG INIFPLGAMR LTQRSSNVRV SSGVPRLDEM CGGGFFKDSI ILATGATGTG KTLLVSKFIE NACQNKERAI LFA YEESRA QLLRNASSWG IDFEEMERQG LLKIICAYPE SAGLEDHLQI IKSEIGDFKP SRIAIDSLSA LARGVSNNAF RQFV IGVTG YAKQEEITGF FTNTSDQFMG SHSITDSHIS TITDTILLLQ YVEIRGEMSR AINVFKMRGS WHDKGIREYV ITDKG PEIK DSFRNFERII SGSPTRITVD EKSENLSRIA RGVQEKEPES UniProtKB: Circadian clock oscillator protein KaiC |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 12 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X