+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Flagellar fibrils from Bacillus amyloliquefaciens | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Flagella / motor / fibril / PROTEIN FIBRIL | |||||||||
| 生物種 |  | |||||||||
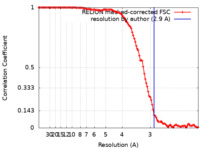
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Cao Q / Cheng Y | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Serine peptidase Vpr forms enzymatically active fibrils outside Bacillus bacteria revealed by cryo-EM. 著者: Yijia Cheng / Jianting Han / Meinai Song / Shuqin Zhang / Qin Cao /  要旨: Bacteria develop a variety of extracellular fibrous structures crucial for their survival, such as flagella and pili. In this study, we use cryo-EM to identify protein fibrils surrounding lab- ...Bacteria develop a variety of extracellular fibrous structures crucial for their survival, such as flagella and pili. In this study, we use cryo-EM to identify protein fibrils surrounding lab-cultured Bacillus amyloiquefaciens and discover an unreported fibril species in addition to the flagellar fibrils. These previously unknown fibrils are composed of Vpr, an extracellular serine peptidase. We find that Vpr assembles into fibrils in an enzymatically active form, potentially representing a strategy of enriching Vpr activities around bacterial cells. Vpr fibrils are also observed under other culture conditions and around other Bacillus bacteria, such as Bacillus subtilis, which may suggest a general mechanism across all Bacillus bacterial groups. Taken together, our study reveals fibrils outside the bacterial cell and sheds light on the physiological role of these extracellular fibrils. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36427.map.gz emd_36427.map.gz | 110.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36427-v30.xml emd-36427-v30.xml emd-36427.xml emd-36427.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36427_fsc.xml emd_36427_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36427.png emd_36427.png | 85.6 KB | ||
| Filedesc metadata |  emd-36427.cif.gz emd-36427.cif.gz | 4.9 KB | ||
| その他 |  emd_36427_half_map_1.map.gz emd_36427_half_map_1.map.gz emd_36427_half_map_2.map.gz emd_36427_half_map_2.map.gz | 97.2 MB 97.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36427 http://ftp.pdbj.org/pub/emdb/structures/EMD-36427 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36427 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36427 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36427_validation.pdf.gz emd_36427_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36427_full_validation.pdf.gz emd_36427_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_36427_validation.xml.gz emd_36427_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_36427_validation.cif.gz emd_36427_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36427 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36427 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36427 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36427 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36427.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36427.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_36427_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
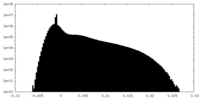
| 投影像・断面図 |
| ||||||||||||
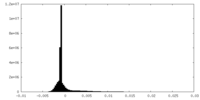
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36427_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
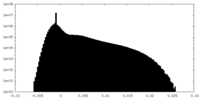
| 投影像・断面図 |
| ||||||||||||
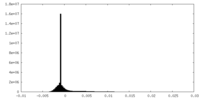
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacillus amyloliquefaciens
| 全体 | 名称:  |
|---|---|
| 要素 |
|
-超分子 #1: Bacillus amyloliquefaciens
| 超分子 | 名称: Bacillus amyloliquefaciens / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Flagella
| 分子 | 名称: Flagella / タイプ: protein_or_peptide / ID: 1 / コピー数: 33 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 35.12884 KDa |
| 配列 | 文字列: MRINHNIAAL NTSRQLNAGS NAASKNMEKL SSGLRINRAG DDAAGLAISE KMRSQIRGLD MASKNAQDGI SLIQTSEGAL NETHSILQR MSELATQAAN DTNTDSDRSE LQKEMDQLAS EVTRISTDTE FNTKKLLDGT AQNLTFQIGA NEGQTMSLSI N KMDSESLK ...文字列: MRINHNIAAL NTSRQLNAGS NAASKNMEKL SSGLRINRAG DDAAGLAISE KMRSQIRGLD MASKNAQDGI SLIQTSEGAL NETHSILQR MSELATQAAN DTNTDSDRSE LQKEMDQLAS EVTRISTDTE FNTKKLLDGT AQNLTFQIGA NEGQTMSLSI N KMDSESLK VGTTYTANDD GSKLVTADGK EATLVTDGSK GPNGYYDDAD KLVYQADSAL AKDTKVTKGI DISSSAKAAS SA LTTIKTA IDTVSSERAK LGAVQNRLEH TINNLGTSSE NLTSAESRIR DVDMASEMME YTKNNILTQA SQAMLAQANQ QPQ QVLQLL KG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 4.6000000000000005 µm 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X