+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the prokaryotic SPARSA system complex | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | SPARSA antiviral system / NADase / Argonaute proteins / ANTIVIRAL PROTEIN | ||||||||||||
| 機能・相同性 | SIR2-like domain / SIR2-like domain / DHS-like NAD/FAD-binding domain superfamily / Ribonuclease H superfamily / nucleic acid binding / Ribonuclease H-like superfamily / Piwi domain protein / Sir2 superfamily protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Geobacter sulfurreducens PCA (バクテリア) / Geobacter sulfurreducens PCA (バクテリア) /  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) | ||||||||||||
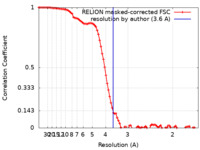
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||
 データ登録者 データ登録者 | Xu X / Zhen X / Long F | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis of antiphage immunity generated by a prokaryotic Argonaute-associated SPARSA system. 著者: Xiangkai Zhen / Xiaolong Xu / Le Ye / Song Xie / Zhijie Huang / Sheng Yang / Yanhui Wang / Jinyu Li / Feng Long / Songying Ouyang /  要旨: Argonaute (Ago) proteins are ubiquitous across all kingdoms of life. Eukaryotic Agos (eAgos) use small RNAs to recognize transcripts for RNA silencing in eukaryotes. In contrast, the functions of ...Argonaute (Ago) proteins are ubiquitous across all kingdoms of life. Eukaryotic Agos (eAgos) use small RNAs to recognize transcripts for RNA silencing in eukaryotes. In contrast, the functions of prokaryotic counterparts (pAgo) are less well known. Recently, short pAgos in conjunction with the associated TIR or Sir2 (SPARTA or SPARSA) were found to serve as antiviral systems to combat phage infections. Herein, we present the cryo-EM structures of nicotinamide adenine dinucleotide (NAD)-bound SPARSA with and without nucleic acids at resolutions of 3.1 Å and 3.6 Å, respectively. Our results reveal that the APAZ (Analogue of PAZ) domain and the short pAgo form a featured architecture similar to the long pAgo to accommodate nucleic acids. We further identified the key residues for NAD binding and elucidated the structural basis for guide RNA and target DNA recognition. Using structural comparisons, molecular dynamics simulations, and biochemical experiments, we proposed a putative mechanism for NAD hydrolysis in which an H186 loop mediates nucleophilic attack by catalytic water molecules. Overall, our study provides mechanistic insight into the antiphage role of the SPARSA system. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36384.map.gz emd_36384.map.gz | 6.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36384-v30.xml emd-36384-v30.xml emd-36384.xml emd-36384.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36384_fsc.xml emd_36384_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36384.png emd_36384.png | 69.5 KB | ||
| マスクデータ |  emd_36384_msk_1.map emd_36384_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-36384.cif.gz emd-36384.cif.gz | 6.5 KB | ||
| その他 |  emd_36384_half_map_1.map.gz emd_36384_half_map_1.map.gz emd_36384_half_map_2.map.gz emd_36384_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36384 http://ftp.pdbj.org/pub/emdb/structures/EMD-36384 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36384 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36384 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36384_validation.pdf.gz emd_36384_validation.pdf.gz | 930.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36384_full_validation.pdf.gz emd_36384_full_validation.pdf.gz | 929.7 KB | 表示 | |
| XML形式データ |  emd_36384_validation.xml.gz emd_36384_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_36384_validation.cif.gz emd_36384_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36384 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36384 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36384 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36384 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jkzMC  8jl0C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36384.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36384.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_36384_msk_1.map emd_36384_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36384_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36384_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sir2/Ago
| 全体 | 名称: Sir2/Ago |
|---|---|
| 要素 |
|
-超分子 #1: Sir2/Ago
| 超分子 | 名称: Sir2/Ago / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens PCA (バクテリア) Geobacter sulfurreducens PCA (バクテリア) |
-超分子 #2: Sir2
| 超分子 | 名称: Sir2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens PCA (バクテリア) Geobacter sulfurreducens PCA (バクテリア) |
-超分子 #3: Ago
| 超分子 | 名称: Ago / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Sir2 superfamily protein
| 分子 | 名称: Sir2 superfamily protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 66.2685 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDVLTDNEFY QHYLQNSQHM MWFLGAGTSR SAGLPTASDI IWDLKHRYYC LHENQDYQKH DINNHAIKSK IQSYMDSKGF PLQWSPEEY SFYFELVFRD DYEAQRKYLL EALASRKVSL NIGHRVLAAL LEMNQTKVVF TTNFDDVIET AFSDISGKHL S VYHLEGSY ...文字列: MDVLTDNEFY QHYLQNSQHM MWFLGAGTSR SAGLPTASDI IWDLKHRYYC LHENQDYQKH DINNHAIKSK IQSYMDSKGF PLQWSPEEY SFYFELVFRD DYEAQRKYLL EALASRKVSL NIGHRVLAAL LEMNQTKVVF TTNFDDVIET AFSDISGKHL S VYHLEGSY AALSALNTEA FPIYAKIHGD FRYQKIKNLT PDLQTNDREI HKCFLAAAIR FGLVVSGYSG RDENVMTMLR AA IDQNNAF PHGLYWTVPS ISKSEPAVQD LITYAQGKGV RAYLVETGTF DEMLSKIWRQ VKDKPAAIDA KVRTARVCPV SIP LPGPGK SFPALRTNAL PVVTQSIRCG VVTLASPITF SELKERISQK SPKALLTYTE KVLFLGGEPE IRKIFSNDEI NSIG QYYID EIAQSVAAST FLKSFVEEAI LTALLREKPI LHRVRHRTHY AVIPNASAKD DRFLDLRKAV GFKGDLGYIT GNVTN AKEL SWAEAVSIRL EERGGKLWIM LKPEIWIKPL DRREEATDFI RSRRRYRFNQ CSYQILDAWI KILFGSIGGG GTVNIS CFP DAEFKAEFEI GTRTAFSLGV G UniProtKB: Sir2 superfamily protein |
-分子 #2: Piwi domain protein
| 分子 | 名称: Piwi domain protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 53.325566 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADNLSQLAA HSTIPEPLLL FKDNRTDTHP LRGLSQYGPY SACFNLPGQV RLAYLAPTEH MRKLDAIVRE LQNPATPKEA TNYYVEYGG FEKVFKVPLV MPQEHLRCLA LDECHGVAAN GNGLALADKI VQSMSGLFRQ KHAFDVLLVY LPASWKKCFE Y DGFDLHDR ...文字列: MADNLSQLAA HSTIPEPLLL FKDNRTDTHP LRGLSQYGPY SACFNLPGQV RLAYLAPTEH MRKLDAIVRE LQNPATPKEA TNYYVEYGG FEKVFKVPLV MPQEHLRCLA LDECHGVAAN GNGLALADKI VQSMSGLFRQ KHAFDVLLVY LPASWKKCFE Y DGFDLHDR IKAKVAPLNL PIQIINDTAL TRQCRANVMW GVSVALYAKA GGIPWKLADW DKDEAYIGLS YAIKKNAEGQ EY TTCCSQV FDPDGTGFEF VAYDTREFIT DRKGNPYLSY QEMQSVLSKS LHLYQSSHNG RMPRKIFIHK TTHFTEDEIQ GAF DSFSSS TEIELVQIIQ STNWYGLKVD GKKGDKPVAP ASYPVDRGLY QPLTESECLL WTQGSVMGVN QQNPGQPVFK EAAL TPLPN PIMLRRFSGN GGWHATCSSI LALTKVDWNN NTLYKKLPVT LVYSQVFADV VKQTPEIVNE IYDYRFFM UniProtKB: Piwi domain protein |
-分子 #3: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 50 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| ソフトウェア | 名称: EPU (ver. FEI) |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8jkz: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)