+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DltB tetramer in complex with inhibitor m-AMSA | |||||||||
 マップデータ マップデータ | This map is reconstructed from cryo-EM images collected from K3 camera of TITAN KRIOSg3i and is analysised mainly by the cryoSPARC and Relion. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | channel / anti-virulence / MBOAT / DltB / MEMBRANE PROTEIN / MEMBRANE PROTEIN-INHIBITOR complex | |||||||||
| 機能・相同性 | D-alanyl transfer protein DltB / Alginate O-acetyltransferase AlgI/D-alanyl transfer protein DltB / lipoteichoic acid biosynthetic process / Membrane bound O-acyl transferase, MBOAT / MBOAT, membrane-bound O-acyltransferase family / acyltransferase activity / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / plasma membrane / Teichoic acid D-alanyltransferase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) /  Streptococcus thermophilus LMG 18311 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus LMG 18311 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.23 Å | |||||||||
 データ登録者 データ登録者 | Zhang P / Liu Z | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural insights into the transporting and catalyzing mechanism of DltB in LTA D-alanylation. 著者: Pingfeng Zhang / Zheng Liu /  要旨: DltB, a model member of the Membrane-Bound O-AcylTransferase (MBOAT) superfamily, plays a crucial role in D-alanylation of the lipoteichoic acid (LTA), a significant component of the cell wall of ...DltB, a model member of the Membrane-Bound O-AcylTransferase (MBOAT) superfamily, plays a crucial role in D-alanylation of the lipoteichoic acid (LTA), a significant component of the cell wall of gram-positive bacteria. This process stabilizes the cell wall structure, influences bacterial virulence, and modulates the host immune response. Despite its significance, the role of DltB is not well understood. Through biochemical analysis and cryo-EM imaging, we discover that Streptococcus thermophilus DltB forms a homo-tetramer on the cell membrane. We further visualize DltB in an apo form, in complex with DltC, and in complex with its inhibitor amsacrine (m-AMSA). Each tetramer features a central hole. The C-tunnel of each protomer faces the intratetramer interface and provides access to the periphery membrane. Each protomer binds a DltC without changing the tetrameric organization. A phosphatidylglycerol (PG) molecule in the substrate-binding site may serve as an LTA carrier. The inhibitor m-AMSA bound to the L-tunnel of each protomer blocks the active site. The tetrameric organization of DltB provides a scaffold for catalyzing D-alanyl transfer and regulating the channel opening and closing. Our findings unveil DltB's dual function in the D-alanylation pathway, and provide insight for targeting DltB as a anti-virulence antibiotic. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36192.map.gz emd_36192.map.gz | 117.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36192-v30.xml emd-36192-v30.xml emd-36192.xml emd-36192.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36192.png emd_36192.png | 209.5 KB | ||
| Filedesc metadata |  emd-36192.cif.gz emd-36192.cif.gz | 6.3 KB | ||
| その他 |  emd_36192_half_map_1.map.gz emd_36192_half_map_1.map.gz emd_36192_half_map_2.map.gz emd_36192_half_map_2.map.gz | 115.8 MB 115.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36192 http://ftp.pdbj.org/pub/emdb/structures/EMD-36192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36192 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36192 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36192_validation.pdf.gz emd_36192_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36192_full_validation.pdf.gz emd_36192_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_36192_validation.xml.gz emd_36192_validation.xml.gz | 13.8 KB | 表示 | |
| CIF形式データ |  emd_36192_validation.cif.gz emd_36192_validation.cif.gz | 16.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36192 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36192 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36192 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36192.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36192.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This map is reconstructed from cryo-EM images collected from K3 camera of TITAN KRIOSg3i and is analysised mainly by the cryoSPARC and Relion. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36192_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36192_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DltB tetramer in complex with inhibitor m-AMSA.
| 全体 | 名称: DltB tetramer in complex with inhibitor m-AMSA. |
|---|---|
| 要素 |
|
-超分子 #1: DltB tetramer in complex with inhibitor m-AMSA.
| 超分子 | 名称: DltB tetramer in complex with inhibitor m-AMSA. / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)株: LMG 18311 |
| 分子量 | 理論値: 48 kDa/nm |
-分子 #1: Teichoic acid D-alanyltransferase
| 分子 | 名称: Teichoic acid D-alanyltransferase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO EC番号: 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus LMG 18311 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus LMG 18311 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)株: LMG 18311 |
| 分子量 | 理論値: 51.780027 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH NYDIPTTENL YFQGSMIDFL KQLPHLEPYG NPFYFIYLGI ALLPIFIGLF FKKRFAIYEC LVSITFIVLA LTGTHASQI LALLFYIVWQ IIWVYSYKRY RSQRDNKWVF YLHSFLVVLP LILVKVEPTI NGTQSLLNFL GISYLTFRAV G MIIEMRDG ...文字列: MGSSHHHHHH NYDIPTTENL YFQGSMIDFL KQLPHLEPYG NPFYFIYLGI ALLPIFIGLF FKKRFAIYEC LVSITFIVLA LTGTHASQI LALLFYIVWQ IIWVYSYKRY RSQRDNKWVF YLHSFLVVLP LILVKVEPTI NGTQSLLNFL GISYLTFRAV G MIIEMRDG VLKEFTLGEF LRFMLFMPTF TSGPIDRFKR FNEDYQSIPN RDELLNMLEQ AVKYIMLGFL YKFVLAQIFG SM LLPPLKA QALSQGGIFN LPTLGVMYVY GFDLFFDFAG YSMFALAVSN LMGIKSPINF DKPFISRDMK EFWNRWHMSL SFW FRDFVF MRLVIVLMRN KVFKNRNTTS NVAYIINMMV MGFWHGITWY YIAYGIFHGI GLVINDAWLR KKKTINKDRK KAGL KPLPE NKWTKALGIF ITFNTVMLSF LIFSGFLNDL WFTKK UniProtKB: Teichoic acid D-alanyltransferase |
-分子 #2: N-[4-(acridin-9-ylamino)-3-methoxyphenyl]methanesulfonamide
| 分子 | 名称: N-[4-(acridin-9-ylamino)-3-methoxyphenyl]methanesulfonamide タイプ: ligand / ID: 2 / コピー数: 4 / 式: ASW |
|---|---|
| 分子量 | 理論値: 393.459 Da |
| Chemical component information | 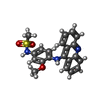 ChemComp-ASW: |
-分子 #3: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
| 分子 | 名称: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-1-[(PALMITOYLOXY)METHYL]ETHYL STEARATE タイプ: ligand / ID: 3 / コピー数: 8 / 式: PGT |
|---|---|
| 分子量 | 理論値: 751.023 Da |
| Chemical component information |  ChemComp-PGT: |
-分子 #4: DODECYL-BETA-D-MALTOSIDE
| 分子 | 名称: DODECYL-BETA-D-MALTOSIDE / タイプ: ligand / ID: 4 / コピー数: 40 / 式: LMT |
|---|---|
| 分子量 | 理論値: 510.615 Da |
| Chemical component information |  ChemComp-LMT: |
-分子 #5: DIACYL GLYCEROL
| 分子 | 名称: DIACYL GLYCEROL / タイプ: ligand / ID: 5 / コピー数: 2 / 式: DGA |
|---|---|
| 分子量 | 理論値: 625.018 Da |
| Chemical component information |  ChemComp-DGA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 25.0 mM / 構成要素 - 名称: Hepes / 詳細: 20 mM Hopes-Na, pH7.5, 0.03% DDM |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: After incubation on the grids at 277K under 100% humidity for 10 s, the grids were bloted for 3.0 s and then plunged frozen into liquid ethane cooled by liquid nitrogen using a Vitrobot.. |
| 詳細 | The DltB protein was purified in DDM, the tetramer fractions from gel filtration column were concentrated to about 10 mg/ml. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: 詳細: Crystal structure of a membrane-bound O-acyltransferase. |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.23 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 294021 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-8jem: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X