+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of type I-B Cascade bound to a PAM-containing dsDNA target at 3.6 angstrom resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Type I-B / CRISPR-Cas / Cascade / RNA BINDING PROTEIN/RNA/DNA / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Xiao Y / Lu M / Yu C / Zhang Y | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structure and genome editing of type I-B CRISPR-Cas. 著者: Meiling Lu / Chenlin Yu / Yuwen Zhang / Wenjun Ju / Zhi Ye / Chenyang Hua / Jinze Mao / Chunyi Hu / Zhenhuang Yang / Yibei Xiao /   要旨: Type I CRISPR-Cas systems employ multi-subunit effector Cascade and helicase-nuclease Cas3 to target and degrade foreign nucleic acids, representing the most abundant RNA-guided adaptive immune ...Type I CRISPR-Cas systems employ multi-subunit effector Cascade and helicase-nuclease Cas3 to target and degrade foreign nucleic acids, representing the most abundant RNA-guided adaptive immune systems in prokaryotes. Their ability to cause long fragment deletions have led to increasing interests in eukaryotic genome editing. While the Cascade structures of all other six type I systems have been determined, the structure of the most evolutionarily conserved type I-B Cascade is still missing. Here, we present two cryo-EM structures of the Synechocystis sp. PCC 6714 (Syn) type I-B Cascade, revealing the molecular mechanisms that underlie RNA-directed Cascade assembly, target DNA recognition, and local conformational changes of the effector complex upon R-loop formation. Remarkably, a loop of Cas5 directly intercalated into the major groove of the PAM and facilitated PAM recognition. We further characterized the genome editing profiles of this I-B Cascade-Cas3 in human CD3 T cells using mRNA-mediated delivery, which led to unidirectional 4.5 kb deletion in TRAC locus and achieved an editing efficiency up to 41.2%. Our study provides the structural basis for understanding target DNA recognition by type I-B Cascade and lays foundation for harnessing this system for long range genome editing in human T cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35629.map.gz emd_35629.map.gz | 52.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35629-v30.xml emd-35629-v30.xml emd-35629.xml emd-35629.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35629.png emd_35629.png | 52.3 KB | ||
| Filedesc metadata |  emd-35629.cif.gz emd-35629.cif.gz | 6.7 KB | ||
| その他 |  emd_35629_half_map_1.map.gz emd_35629_half_map_1.map.gz emd_35629_half_map_2.map.gz emd_35629_half_map_2.map.gz | 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35629 http://ftp.pdbj.org/pub/emdb/structures/EMD-35629 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35629 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35629 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35629_validation.pdf.gz emd_35629_validation.pdf.gz | 994.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35629_full_validation.pdf.gz emd_35629_full_validation.pdf.gz | 993.9 KB | 表示 | |
| XML形式データ |  emd_35629_validation.xml.gz emd_35629_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_35629_validation.cif.gz emd_35629_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35629 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35629 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35629 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35629 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ip0MC  8h67C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35629.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35629.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||
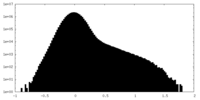
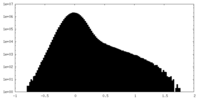
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35629_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
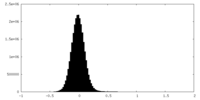
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35629_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
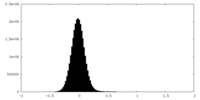
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of Synechocystis sp. PCC6714 Cascade bound to a...
| 全体 | 名称: Cryo-EM structure of Synechocystis sp. PCC6714 Cascade bound to a PAM-containing dsDNA target presented as full R-loop formed state at 3.6 angstrom resolution |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of Synechocystis sp. PCC6714 Cascade bound to a...
| 超分子 | 名称: Cryo-EM structure of Synechocystis sp. PCC6714 Cascade bound to a PAM-containing dsDNA target presented as full R-loop formed state at 3.6 angstrom resolution タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#8 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Type I-MYXAN CRISPR-associated protein Cmx8
| 分子 | 名称: Type I-MYXAN CRISPR-associated protein Cmx8 / タイプ: protein_or_peptide / ID: 1 / 詳細: GenBank ID WP_071880939 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.703344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPKTQAEILT LDFNLAELPS AQHRAGLAGL ILMIRELKKW PWFKIRQKEK DVLLSIENLD QYGASIQLNL EGLIALFDLA YLSFTEERK SKSKIKDFKR VDEIEIEENG KNKIQKYYFY DVITPQGGFL AGWDKSDGQI WLRIWRDMFW SIIKGVPATR N PFNNRCGL ...文字列: MPKTQAEILT LDFNLAELPS AQHRAGLAGL ILMIRELKKW PWFKIRQKEK DVLLSIENLD QYGASIQLNL EGLIALFDLA YLSFTEERK SKSKIKDFKR VDEIEIEENG KNKIQKYYFY DVITPQGGFL AGWDKSDGQI WLRIWRDMFW SIIKGVPATR N PFNNRCGL NLNAGDSFSK DVESVWKSLQ NAEKTTGQSG AFYLGAMAVN AENVSTDDLI KWQFLLHFWA FVAQVYCPYI LD KDGKRNF NGYVIVIPDI ANLEDFCDIL PDVLSNRNSK AFGFRPQESV IDVPEQGALE LLNLIKQRIA KKAGSGLLSD LIV GVEVIH AEKQGNSIKL HSVSYLQPNE ESVDDYNAIK NSYYCPWFRR QLLLNLVNPK FDLASQSWLK RHPWYGFGDL LSRI PQRWL KENNSYFSHD ARQLFTQKGD FDMTVATTKT REYAEIVYKI AQGFVLSKLS SKHDLQWSKC KGNPKLEREY NDKKE KVVN EAFLAIRSRT EKQAFIDYFV STLYPHVRQD EFVDFAQKLF QDTDEIRSLT LLALSSQYPI KRQGETE UniProtKB: Type I-MYXAN CRISPR-associated protein Cmx8 |
-分子 #2: Type I-MYXAN CRISPR-associated protein Cas5/Cmx5/DevS
| 分子 | 名称: Type I-MYXAN CRISPR-associated protein Cas5/Cmx5/DevS タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.592424 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAQLALALDT VTRYLRLKAP FAAFRPFQSG SFRSTTPVPS FSAVYGLLLN LAGIEQRQEV EGKVTLIKPK AELPKLAIAI GQVKPSSTS LINQQLHNYP VGNSGKEFAS RTFGSKYWIA PVRREVLVNL DLIIGLQSPV EFWQKLDQGL KGETVINRYG L PFAGDNNF ...文字列: MAQLALALDT VTRYLRLKAP FAAFRPFQSG SFRSTTPVPS FSAVYGLLLN LAGIEQRQEV EGKVTLIKPK AELPKLAIAI GQVKPSSTS LINQQLHNYP VGNSGKEFAS RTFGSKYWIA PVRREVLVNL DLIIGLQSPV EFWQKLDQGL KGETVINRYG L PFAGDNNF LFDEIYPIEK PDLASWYCPL EPDTRPNQGA CRLTLWIDRE NNTQTTIKVF SPSDFRLEPP AKAWQQLPG UniProtKB: Uncharacterized protein |
-分子 #3: Fruiting body developmental protein R-like protein
| 分子 | 名称: Fruiting body developmental protein R-like protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 33.789074 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSNLNLFATI LTYPAPASNY RGESEENRSV IQKILKDGQK YAIISPESMR NALREMLIEL GQPNNRTRLH SEDQLAVEFK EYPNPDKFA DDFLFGYMVA QTNDAKEMKK LNRPAKRDSI FRCNMAVAVN PYKYDTVFYQ SPLNAGDSAW KNSTSSALLH R EVTHTAFQ ...文字列: MSNLNLFATI LTYPAPASNY RGESEENRSV IQKILKDGQK YAIISPESMR NALREMLIEL GQPNNRTRLH SEDQLAVEFK EYPNPDKFA DDFLFGYMVA QTNDAKEMKK LNRPAKRDSI FRCNMAVAVN PYKYDTVFYQ SPLNAGDSAW KNSTSSALLH R EVTHTAFQ YPFALAGKDC AAKPEWVKAL LQAIAELNGV AGGHARAYYE FAPRSVVARL TPKLVAGYQT YGFDAEGNWL EL SRLTATD SDNLDLPANE FWLGGELVRK MDQEQKAQLE AMGAHLYANP EKLFADLADS FLGV UniProtKB: Fruiting body developmental protein R-like protein |
-分子 #8: CRISPR associated protein Cas11b
| 分子 | 名称: CRISPR associated protein Cas11b / タイプ: protein_or_peptide / ID: 8 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.5385 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTVATTKTRE YAEIVYKIAQ GFVLSKLSSK HDLQWSKCKG NPKLEREYND KKEKVVNEAF LAIRSRTEKQ AFIDYFVSTL YPHVRQDEF VDFAQKLFQD TDEIRSLTLL ALSSQYPIKR QGETE UniProtKB: Type I-MYXAN CRISPR-associated protein Cmx8 |
-分子 #4: DNA (5'-D(P*TP*CP*CP*AP*TP*GP*TP*TP*TP*AP*TP*CP*AP*CP*C)-3')
| 分子 | 名称: DNA (5'-D(P*TP*CP*CP*AP*TP*GP*TP*TP*TP*AP*TP*CP*AP*CP*C)-3') タイプ: dna / ID: 4 / 詳細: PAM proximal non target DNA / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.494934 KDa |
| 配列 | 文字列: (DT)(DC)(DC)(DA)(DT)(DG)(DT)(DT)(DT)(DA) (DT)(DC)(DA)(DC)(DC) |
-分子 #6: DNA (41-MER)
| 分子 | 名称: DNA (41-MER) / タイプ: dna / ID: 6 / 詳細: Target DNA / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.725236 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DC)(DA)(DA)(DA)(DA)(DT)(DA) (DT)(DC)(DC)(DA)(DG)(DA)(DT)(DT)(DG)(DG) (DG)(DG)(DA)(DC)(DA)(DC)(DG)(DG)(DT) (DG)(DA)(DT)(DA)(DA)(DA)(DC)(DA)(DT)(DG) (DG) (DA) |
-分子 #7: DNA (5'-D(P*AP*AP*AP*AP*AP*AP*AP*AP*AP*A)-3')
| 分子 | 名称: DNA (5'-D(P*AP*AP*AP*AP*AP*AP*AP*AP*AP*A)-3') / タイプ: dna / ID: 7 / 詳細: PAM distal non target DNA / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 3.08711 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) |
-分子 #5: RNA (44-MER)
| 分子 | 名称: RNA (44-MER) / タイプ: rna / ID: 5 / 詳細: CRISPR RNA / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.975212 KDa |
| 配列 | 文字列: AGAGCACUUU UAUCACCGUG UCCCCAAUCU GGAUAUUUUG UGUG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 92454 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: OTHER |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)