+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Rpd3S core complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HDAC / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Snt2C complex / negative regulation of silent mating-type cassette heterochromatin formation / negative regulation of reciprocal meiotic recombination / Rpd3L complex / protein localization to nucleolar rDNA repeats / negative regulation of rDNA heterochromatin formation / Rpd3L-Expanded complex / Rpd3S complex / rDNA chromatin condensation / nucleophagy ...Snt2C complex / negative regulation of silent mating-type cassette heterochromatin formation / negative regulation of reciprocal meiotic recombination / Rpd3L complex / protein localization to nucleolar rDNA repeats / negative regulation of rDNA heterochromatin formation / Rpd3L-Expanded complex / Rpd3S complex / rDNA chromatin condensation / nucleophagy / HDACs deacetylate histones / histone deacetylase / SUMOylation of chromatin organization proteins / regulation of DNA-templated DNA replication initiation / cellular response to nitrogen starvation / negative regulation of transcription by RNA polymerase I / histone deacetylase activity / NuA4 histone acetyltransferase complex / Sin3-type complex / Estrogen-dependent gene expression / positive regulation of macroautophagy / histone deacetylase complex / nuclear periphery / meiotic cell cycle / transcription elongation by RNA polymerase II / heterochromatin formation / double-strand break repair via nonhomologous end joining / G1/S transition of mitotic cell cycle / structural constituent of chromatin / transcription corepressor activity / G2/M transition of mitotic cell cycle / nucleosome / nucleosome assembly / cellular response to heat / response to oxidative stress / transcription coactivator activity / chromatin remodeling / protein heterodimerization activity / cell division / DNA repair / regulation of DNA-templated transcription / chromatin / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA binding / identical protein binding / nucleus / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.37 Å | |||||||||
 データ登録者 データ登録者 | Zhang Y / Gang C | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Res / 年: 2023 ジャーナル: Cell Res / 年: 2023タイトル: Structural basis for nucleosome binding and catalysis by the yeast Rpd3S/HDAC holoenzyme. 著者: Yueyue Zhang / Mengxue Xu / Po Wang / Jiahui Zhou / Guangxian Wang / Shuailong Han / Gang Cai / Xuejuan Wang /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35450.map.gz emd_35450.map.gz | 136.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35450-v30.xml emd-35450-v30.xml emd-35450.xml emd-35450.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35450.png emd_35450.png | 63.8 KB | ||
| Filedesc metadata |  emd-35450.cif.gz emd-35450.cif.gz | 7.7 KB | ||
| その他 |  emd_35450_half_map_1.map.gz emd_35450_half_map_1.map.gz emd_35450_half_map_2.map.gz emd_35450_half_map_2.map.gz | 134.2 MB 134.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35450 http://ftp.pdbj.org/pub/emdb/structures/EMD-35450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35450 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35450_validation.pdf.gz emd_35450_validation.pdf.gz | 975.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35450_full_validation.pdf.gz emd_35450_full_validation.pdf.gz | 975.1 KB | 表示 | |
| XML形式データ |  emd_35450_validation.xml.gz emd_35450_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_35450_validation.cif.gz emd_35450_validation.cif.gz | 17.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35450 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35450 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ihnMC  8ihmC  8ihtC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35450.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35450.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35450_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
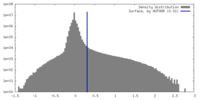
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35450_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Eaf3 CHD domain bound to the nucleosome
| 全体 | 名称: Eaf3 CHD domain bound to the nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: Eaf3 CHD domain bound to the nucleosome
| 超分子 | 名称: Eaf3 CHD domain bound to the nucleosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種: |
-分子 #1: Histone H3
| 分子 | 名称: Histone H3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 2.563012 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKA UniProtKB: Histone H3 |
-分子 #2: Transcriptional regulatory protein SIN3
| 分子 | 名称: Transcriptional regulatory protein SIN3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 175.047266 KDa |
| 配列 | 文字列: MSQVWHNSNS QSNDVATSND ATGSNERNEK EPSLQGNKPG FVQQQQRITL PSLSALSTKE EDRRDSNGQQ ALTSHAAHIL GYPPPHSNA MPSIATDSAL KQPHEYHPRP KSSSSSPSIN ASLMNAGPAP LPTVGAASFS LSRFDNPLPI KAPVHTEEPK S YNGLQEEE ...文字列: MSQVWHNSNS QSNDVATSND ATGSNERNEK EPSLQGNKPG FVQQQQRITL PSLSALSTKE EDRRDSNGQQ ALTSHAAHIL GYPPPHSNA MPSIATDSAL KQPHEYHPRP KSSSSSPSIN ASLMNAGPAP LPTVGAASFS LSRFDNPLPI KAPVHTEEPK S YNGLQEEE KATQRPQDCK EVPAGVQPAD APDPSSNHAD ANDDNNNNEN SHDEDADYRP LNVKDALSYL EQVKFQFSSR PD IYNLFLD IMKDFKSQAI DTPGVIERVS TLFRGYPILI QGFNTFLPQG YRIECSSNPD DPIRVTTPMG TTTVNNNISP SGR GTTDAQ ELGSFPESDG NGVQQPSNVP MVPSSVYQSE QNQDQQQSLP LLATSSGLPS IQQPEMPAHR QIPQSQSLVP QEDA KKNVD VEFSQAISYV NKIKTRFADQ PDIYKHFLEI LQTYQREQKP INEVYAQVTH LFQNAPDLLE DFKKFLPDSS ASANQ QVQH AQQHAQQQHE AQMHAQAQAQ AQAQAQVEQQ KQQQQFLYPA SGYYGHPSNR GIPQQNLPPI GSFSPPTNGS TVHEAY QDQ QHMQPPHFMP LPSIVQHGPN MVHQGIANEN PPLSDLRTSL TEQYAPSSIQ HQQQHPQSIS PIANTQYGDI PVRPEID LD PSIVPVVPEP TEPIENNISL NEEVTFFEKA KRYIGNKHLY TEFLKILNLY SQDILDLDDL VEKVDFYLGS NKELFTWF K NFVGYQEKTK CIENIVHEKH RLDLDLCEAF GPSYKRLPKS DTFMPCSGRD DMCWEVLNDE WVGHPVWASE DSGFIAHRK NQYEETLFKI EEERHEYDFY IESNLRTIQC LETIVNKIEN MTENEKANFK LPPGLGHTSM TIYKKVIRKV YDKERGFEII DALHEHPAV TAPVVLKRLK QKDEEWRRAQ REWNKVWREL EQKVFFKSLD HLGLTFKQAD KKLLTTKQLI SEISSIKVDQ T NKKIHWLT PKPKSQLDFD FPDKNIFYDI LCLADTFITH TTAYSNPDKE RLKDLLKYFI SLFFSISFEK IEESLYSHKQ NV SESSGSD DGSSIASRKR PYQQEMSLLD ILHRSRYQKL KRSNDEDGKV PQLSEPPEEE PNTIEEEELI DEEAKNPWLT GNL VEEANS QGIIQNRSIF NLFANTNIYI FFRHWTTIYE RLLEIKQMNE RVTKEINTRS TVTFAKDLDL LSSQLSEMGL DFVG EDAYK QVLRLSRRLI NGDLEHQWFE ESLRQAYNNK AFKLYTIDKV TQSLVKHAHT LMTDAKTAEI MALFVKDRNA STTSA KDQI IYRLQVRSHM SNTENMFRIE FDKRTLHVSI QYIALDDLTL KEPKADEDKW KYYVTSYALP HPTEGIPHEK LKIPFL ERL IEFGQDIDGT EVDEEFSPEG ISVSTLKIKI QPITYQLHIE NGSYDVFTRK ATNKYPTIAN DNTQKGMVSQ KKELISK FL DCAVGLRNNL DEAQKLSMQK KWENLKDSIA KTSAGNQGIE SETEKGKITK QEQSDNLDSS TASVLPASIT TVPQDDNI E TTGNTESSDK GAKIQ UniProtKB: Transcriptional regulatory protein SIN3 |
-分子 #3: Histone deacetylase RPD3
| 分子 | 名称: Histone deacetylase RPD3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: histone deacetylase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.961957 KDa |
| 配列 | 文字列: MVYEATPFDP ITVKPSDKRR VAYFYDADVG NYAYGAGHPM KPHRIRMAHS LIMNYGLYKK MEIYRAKPAT KQEMCQFHTD EYIDFLSRV TPDNLEMFKR ESVKFNVGDD CPVFDGLYEY CSISGGGSME GAARLNRGKC DVAVNYAGGL HHAKKSEASG F CYLNDIVL ...文字列: MVYEATPFDP ITVKPSDKRR VAYFYDADVG NYAYGAGHPM KPHRIRMAHS LIMNYGLYKK MEIYRAKPAT KQEMCQFHTD EYIDFLSRV TPDNLEMFKR ESVKFNVGDD CPVFDGLYEY CSISGGGSME GAARLNRGKC DVAVNYAGGL HHAKKSEASG F CYLNDIVL GIIELLRYHP RVLYIDIDVH HGDGVEEAFY TTDRVMTCSF HKYGEFFPGT GELRDIGVGA GKNYAVNVPL RD GIDDATY RSVFEPVIKK IMEWYQPSAV VLQCGGDSLS GDRLGCFNLS MEGHANCVNY VKSFGIPMMV VGGGGYTMRN VAR TWCFET GLLNNVVLDK DLPYNEYYEY YGPDYKLSVR PSNMFNVNTP EYLDKVMTNI FANLENTKYA PSVQLNHTPR DAED LGDVE EDSAEAKDTK GGSQYARDLH VEHDNEFY UniProtKB: Histone deacetylase RPD3 |
-分子 #4: RCO1 isoform 1
| 分子 | 名称: RCO1 isoform 1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 78.951305 KDa |
| 配列 | 文字列: MDTSKKDTTR SPSHSNSSSP SSSSLSSSSS KEKKRPKRLS SQNVNYDLKR RKIITSEGIE RSFKNEHSNL AVEDNIPEEE PKELLEKDS KGNIIKLNEP STISEDSKVS VTGLPLNKGP SEKIKRESLW NYRKNLGGQS NNSEMTLVPS KRFTQVPKNF Q DLNRNDLK ...文字列: MDTSKKDTTR SPSHSNSSSP SSSSLSSSSS KEKKRPKRLS SQNVNYDLKR RKIITSEGIE RSFKNEHSNL AVEDNIPEEE PKELLEKDS KGNIIKLNEP STISEDSKVS VTGLPLNKGP SEKIKRESLW NYRKNLGGQS NNSEMTLVPS KRFTQVPKNF Q DLNRNDLK TFLTENMTEE SNIRSTIGWN GDIINRTRDR EPESDRDNKK LSNIRTKIIL STNATYDSKS KLFGQNSIKS TS NASEKIF RDKNNSTIDF ENEDFCSACN QSGSFLCCDT CPKSFHFLCL DPPIDPNNLP KGDWHCNECK FKIFINNSMA TLK KIESNF IKQNNNVKIF AKLLFNIDSH NPKQFQLPNY IKETFPAVKT GSRGQYSDEN DKIPLTDRQL FNTSYGQSIT KLDS YNPDT HIDSNSGKFL ICYKCNQTRL GSWSHPENSR LIMTCDYCQT PWHLDCVPRA SFKNLGSKWK CPLHSPTKVY KKIHH CQED NSVNYKVWKK QRLINKKNQL YYEPLQKIGY QNNGNIQIIP TTSHTDYDFN QDFKITQIDE NSIKYDFFDK IYKSKM VQK RKLFQFQESL IDKLVSNGSQ NGNSEDNMVK DIASLIYFQV SNNDKSSNNK SASKSNNLRK LWDLKELTNV VVPNELD SI QFNDFSSDEI KHLLYLKKII ESKPKEELLK FLNIENPENQ SE UniProtKB: RCO1 isoform 1 |
-分子 #5: Chromatin modification-related protein EAF3
| 分子 | 名称: Chromatin modification-related protein EAF3 / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.266406 KDa |
| 配列 | 文字列: MVDLEQEFAL GGRCLAFHGP LMYEAKILKI WDPSSKMYTS IPNDKPGGSS QATKEIKPQK LGEDESIPEE IINGKCFFIH YQGWKSSWD EWVGYDRIRA YNEENIAMKK RLANEAKEAK KSLLEQQKKK KLSTSLGGPS NGGKRKGDSR SNASISKSTS Q SFLTSSVS ...文字列: MVDLEQEFAL GGRCLAFHGP LMYEAKILKI WDPSSKMYTS IPNDKPGGSS QATKEIKPQK LGEDESIPEE IINGKCFFIH YQGWKSSWD EWVGYDRIRA YNEENIAMKK RLANEAKEAK KSLLEQQKKK KLSTSLGGPS NGGKRKGDSR SNASISKSTS Q SFLTSSVS GRKSGRSSAN SLHPGSSLRS SSDQNGNDDR RRSSSLSPNM LHHIAGYPTP KISLQIPIKL KSVLVDDWEY VT KDKKICR LPADVTVEMV LNKYEHEVSQ ELESPGSQSQ LSEYCAGLKL YFDKCLGNML LYRLERLQYD ELLKKSSKDQ KPL VPIRIY GAIHLLRLIS VLPELISSTT MDLQSCQLLI KQTEDFLVWL LMHVDEYFND KDPNRSDDAL YVNTSSQYEG VALG M UniProtKB: Chromatin modification-related protein EAF3 |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.37 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 107252 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)