登録情報 データベース : EMDB / ID : EMD-35274タイトル The PKR and E3L complex 複合体 : The PKR and E3L complexタンパク質・ペプチド : RNA-binding protein E3タンパク質・ペプチド : Interferon-induced, double-stranded RNA-activated protein kinaseタンパク質・ペプチド : RNA-binding protein E3 / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
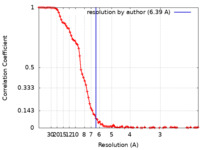
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / 手法 / / 解像度 : 6.39 Å Han CW / Kim HJ 資金援助 1件 Organization Grant number 国 Not funded
ジャーナル : Biochem Biophys Res Commun / 年 : 2023タイトル : Structural study of novel vaccinia virus E3L and dsRNA-dependent protein kinase complex.著者 : Hyeon Jin Kim / Chang Woo Han / Mi Suk Jeong / Se Bok Jang / 要旨 : E3L (RNA-binding protein E3) is one of the key IFN resistance genes encoded by VV and consists of 190 amino acids with a highly conserved carboxy-terminal double-stranded RNA-binding domain (dsRBD). ... E3L (RNA-binding protein E3) is one of the key IFN resistance genes encoded by VV and consists of 190 amino acids with a highly conserved carboxy-terminal double-stranded RNA-binding domain (dsRBD). PKR (dsRNA-dependent protein kinase) is an IFN-induced protein involved in anti-cell and antiviral activity. PKR inhibits the initiation of translation through alpha subunit of the initiation factor eIF2 (eIF2α) and mediates several transcription factors such as NF-κB, p53 or STATs. Activated PKR also induces apoptosis in vaccinia virus infection. E3L is required for viral IFN resistance and directly binds to PKR to block activation of PKR. In this work, we determined the three-dimensional complex structure of E3L and PKR using cryo-EM and determined the important residues involved in the interaction. In addition, PKR peptide binds to E3L and can increase protein levels of phosphorus-PKR and phosphorus-eIF2α-induced cell apoptosis through upregulation of phosphorus-PKR in HEK293 cells. Taken together, structural insights into E3L and PKR will provide a new optimization and development of vaccinia virus drugs. 履歴 登録 2023年2月7日 - ヘッダ(付随情報) 公開 2023年6月28日 - マップ公開 2023年6月28日 - 更新 2024年7月3日 - 現状 2024年7月3日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) /  Vaccinia virus WR (ウイルス)
Vaccinia virus WR (ウイルス) データ登録者
データ登録者 引用
引用 ジャーナル: Biochem Biophys Res Commun / 年: 2023
ジャーナル: Biochem Biophys Res Commun / 年: 2023
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_35274.map.gz
emd_35274.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-35274-v30.xml
emd-35274-v30.xml emd-35274.xml
emd-35274.xml EMDBヘッダ
EMDBヘッダ emd_35274_fsc.xml
emd_35274_fsc.xml FSCデータファイル
FSCデータファイル emd_35274.png
emd_35274.png emd-35274.cif.gz
emd-35274.cif.gz emd_35274_additional_1.map.gz
emd_35274_additional_1.map.gz emd_35274_half_map_1.map.gz
emd_35274_half_map_1.map.gz emd_35274_half_map_2.map.gz
emd_35274_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-35274
http://ftp.pdbj.org/pub/emdb/structures/EMD-35274 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35274
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35274 emd_35274_validation.pdf.gz
emd_35274_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_35274_full_validation.pdf.gz
emd_35274_full_validation.pdf.gz emd_35274_validation.xml.gz
emd_35274_validation.xml.gz emd_35274_validation.cif.gz
emd_35274_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35274
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35274 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35274
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35274
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_35274.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_35274.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Vaccinia virus WR (ウイルス) / 株: Western Reserve
Vaccinia virus WR (ウイルス) / 株: Western Reserve
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Vaccinia virus WR (ウイルス) / 株: Western Reserve
Vaccinia virus WR (ウイルス) / 株: Western Reserve
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X