+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc4/Vtc3/Vtc1) | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | polyphosphate polymerase / VTC complex / coupled synthesis and translocation / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報vacuolar transporter chaperone complex / ATP-polyphosphate phosphotransferase / polyphosphate biosynthetic process / engulfment of target by autophagosome / polyphosphate kinase activity / microautophagy / vacuole fusion, non-autophagic / polyphosphate metabolic process / intracellular phosphate ion homeostasis / inositol hexakisphosphate binding ...vacuolar transporter chaperone complex / ATP-polyphosphate phosphotransferase / polyphosphate biosynthetic process / engulfment of target by autophagosome / polyphosphate kinase activity / microautophagy / vacuole fusion, non-autophagic / polyphosphate metabolic process / intracellular phosphate ion homeostasis / inositol hexakisphosphate binding / vacuolar transport / fungal-type vacuole membrane / vacuolar membrane / autophagosome membrane / cell periphery / cell cortex / cytoplasmic vesicle / nuclear membrane / calmodulin binding / mRNA binding / endoplasmic reticulum membrane / endoplasmic reticulum / membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
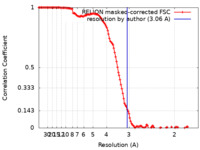
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.06 Å | |||||||||||||||
 データ登録者 データ登録者 | Mayer A / Wu S / Ye S | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2023 ジャーナル: EMBO J / 年: 2023タイトル: Cryo-EM structure of the polyphosphate polymerase VTC reveals coupling of polymer synthesis to membrane transit. 著者: Wei Liu / Jiening Wang / Véronique Comte-Miserez / Mengyu Zhang / Xuejing Yu / Qingfeng Chen / Henning Jacob Jessen / Andreas Mayer / Shan Wu / Sheng Ye /    要旨: The eukaryotic vacuolar transporter chaperone (VTC) complex acts as a polyphosphate (polyP) polymerase that synthesizes polyP from adenosine triphosphate (ATP) and translocates polyP across the ...The eukaryotic vacuolar transporter chaperone (VTC) complex acts as a polyphosphate (polyP) polymerase that synthesizes polyP from adenosine triphosphate (ATP) and translocates polyP across the vacuolar membrane to maintain an intracellular phosphate (P ) homeostasis. To discover how the VTC complex performs its function, we determined a cryo-electron microscopy structure of an endogenous VTC complex (Vtc4/Vtc3/Vtc1) purified from Saccharomyces cerevisiae at 3.1 Å resolution. The structure reveals a heteropentameric architecture of one Vtc4, one Vtc3, and three Vtc1 subunits. The transmembrane region forms a polyP-selective channel, likely adopting a resting state conformation, in which a latch-like, horizontal helix of Vtc4 limits the entrance. The catalytic Vtc4 central domain is located on top of the pseudo-symmetric polyP channel, creating a strongly electropositive pathway for nascent polyP that can couple synthesis to translocation. The SPX domain of the catalytic Vtc4 subunit positively regulates polyP synthesis by the VTC complex. The noncatalytic Vtc3 regulates VTC through a phosphorylatable loop. Our findings, along with the functional data, allow us to propose a mechanism of polyP channel gating and VTC complex activation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35208.map.gz emd_35208.map.gz | 166.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35208-v30.xml emd-35208-v30.xml emd-35208.xml emd-35208.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35208_fsc.xml emd_35208_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35208.png emd_35208.png | 156.3 KB | ||
| Filedesc metadata |  emd-35208.cif.gz emd-35208.cif.gz | 6.9 KB | ||
| その他 |  emd_35208_additional_1.map.gz emd_35208_additional_1.map.gz emd_35208_half_map_1.map.gz emd_35208_half_map_1.map.gz emd_35208_half_map_2.map.gz emd_35208_half_map_2.map.gz | 155.9 MB 140.4 MB 140.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35208 http://ftp.pdbj.org/pub/emdb/structures/EMD-35208 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35208 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35208 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35208_validation.pdf.gz emd_35208_validation.pdf.gz | 937.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35208_full_validation.pdf.gz emd_35208_full_validation.pdf.gz | 936.7 KB | 表示 | |
| XML形式データ |  emd_35208_validation.xml.gz emd_35208_validation.xml.gz | 20.1 KB | 表示 | |
| CIF形式データ |  emd_35208_validation.cif.gz emd_35208_validation.cif.gz | 26.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35208 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35208 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35208 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35208 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8i6vMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35208.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35208.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.851 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_35208_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35208_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35208_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc...
| 全体 | 名称: Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc4/Vtc3/Vtc1) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc...
| 超分子 | 名称: Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc4/Vtc3/Vtc1) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Vacuolar transporter chaperone complex subunit 1
| 分子 | 名称: Vacuolar transporter chaperone complex subunit 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.388053 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSAPLLQRT PGKKIALPTR VEPKVFFANE RTFLSWLNFT VMLGGLGVGL LNFGDKIGRV SAGLFTFVAM GTMIYALVTY HWRAAAIRR RGSGPYDDRL GPTLLCFFLL VAVIINFILR LKYNDANTKL UniProtKB: Vacuolar transporter chaperone complex subunit 1 |
-分子 #2: Vacuolar transporter chaperone 3 complex subunit 3
| 分子 | 名称: Vacuolar transporter chaperone 3 complex subunit 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 96.684312 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLFGIKLAND VYPPWKDSYI DYERLKKLLK ESVIHDGRSS VDSWSERNES DFVEALDKEL EKVYTFQISK YNAVLRKLDD LEENTKSAE KIQKINSEQF KNTLEECLDE AQRLDNFDRL NFTGFIKIVK KHDKLHPNYP SVKSLLQVRL KELPFNNSEE Y SPLLYRIS ...文字列: MLFGIKLAND VYPPWKDSYI DYERLKKLLK ESVIHDGRSS VDSWSERNES DFVEALDKEL EKVYTFQISK YNAVLRKLDD LEENTKSAE KIQKINSEQF KNTLEECLDE AQRLDNFDRL NFTGFIKIVK KHDKLHPNYP SVKSLLQVRL KELPFNNSEE Y SPLLYRIS YLYEFLRSNY DHPNTVSKSL ASTSKLSHFS NLEDASFKSY KFWVHDDNIM EVKARILRHL PALVYASVPN EN DDFVDNL ESDVRVQPEA RLNIGSKSNS LSSDGNSNQD VEIGKSKSVI FPQSYDPTIT TLYFDNDFFD LYNNRLLKIS GAP TLRLRW IGKLLDKPDI FLEKRTFTEN TETGNSSFEE IRLQMKAKFI NNFIFKNDPS YKNYLINQLR ERGTQKEELE KLSR DFDNI QNFIVEEKLQ PVLRATYNRT AFQIPGDQSI RVTIDSNIMY IREDSLDKNR PIRNPENWHR DDIDSNIPNP LRFLR AGEY SKFPYSVMEI KVINQDNSQM PNYEWIKDLT NSHLVNEVPK FSLYLQGVAS LFGEDDKYVN ILPFWLPDLE TDIRKN PQE AYEEEKKTLQ KQKSIHDKLD NMRRLSKISV PDGKTTERQG QKDQNTRHVI ADLEDHESSD EEGTALPKKS AVKKGKK FK TNAAFLKILA GKNISENGND PYSDDTDSAS SFQLPPGVKK PVHLLKNAGP VKVEAKVWLA NERTFNRWLS VTTLLSVL T FSIYNSVQKA EFPQLADLLA YVYFFLTLFC GVWAYRTYLK RLTLIKGRSG KHLDAPVGPI LVAVVLIVTL VVNFSVAFK EAARRERGLV NVSSQPSLPR TLKPIQDFIF NLVGE UniProtKB: Vacuolar transporter chaperone 3 complex subunit 3 |
-分子 #3: Vacuolar transporter chaperone complex subunit 4
| 分子 | 名称: Vacuolar transporter chaperone complex subunit 4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: ATP-polyphosphate phosphotransferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 83.268664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKFGEHLSKS LIRQYSYYYI SYDDLKTELE DNLSKNNGQW TQELETDFLE SLEIELDKVY TFCKVKHSEV FRRVKEVQEQ VQHTVRLLD SNNPPTQLDF EILEEELSDI IADVHDLAKF SRLNYTGFQK IIKKHDKKTG FILKPVFQVR LDSKPFFKEN Y DELVVKIS ...文字列: MKFGEHLSKS LIRQYSYYYI SYDDLKTELE DNLSKNNGQW TQELETDFLE SLEIELDKVY TFCKVKHSEV FRRVKEVQEQ VQHTVRLLD SNNPPTQLDF EILEEELSDI IADVHDLAKF SRLNYTGFQK IIKKHDKKTG FILKPVFQVR LDSKPFFKEN Y DELVVKIS QLYDIARTSG RPIKGDSSAG GKQQNFVRQT TKYWVHPDNI TELKLIILKH LPVLVFNTNK EFEREDSAIT SI YFDNENL DLYYGRLRKD EGAEAHRLRW YGGMSTDTIF VERKTHREDW TGEKSVKARF ALKERHVNDF LKGKYTVDQV FAK MRKEGK KPMNEIENLE ALASEIQYVM LKKKLRPVVR SFYNRTAFQL PGDARVRISL DTELTMVRED NFDGVDRTHK NWRR TDIGV DWPFKQLDDK DICRFPYAVL EVKLQTQLGQ EPPEWVRELV GSHLVEPVPK FSKFIHGVAT LLNDKVDSIP FWLPQ MDVD IRKPPLPTNI EITRPGRSDN EDNDFDEDDE DDAALVAAMT NAPGNSLDIE ESVGYGATSA PTSNTNHVVE SANAAY YQR KIRNAENPIS KKYYEIVAFF DHYFNGDQIS KIPKGTTFDT QIRAPPGKTI CVPVRVEPKV YFATERTYLS WLSISIL LG GVSTTLLTYG SPTAMIGSIG FFITSLAVLI RTVMVYAKRV VNIRLKRAVD YEDKIGPGMV SVFLILSILF SFFCNLVA K UniProtKB: Vacuolar transporter chaperone complex subunit 4 |
-分子 #4: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(tri...
| 分子 | 名称: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(trimethylammonio)ethyl phosphate タイプ: ligand / ID: 4 / コピー数: 1 / 式: POV |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-POV: |
-分子 #5: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 5 / コピー数: 3 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-分子 #6: TRIPHOSPHATE
| 分子 | 名称: TRIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: 3PO |
|---|---|
| 分子量 | 理論値: 257.955 Da |
| Chemical component information |  ChemComp-3PO: |
-分子 #7: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-分子 #8: water
| 分子 | 名称: water / タイプ: ligand / ID: 8 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X