+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lb2Cas12a RNA DNA complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | complex / ANTIMICROBIAL PROTEIN | |||||||||
| 生物種 |  Lachnospiraceae bacterium MA2020 (バクテリア) / synthetic construct (人工物) Lachnospiraceae bacterium MA2020 (バクテリア) / synthetic construct (人工物) | |||||||||
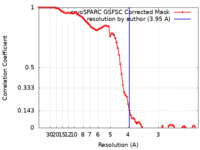
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.95 Å | |||||||||
 データ登録者 データ登録者 | Li J / sivaraman J / Satoru M | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Biol / 年: 2023 ジャーナル: PLoS Biol / 年: 2023タイトル: Structures of apo Cas12a and its complex with crRNA and DNA reveal the dynamics of ternary complex formation and target DNA cleavage. 著者: Li Jianwei / Chacko Jobichen / Satoru Machida / Sun Meng / Randy J Read / Chen Hongying / Shi Jian / Yuren Adam Yuan / J Sivaraman /   要旨: Cas12a is a programmable nuclease for adaptive immunity against invading nucleic acids in CRISPR-Cas systems. Here, we report the crystal structures of apo Cas12a from Lachnospiraceae bacterium ...Cas12a is a programmable nuclease for adaptive immunity against invading nucleic acids in CRISPR-Cas systems. Here, we report the crystal structures of apo Cas12a from Lachnospiraceae bacterium MA2020 (Lb2) and the Lb2Cas12a+crRNA complex, as well as the cryo-EM structure and functional studies of the Lb2Cas12a+crRNA+DNA complex. We demonstrate that apo Lb2Cas12a assumes a unique, elongated conformation, whereas the Lb2Cas12a+crRNA binary complex exhibits a compact conformation that subsequently rearranges to a semi-open conformation in the Lb2Cas12a+crRNA+DNA ternary complex. Notably, in solution, apo Lb2Cas12a is dynamic and can exist in both elongated and compact forms. Residues from Met493 to Leu523 of the WED domain undergo major conformational changes to facilitate the required structural rearrangements. The REC lobe of Lb2Cas12a rotates 103° concomitant with rearrangement of the hinge region close to the WED and RuvC II domains to position the RNA-DNA duplex near the catalytic site. Our findings provide insight into crRNA recognition and the mechanism of target DNA cleavage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35191.map.gz emd_35191.map.gz | 31.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35191-v30.xml emd-35191-v30.xml emd-35191.xml emd-35191.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35191_fsc.xml emd_35191_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35191.png emd_35191.png | 60.1 KB | ||
| Filedesc metadata |  emd-35191.cif.gz emd-35191.cif.gz | 6.6 KB | ||
| その他 |  emd_35191_additional_1.map.gz emd_35191_additional_1.map.gz emd_35191_half_map_1.map.gz emd_35191_half_map_1.map.gz emd_35191_half_map_2.map.gz emd_35191_half_map_2.map.gz | 2.1 MB 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35191 http://ftp.pdbj.org/pub/emdb/structures/EMD-35191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35191 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35191 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_35191_validation.pdf.gz emd_35191_validation.pdf.gz | 720.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_35191_full_validation.pdf.gz emd_35191_full_validation.pdf.gz | 720 KB | 表示 | |
| XML形式データ |  emd_35191_validation.xml.gz emd_35191_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_35191_validation.cif.gz emd_35191_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35191 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35191 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35191 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-35191 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35191.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35191.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.105 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_35191_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35191_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35191_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LbCas12a-crRNA-DNA ternary complex
| 全体 | 名称: LbCas12a-crRNA-DNA ternary complex |
|---|---|
| 要素 |
|
-超分子 #1: LbCas12a-crRNA-DNA ternary complex
| 超分子 | 名称: LbCas12a-crRNA-DNA ternary complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium MA2020 (バクテリア) Lachnospiraceae bacterium MA2020 (バクテリア) |
| 分子量 | 理論値: 140 KDa |
-分子 #1: Lb2Cas12a
| 分子 | 名称: Lb2Cas12a / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium MA2020 (バクテリア) Lachnospiraceae bacterium MA2020 (バクテリア) |
| 分子量 | 理論値: 141.200609 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYYESLTKQY PVSKTIRNEL IPIGKTLDNI RQNNILESDV KRKQNYEHVK GILDEYHKQL INEALDNCTL PSLKIAAEIY LKNQKEVSD REDFNKTQDL LRKEVVEKLK AHENFTKIGK KDILDLLEKL PSISEDDYNA LESFRNFYTY FTSYNKVREN L YSDKEKSS ...文字列: MYYESLTKQY PVSKTIRNEL IPIGKTLDNI RQNNILESDV KRKQNYEHVK GILDEYHKQL INEALDNCTL PSLKIAAEIY LKNQKEVSD REDFNKTQDL LRKEVVEKLK AHENFTKIGK KDILDLLEKL PSISEDDYNA LESFRNFYTY FTSYNKVREN L YSDKEKSS TVAYRLINEN FPKFLDNVKS YRFVKTAGIL ADGLGEEEQD SLFIVETFNK TLTQDGIDTY NSQVGKINSS IN LYNQKNQ KANGFRKIPK MKMLYKQILS DREESFIDEF QSDEVLIDNV ESYGSVLIES LKSSKVSAFF DALRESKGKN VYV KNDLAK TAMSNIVFEN WRTFDDLLNQ EYDLANENKK KDDKYFEKRQ KELKKNKSYS LEHLCNLSED SCNLIENYIH QISD DIENI IINNETFLRI VINEHDRSRK LAKNRKAVKA IKDFLDSIKV LERELKLINS SGQELEKDLI VYSAHEELLV ELKQV DSLY NMTRNYLTKK PFSTEKVKLN FNRSTLLNGW DRNKETDNLG VLLLKDGKYY LGIMNTSANK AFVNPPVAKT EKVFKK VDY KLLPVPNQML PKVFFAKSNI DFYNPSSEIY SNYKKGTHKK GNMFSLEDCH NLIDFFKESI SKHEDWSKFG FKFSDTA SY NDISEFYREV EKQGYKLTYT DIDETYINDL IERNELYLFQ IYNKDFSMYS KGKLNLHTLY FMMLFDQRNI DDVVYKLN G EAEVFYRPAS ISEDELIIHK AGEEIKNKNP NRARTKETST FSYDIVKDKR YSKDKFTLHI PITMNFGVDE VKRFNDAVN SAIRIDENVN VIGIDRGERN LLYVVVIDSK GNILEQISLN SIINKEYDIE TDYHALLDER EGGRDKARKD WNTVENIRDL KAGYLSQVV NVVAKLVLKY NAIICLEDLN FGFKRGRQKV EKQVYQKFEK MLIDKLNYLV IDKSREQTSP KELGGALNAL Q LTSKFKSF KELGKQSGVI YYVPAYLTSK IDPTTGFANL FYMKCENVEK SKRFFDGFDF IRFNALENVF EFGFDYRSFT QR ACGINSK WTVCTNGERI IKYRNPDKNN MFDEKVVVVT DEMKNLFEQY KIPYEDGRNV KDMIISNEEA EFYRRLYRLL QQT LQMRNS TSDGTRDYII SPVKNKREAY FNSELSDGSV PKDADANGAY NIARKGLWVL EQIRQKSEGE KINLAMTNAE WLEY AQTHL L |
-分子 #2: RNA (33-MER)
| 分子 | 名称: RNA (33-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium MA2020 (バクテリア) Lachnospiraceae bacterium MA2020 (バクテリア) |
| 分子量 | 理論値: 10.452219 KDa |
| 配列 | 文字列: AAUUUCUACU AAUUGUAGAU GCCGCUACCC CGA |
-分子 #3: DNA (25-MER)
| 分子 | 名称: DNA (25-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.780007 KDa |
| 配列 | 文字列: (DT)(DG)(DG)(DT)(DC)(DG)(DG)(DG)(DG)(DT) (DA)(DG)(DC)(DG)(DG)(DC)(DT)(DA)(DA)(DA) (DG)(DC)(DA)(DC)(DT) |
-分子 #4: DNA (5'-D(*AP*GP*TP*GP*CP*TP*TP*TP*A)-3')
| 分子 | 名称: DNA (5'-D(*AP*GP*TP*GP*CP*TP*TP*TP*A)-3') / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 2.745821 KDa |
| 配列 | 文字列: (DA)(DG)(DT)(DG)(DC)(DT)(DT)(DT)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 50 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295.15 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 温度 | 最低: 140.0 K / 最高: 150.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 2560 / 平均露光時間: 3.49 sec. / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)