+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cryo-EM structure of cellobiose phosphorylase from Clostridium thermocellum ( varient) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cellobiose phosphorylase / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellobiose phosphorylase / cellobiose phosphorylase activity / cellulose catabolic process / carbohydrate binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Acetivibrio thermocellus (バクテリア) Acetivibrio thermocellus (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.19 Å | |||||||||
 データ登録者 データ登録者 | Iriya S / Kuga T / Sunagawa N / Igarashi K | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: The cryo-EM structure of cellobiose phosphorylase from Clostridium thermocellum 著者: Iriya S / Kuga T / Sunagawa N / Igarashi K | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34924.map.gz emd_34924.map.gz | 219.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34924-v30.xml emd-34924-v30.xml emd-34924.xml emd-34924.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
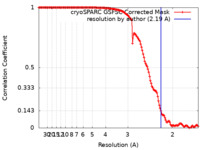
| FSC (解像度算出) |  emd_34924_fsc.xml emd_34924_fsc.xml | 13.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34924.png emd_34924.png | 110.8 KB | ||
| マスクデータ |  emd_34924_msk_1.map emd_34924_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34924.cif.gz emd-34924.cif.gz | 6.1 KB | ||
| その他 |  emd_34924_half_map_1.map.gz emd_34924_half_map_1.map.gz emd_34924_half_map_2.map.gz emd_34924_half_map_2.map.gz | 226.7 MB 226.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34924 http://ftp.pdbj.org/pub/emdb/structures/EMD-34924 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34924 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34924 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34924_validation.pdf.gz emd_34924_validation.pdf.gz | 904.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34924_full_validation.pdf.gz emd_34924_full_validation.pdf.gz | 904.2 KB | 表示 | |
| XML形式データ |  emd_34924_validation.xml.gz emd_34924_validation.xml.gz | 22 KB | 表示 | |
| CIF形式データ |  emd_34924_validation.cif.gz emd_34924_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34924 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34924 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34924 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34924 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34924.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34924.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
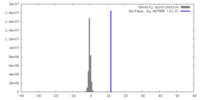
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34924_msk_1.map emd_34924_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
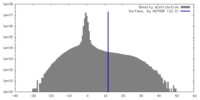
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34924_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34924_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cellobiose phosphorylase
| 全体 | 名称: Cellobiose phosphorylase |
|---|---|
| 要素 |
|
-超分子 #1: Cellobiose phosphorylase
| 超分子 | 名称: Cellobiose phosphorylase / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Acetivibrio thermocellus (バクテリア) Acetivibrio thermocellus (バクテリア) |
| 分子量 | 理論値: 180 KDa |
-分子 #1: Cellobiose phosphorylase
| 分子 | 名称: Cellobiose phosphorylase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: cellobiose phosphorylase |
|---|---|
| 由来(天然) | 生物種:  Acetivibrio thermocellus (バクテリア) Acetivibrio thermocellus (バクテリア) |
| 分子量 | 理論値: 93.973258 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKFGFFDDA NKEYVITVPR TPYPWINYLG TENFFSLISN TAGGYSFYRD ARLRRITRYR YNNVPIDMGG RYFYIYDNGD FWSPGWSPV KRELESYESR HGLGYTKIAG KRNGIKAEVT FFVPLNYNGE VQKLILKNEG QDKKKITLFS FIEFSLWNAY D DMTNFQRN ...文字列: MGKFGFFDDA NKEYVITVPR TPYPWINYLG TENFFSLISN TAGGYSFYRD ARLRRITRYR YNNVPIDMGG RYFYIYDNGD FWSPGWSPV KRELESYESR HGLGYTKIAG KRNGIKAEVT FFVPLNYNGE VQKLILKNEG QDKKKITLFS FIEFSLWNAY D DMTNFQRN FSTGEVEIEG SVIYHKTEYR ERRNHYAFYS VNAKISGFDS DRDSFIGLYN GFDAPQAVVN GKSNNSVADG WA PIASHSI EIELNPGEQK EYVFIIGYVE NKDEEKWESK GVINKKKAYE MIEQFNTVEK VDKAFEELKS YWNALLSKYF LES HDEKLN RMVNIWNQYQ SMVTFNMSRS ASYFESGIGR GMGFRDSNQD LLGFVHQIPE RARERLLDLA ATQLEDGSAY HQYQ PLTKK GNNEIGSNFN DDPLWLILAT AAYIKETGDY SILKEQVPFN NDPSKADTMF EHLTRSFYHV VNNLGPHGLP LIGRA DWND CLNLNCFSTV PDESFQTTTS KDGKVAESVM IAGMFVFIGK DYVKLSEYMG LEEEARKAQQ HIDAMKEAIL KYGYDG EWF LRAYDDFGRK VGSKENEEGK IFIESQGFSV MAEIGLEDGK ALKALDSVKK YLDTPYGLVL QNPAFTRYYI EYGEIST YP PGYKENAGIF SHNNAWIISA ETVVGRGDMA FDYYRKIAPA YIEDVSDIHK LEPYVYAQMV AGKDAKRHGE AKNSWLTG T AAWNFVAISQ WILGVKPDYD GLKIDPSIPK AWDGYKVTRY FRGSTYEITV KNPNHVSKGV AKITVDGNEI SGNILPVFN DGKTHKVEVI MGLEHHHHHH UniProtKB: Cellobiose phosphorylase |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 333 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.5 構成要素:
詳細: 20 mM MES, 70 mM NaCl | |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV / 詳細: Vitrification carried out in nitrogen atmosphere.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3744 / 平均露光時間: 5.567 sec. / 平均電子線量: 49.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)