+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of MPXV polymerase holoenzyme in replicating state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MPXV / Polymerase / Replication / REPLICATION-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral DNA genome replication / uracil DNA N-glycosylase activity / nucleotide-excision repair, DNA gap filling / DNA replication proofreading / 3'-5'-DNA exonuclease activity / SOS response / base-excision repair, gap-filling / DNA recombination / DNA replication / DNA-directed DNA polymerase ...viral DNA genome replication / uracil DNA N-glycosylase activity / nucleotide-excision repair, DNA gap filling / DNA replication proofreading / 3'-5'-DNA exonuclease activity / SOS response / base-excision repair, gap-filling / DNA recombination / DNA replication / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / DNA repair / nucleotide binding / DNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) | |||||||||
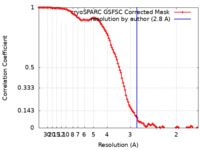
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Peng Q / Xie YF / Kuai L / Wang H / Qi JX / Gao F / Shi Y | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structure of monkeypox virus DNA polymerase holoenzyme. 著者: Qi Peng / Yufeng Xie / Lu Kuai / Han Wang / Jianxun Qi / George F Gao / Yi Shi /  要旨: The World Health Organization declared mpox (or monkeypox) a public health emergency of international concern in July 2022, and prophylactic and therapeutic measures are in urgent need. The monkeypox ...The World Health Organization declared mpox (or monkeypox) a public health emergency of international concern in July 2022, and prophylactic and therapeutic measures are in urgent need. The monkeypox virus (MPXV) has its own DNA polymerase F8, together with the processive cofactors A22 and E4, constituting the polymerase holoenzyme for genome replication. Here, we determined the holoenzyme structure in complex with DNA using cryo-electron microscopy at the global resolution of ~2.8 angstroms. The holoenzyme possesses an architecture that suggests a "forward sliding clamp" processivity mechanism for viral DNA replication. MPXV polymerase has a DNA binding mode similar to that of other B-family DNA polymerases from different species. These findings reveal the mechanism of the MPXV genome replication and may guide the development of anti-poxvirus drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34731.map.gz emd_34731.map.gz | 111 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34731-v30.xml emd-34731-v30.xml emd-34731.xml emd-34731.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34731_fsc.xml emd_34731_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34731.png emd_34731.png | 35.7 KB | ||
| Filedesc metadata |  emd-34731.cif.gz emd-34731.cif.gz | 7 KB | ||
| その他 |  emd_34731_half_map_1.map.gz emd_34731_half_map_1.map.gz emd_34731_half_map_2.map.gz emd_34731_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34731 http://ftp.pdbj.org/pub/emdb/structures/EMD-34731 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34731 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34731 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34731_validation.pdf.gz emd_34731_validation.pdf.gz | 742.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34731_full_validation.pdf.gz emd_34731_full_validation.pdf.gz | 742.3 KB | 表示 | |
| XML形式データ |  emd_34731_validation.xml.gz emd_34731_validation.xml.gz | 18.9 KB | 表示 | |
| CIF形式データ |  emd_34731_validation.cif.gz emd_34731_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34731 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34731 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34731 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34731 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hg1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34731.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34731.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_34731_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
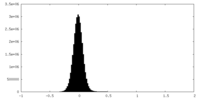
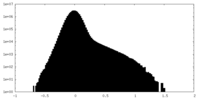
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34731_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
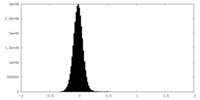
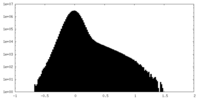
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MPXV DNA polymerase holoenzyme
| 全体 | 名称: MPXV DNA polymerase holoenzyme |
|---|---|
| 要素 |
|
-超分子 #1: MPXV DNA polymerase holoenzyme
| 超分子 | 名称: MPXV DNA polymerase holoenzyme / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
-分子 #1: DNA polymerase
| 分子 | 名称: DNA polymerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 119.939062 KDa |
| 組換発現 | 生物種:  Spodoptera (蝶・蛾) Spodoptera (蝶・蛾) |
| 配列 | 文字列: MWSHPQFEKG SGSWSHPQFE KGSGSMDVRC INWFESHGEN RFLYLKSRCR NGETVFIRFP HYFYYVVTDE IYQSLSPPPF NARPMGKMR TIDIDETISY NLDIKDRKCS VADMWLIEEP KKRSIQNATM DEFFNISWFY ISNGISPDGC YSLDEQYLTK I NNGCYHCD ...文字列: MWSHPQFEKG SGSWSHPQFE KGSGSMDVRC INWFESHGEN RFLYLKSRCR NGETVFIRFP HYFYYVVTDE IYQSLSPPPF NARPMGKMR TIDIDETISY NLDIKDRKCS VADMWLIEEP KKRSIQNATM DEFFNISWFY ISNGISPDGC YSLDEQYLTK I NNGCYHCD DPRNCFAKEI PRFDIPRSYL FLDIECHFDK KFPSVFINPI SHTSYCYIDL SGKRLLFTLI NEEMLTEQEI QE AVDRGCL RIQSLMEMDY ERELVLCSEI VLLRIAKQLL ELTFDYVVTF NGHNFDLRYI TNRLELLTGE KIIFRSPDKK EAV HLCIYE RNQSSHKGVC GMANTTFHVN NNNGTIFFDL YSFIQKSEKL DSYKLDSISK NAFSCMGKVL NRGVREMTFI GDDT TDAKG KADTFAKVLT TGNYVTVDED IICKVIRKDI LENGFKVVLS CPTLPNDIYK LSFGKDDIDL AQMYKDYNLN IALDM ARYC IHDACLCQYL WEYYGVETKT DAGAATYVLP QSMVFEYRAS TIIKGPLLKL LLETKTILVR SETKQKFPYE GGKVFA PKQ KMFSNNVLIF DYNSLYPNVC IFGNLSPETL VGVVVSTNRL EEEINNQLLL QKYPPPRYIT VHCEPRLPNL ISEIAIF DR SIEGTIPRLL RTFLAERARY KKMLKQATSS TEKAIYDSMQ YTYKIVANSV YGLMGFRNSA LYSYASAKSC TSIGRRMI L YLESVLNGAE LSNGMLRFAN TLSNPFYMDD RDINPIVKTS LPIDYRFRFR SVYGDTDSVF TEIDSQDVDK SIEIAKELE RLINSRVLFN NFKIEFEAVY KNLIMQSKKK YTTMKYSASS NSKSVPERIN KGTSETRRDV SKFHKNMIKT YKTRLSEMLS EGRMNSNQV CIDILRSLET DLRSEFDSRS SPLELFMLSR MHHSNYKSAD NPNMYLVTEY NKNNPETIEL GERYYFAYIC P ANVPWTKK LVNIKTYETI IDRSFKLGSN QRIFYEVYFK RLTSEIVNLL DNKVLCISFF QRMFGSRPTF YEA UniProtKB: DNA polymerase |
-分子 #2: E4R
| 分子 | 名称: E4R / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: uracil-DNA glycosylase |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 25.107742 KDa |
| 組換発現 | 生物種:  Spodoptera (蝶・蛾) Spodoptera (蝶・蛾) |
| 配列 | 文字列: MNSVTISHAP YTITYHDDWE PVMSQLVEFY NEVASWLLRD ETSPIPDKFF IQLKQPLRNK RVCVCGIDPY PKDGTGVPFE SPNFTKKSI KEIASSISRL TGVIDYKGYN LNIIDGVIPW NYYLSCKLGE TKSHAIYWDK ISKLLLQHIT KHVSVLYCLG K TDFSNIRA ...文字列: MNSVTISHAP YTITYHDDWE PVMSQLVEFY NEVASWLLRD ETSPIPDKFF IQLKQPLRNK RVCVCGIDPY PKDGTGVPFE SPNFTKKSI KEIASSISRL TGVIDYKGYN LNIIDGVIPW NYYLSCKLGE TKSHAIYWDK ISKLLLQHIT KHVSVLYCLG K TDFSNIRA KLESPVTTIV GYHPAARDHQ FEKDRSFEII NVLLELDNKT PINWAQGFIY UniProtKB: Uracil-DNA glycosylase |
-分子 #3: DNA polymerase processivity factor component A20
| 分子 | 名称: DNA polymerase processivity factor component A20 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 49.203926 KDa |
| 組換発現 | 生物種:  Spodoptera (蝶・蛾) Spodoptera (蝶・蛾) |
| 配列 | 文字列: MTSSADLTNL KELLSLYKSL RFSDSVAIEK YNSLVEWGTS TYWKIGVQKV TNVETSISDY YDEVKNKPFN IDPGYYIFLP VYFGSVFIY SKGKNMVELG SGNSFQIPDE IRSACNKVLD SDNGIDFLRF VLLNNRWIME DAISKYQSPV NIFKLASEYG L NIPNYLEI ...文字列: MTSSADLTNL KELLSLYKSL RFSDSVAIEK YNSLVEWGTS TYWKIGVQKV TNVETSISDY YDEVKNKPFN IDPGYYIFLP VYFGSVFIY SKGKNMVELG SGNSFQIPDE IRSACNKVLD SDNGIDFLRF VLLNNRWIME DAISKYQSPV NIFKLASEYG L NIPNYLEI EIEEDTLFDD ELYSIMERSF DDTFPKISIS YIKLGELKRQ VVDFFKFSFM YIESIKVDRI GDNIFIPSVI TK SGKKILV KDVDHLIRSK VREHTFVKVK KKNTFSILYD YDGNGTETRG EVIKRIIDTI GRDYYVNGKY FSKVGIAGLK QLT NKLDIN ECATVDELVD EINKSGTVKR KIKNQSVFDL SRECLGYPEA DFITLVNNMR FKIENCKVVN FNIENTNCLN NPSI ETIYG NFNQFVSIFN TVTDVKKRLF E UniProtKB: DNA polymerase processivity factor |
-分子 #4: DNA (25-MER)
| 分子 | 名称: DNA (25-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 7.681985 KDa |
| 配列 | 文字列: (DA)(DG)(DC)(DT)(DA)(DT)(DG)(DA)(DC)(DC) (DA)(DT)(DG)(DA)(DT)(DT)(DA)(DC)(DG)(DA) (DA)(DT)(DT)(DG)(DC) |
-分子 #5: DNA (38-MER)
| 分子 | 名称: DNA (38-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Monkeypox virus (サル痘ウイルス) Monkeypox virus (サル痘ウイルス) |
| 分子量 | 理論値: 11.677539 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(DC)(DA)(DC)(DG)(DA)(DA)(DT) (DT)(DA)(DA)(DG)(DC)(DA)(DA)(DT)(DT)(DC) (DG)(DT)(DA)(DA)(DT)(DC)(DA)(DT)(DG) (DG)(DT)(DC)(DA)(DT)(DA)(DG)(DC)(DT) |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: THYMIDINE-5'-TRIPHOSPHATE
| 分子 | 名称: THYMIDINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: TTP |
|---|---|
| 分子量 | 理論値: 482.168 Da |
| Chemical component information |  ChemComp-TTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X