+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | NARROW LEAF 1-close from Japonica | |||||||||
 マップデータ マップデータ | Electron Microscopy Volume Map of Protein NARROW LEAF 1. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Rice / panicle shape / photosynthetic efficiency / auxin transport / SURFACTANT PROTEIN | |||||||||
| 機能・相同性 | stem vascular tissue pattern formation / internode patterning / regulation of leaf development / leaf vascular tissue pattern formation / Peptidase S1, PA clan / nucleoplasm / cytoplasm / Protein NARROW LEAF 1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.89 Å | |||||||||
 データ登録者 データ登録者 | Zhang SJ / He YJ / Wang N / Zhang WJ / Liu CM | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: NARROW LEAF 1-close from Japonica 著者: Zhang SJ / He YJ / Wang N / Zhang WJ / Liu CM | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34607.map.gz emd_34607.map.gz | 28.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34607-v30.xml emd-34607-v30.xml emd-34607.xml emd-34607.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34607.png emd_34607.png | 40.2 KB | ||
| マスクデータ |  emd_34607_msk_1.map emd_34607_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34607.cif.gz emd-34607.cif.gz | 6.3 KB | ||
| その他 |  emd_34607_half_map_1.map.gz emd_34607_half_map_1.map.gz emd_34607_half_map_2.map.gz emd_34607_half_map_2.map.gz | 28.2 MB 28.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34607 http://ftp.pdbj.org/pub/emdb/structures/EMD-34607 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34607 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34607 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34607_validation.pdf.gz emd_34607_validation.pdf.gz | 908.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34607_full_validation.pdf.gz emd_34607_full_validation.pdf.gz | 908.5 KB | 表示 | |
| XML形式データ |  emd_34607_validation.xml.gz emd_34607_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_34607_validation.cif.gz emd_34607_validation.cif.gz | 12.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34607 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34607 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34607 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34607 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hasMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34607.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34607.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Electron Microscopy Volume Map of Protein NARROW LEAF 1. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0738 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34607_msk_1.map emd_34607_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
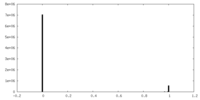
| 投影像・断面図 |
| ||||||||||||
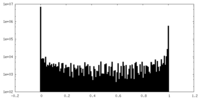
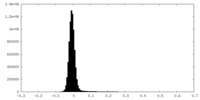
| 密度ヒストグラム |
-ハーフマップ: Electron Microscopy Half Map A of Protein NARROW LEAF 1.
| ファイル | emd_34607_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Electron Microscopy Half Map A of Protein NARROW LEAF 1. | ||||||||||||
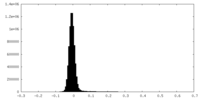
| 投影像・断面図 |
| ||||||||||||
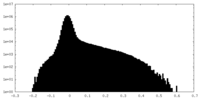
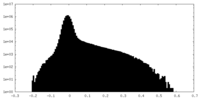
| 密度ヒストグラム |
-ハーフマップ: Electron Microscopy Half Map B of Protein NARROW LEAF 1.
| ファイル | emd_34607_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Electron Microscopy Half Map B of Protein NARROW LEAF 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NARROW LEAF 1-close from Japonica
| 全体 | 名称: NARROW LEAF 1-close from Japonica |
|---|---|
| 要素 |
|
-超分子 #1: NARROW LEAF 1-close from Japonica
| 超分子 | 名称: NARROW LEAF 1-close from Japonica / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 379.51 kDa/nm |
-分子 #1: Protein NARROW LEAF 1
| 分子 | 名称: Protein NARROW LEAF 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.317902 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKPSDDKAQL SGLAQSEESS LDVDHQSFPC SPSIQPVASG CTHTENSAAY FLWPTSNLQH CAAEGRANYF GNLQKGLLPR HPGRLPKGQ QANSLLDLMT IRAFHSKILR RFSLGTAVGF RIRKGDLTDI PAILVFVARK VHKKWLNPAQ CLPAILEGPG G VWCDVDVV ...文字列: MKPSDDKAQL SGLAQSEESS LDVDHQSFPC SPSIQPVASG CTHTENSAAY FLWPTSNLQH CAAEGRANYF GNLQKGLLPR HPGRLPKGQ QANSLLDLMT IRAFHSKILR RFSLGTAVGF RIRKGDLTDI PAILVFVARK VHKKWLNPAQ CLPAILEGPG G VWCDVDVV EFSYYGAPAQ TPKEQMFSEL VDKLCGSDEC IGSGSQVASH ETFGTLGAIV KRRTGNKQVG FLTNHHVAVD LD YPNQKMF HPLPPNLGPG VYLGAVERAT SFITDDVWYG IYAGTNPETF VRADGAFIPF ADDFDISTVT TVVRGVGDIG DVK VIDLQC PLNSLIGRQV CKVGRSSGHT TGTVMAYALE YNDEKGICFF TDILVVGENR QTFDLEGDSG SLIILTSQDG EKPR PIGII WGGTANRGRL KLTSDHGPEN WTSGVDLGRL LDRLELDIII TNESLQDAVQ QQRFALVAAV TSAVGESSGV PVAIP EEKI EEIFEPLGIQ IQQLPRHDVA ASGTEGEEAS NTVVNVEEHQ FISNFVGMSP VRDDQDAPRS ITNLNNPSEE ELAMSL HLG DREPKRLRSD SGSSLDLEK UniProtKB: Protein NARROW LEAF 1 |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 詳細: The grid was coated with gold prior to use |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: Vitrification carried out in Ethane. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 3770 / 平均露光時間: 0.972 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 22500 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial local fitting was done using ChimeraX |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 153.3 |
| 得られたモデル |  PDB-8has: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X