+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Echo 18 at pH5.5 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Echo 18 / pH5.5 / VIRUS | |||||||||
| 生物種 |  Echovirus E18 (ウイルス) Echovirus E18 (ウイルス) | |||||||||
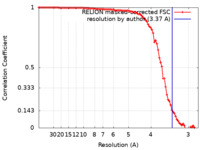
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.37 Å | |||||||||
 データ登録者 データ登録者 | Liu CC / Qu X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2022 ジャーナル: mBio / 年: 2022タイトル: Human FcRn Is a Two-in-One Attachment-Uncoating Receptor for Echovirus 18. 著者: Xiangpeng Chen / Xiao Qu / Congcong Liu / Yong Zhang / Guigen Zhang / Pu Han / Yali Duan / Qi Li / Liang Wang / Wenjing Ruan / Peiyi Wang / Wensheng Wei / George F Gao / Xin Zhao / Zhengde Xie /  要旨: Virus-receptor interactions determine viral host range and tissue tropism. CD55 and human neonatal Fc receptor (FcRn) were found to be the binding and uncoating receptors for some of the echovirus- ...Virus-receptor interactions determine viral host range and tissue tropism. CD55 and human neonatal Fc receptor (FcRn) were found to be the binding and uncoating receptors for some of the echovirus-related enterovirus species B serotypes in our previous study. Echovirus 18 (E18), as a member of enterovirus species B, is a significant causative agent of aseptic meningitis and viral encephalitis in children. However, it does not use CD55 as a critical host factor. We conducted CRISPR/Cas9 knockout screening to determine the receptors and entry mechanisms and identified FcRn working as a dual-function receptor for E18. Knockout of and , which encode the two subunits of FcRn, prevented infection by E18 and other echoviruses in the same physiological cluster. We then elucidated the underlying molecular mechanism of receptor recognition by E18 using cryogenic electron microscopy. The binding of the FCGRT subunit to the canyon region rotates the residues around the pocket, triggering the release of the pocket factor as observed for other enterovirus species B members. E18 is a member of enterovirus species B. As one of the most common enterovirus serotypes in nonpolio enterovirus detection, it easily infects children and causes various clinical symptoms. Aseptic meningitis and viral encephalitis are the most commonly reported syndromes associated with E18. No effective antiviral drugs or approved vaccines are available. Previous studies showed that CD55 and FcRn were the binding and uncoating receptors for some echoviruses. However, we found that CD55 is not the critical host factor for E18. Thus, we want to determine the receptors and elucidate the entry mechanism of E18. Our findings reveal that FcRn is a two-in-one attachment-uncoating receptor for E18. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33502.map.gz emd_33502.map.gz | 32.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33502-v30.xml emd-33502-v30.xml emd-33502.xml emd-33502.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33502_fsc.xml emd_33502_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33502.png emd_33502.png | 165.8 KB | ||
| マスクデータ |  emd_33502_msk_1.map emd_33502_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-33502.cif.gz emd-33502.cif.gz | 6.4 KB | ||
| その他 |  emd_33502_half_map_1.map.gz emd_33502_half_map_1.map.gz emd_33502_half_map_2.map.gz emd_33502_half_map_2.map.gz | 46.1 MB 46.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33502 http://ftp.pdbj.org/pub/emdb/structures/EMD-33502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33502 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33502_validation.pdf.gz emd_33502_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33502_full_validation.pdf.gz emd_33502_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_33502_validation.xml.gz emd_33502_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_33502_validation.cif.gz emd_33502_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33502 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33502 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33502 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33502 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33502.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33502.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.41 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33502_msk_1.map emd_33502_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33502_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33502_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Echovirus E18
| 全体 | 名称:  Echovirus E18 (ウイルス) Echovirus E18 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Echovirus E18
| 超分子 | 名称: Echovirus E18 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 47506 / 生物種: Echovirus E18 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Echovirus E18 (ウイルス) Echovirus E18 (ウイルス) |
| 分子量 | 理論値: 31.308102 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GDNQDRTVAN TQPSGPSNSK EIPALTAVET GHTSQVDPSD TLQTRHVVNF HSRSESTVEN FMGRAACVFM DQYKLNGEET STDNFAVWT INVREMAQLR RKCELFTYMR FDIEMTMVIT SCQDQGTQLE QDMPVLTHQI MYVPPGGPIP AKVDSYEWQT S TNPSVFWT ...文字列: GDNQDRTVAN TQPSGPSNSK EIPALTAVET GHTSQVDPSD TLQTRHVVNF HSRSESTVEN FMGRAACVFM DQYKLNGEET STDNFAVWT INVREMAQLR RKCELFTYMR FDIEMTMVIT SCQDQGTQLE QDMPVLTHQI MYVPPGGPIP AKVDSYEWQT S TNPSVFWT EGNAPARMSI PFISVGNAYS LFYDGWSHFT QDGTYGYTTL NAMGKLFVRH VNKSSPHQIT STIRVYFKPK HI KAWVPRP PRLCPYINKG DVNFVVTEVT DARKSITDTP |
-分子 #2: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Echovirus E18 (ウイルス) Echovirus E18 (ウイルス) |
| 分子量 | 理論値: 28.805348 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SPSAEECGYS DRVRSMTLGN STITTQESAN VVVGYGEWPS YLSDKEATAE DQPTQPDVAT CRFYTLESVQ WEKSSPGWWW KFPEALKNM GLFGQNMHYH YLGRAGYTIH VQCNASKFHQ GCLLVVCVPE AEMGCADTST TFPATELTTE EEPHVFTSDS I TGKKVQAA ...文字列: SPSAEECGYS DRVRSMTLGN STITTQESAN VVVGYGEWPS YLSDKEATAE DQPTQPDVAT CRFYTLESVQ WEKSSPGWWW KFPEALKNM GLFGQNMHYH YLGRAGYTIH VQCNASKFHQ GCLLVVCVPE AEMGCADTST TFPATELTTE EEPHVFTSDS I TGKKVQAA VCNAGMGVGV GNLTIFPHQW INLRTNNSAT IVMPYINSVP MDNMFRHYNF TLMIIPFAPL NFNEGATAYV PV TVTIAPM YAEYNGLRLA STQ |
-分子 #3: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Echovirus E18 (ウイルス) Echovirus E18 (ウイルス) |
| 分子量 | 理論値: 26.134842 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GVPVLNTPGS TQFLTSDDFQ SPSAMPQFDE TPEMHIPGEV RNLMEMAEVD SVVPVNNITG KTKSMEAYQI AVGTGNTDKT KPIFSFQMD PGYSSVLKRT LLGEMLNYYA HWSGSVKLTF LFCGSAMATG KLLISYSPPG ASVPSSRKDA MLGTHIIWDI G LQSSCVLC ...文字列: GVPVLNTPGS TQFLTSDDFQ SPSAMPQFDE TPEMHIPGEV RNLMEMAEVD SVVPVNNITG KTKSMEAYQI AVGTGNTDKT KPIFSFQMD PGYSSVLKRT LLGEMLNYYA HWSGSVKLTF LFCGSAMATG KLLISYSPPG ASVPSSRKDA MLGTHIIWDI G LQSSCVLC VPWISQSHYR MVQQDPYTSA GYITCWYQTN IVVPPGAPTS CDVLCFASAC NDFSVRLLRD TPFMAQPGKL Q |
-分子 #4: VP4
| 分子 | 名称: VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Echovirus E18 (ウイルス) Echovirus E18 (ウイルス) |
| 分子量 | 理論値: 7.502346 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGAQVSTQKT GAHETSLNAK GNSIIHYTNI NFYKDAASSA SNRQELQQDP GKFTDPVKDL MVKTLPALN |
-分子 #5: PALMITIC ACID
| 分子 | 名称: PALMITIC ACID / タイプ: ligand / ID: 5 / コピー数: 1 / 式: PLM |
|---|---|
| 分子量 | 理論値: 256.424 Da |
| Chemical component information |  ChemComp-PLM: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 5.5 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 457 / 平均露光時間: 0.09 sec. / 平均電子線量: 1.025 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)