+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the T=3 lake sinai virus 1 (delta-N48) virus-like capsid at pH 6.5 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | virion component / Viral coat protein subunit / Capsid protein alpha 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Lake Sinai virus 1 (ウイルス) / Lake Sinai virus 1 (ウイルス) /   Lake Sinai virus 2 (ウイルス) Lake Sinai virus 2 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.51 Å | |||||||||
 データ登録者 データ登録者 | Chen NC / Wang CH / Chen CJ / Yoshimura M / Guan HH / Chuankhayan P / Lin CC | |||||||||
| 資金援助 |  台湾, 1件 台湾, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structures of honeybee-infecting Lake Sinai virus reveal domain functions and capsid assembly with dynamic motions. 著者: Nai-Chi Chen / Chun-Hsiung Wang / Masato Yoshimura / Yi-Qi Yeh / Hong-Hsiang Guan / Phimonphan Chuankhayan / Chien-Chih Lin / Pei-Ju Lin / Yen-Chieh Huang / Soichi Wakatsuki / Meng-Chiao Ho / Chun-Jung Chen /   要旨: Understanding the structural diversity of honeybee-infecting viruses is critical to maintain pollinator health and manage the spread of diseases in ecology and agriculture. We determine cryo-EM ...Understanding the structural diversity of honeybee-infecting viruses is critical to maintain pollinator health and manage the spread of diseases in ecology and agriculture. We determine cryo-EM structures of T = 4 and T = 3 capsids of virus-like particles (VLPs) of Lake Sinai virus (LSV) 2 and delta-N48 LSV1, belonging to tetraviruses, at resolutions of 2.3-2.6 Å in various pH environments. Structural analysis shows that the LSV2 capsid protein (CP) structural features, particularly the protruding domain and C-arm, differ from those of other tetraviruses. The anchor loop on the central β-barrel domain interacts with the neighboring subunit to stabilize homo-trimeric capsomeres during assembly. Delta-N48 LSV1 CP interacts with ssRNA via the rigid helix α1', α1'-α1 loop, β-barrel domain, and C-arm. Cryo-EM reconstructions, combined with X-ray crystallographic and small-angle scattering analyses, indicate that pH affects capsid conformations by regulating reversible dynamic particle motions and sizes of LSV2 VLPs. C-arms exist in all LSV2 and delta-N48 LSV1 VLPs across varied pH conditions, indicating that autoproteolysis cleavage is not required for LSV maturation. The observed linear domino-scaffold structures of various lengths, made up of trapezoid-shape capsomeres, provide a basis for icosahedral T = 4 and T = 3 architecture assemblies. These findings advance understanding of honeybee-infecting viruses that can cause Colony Collapse Disorder. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33373.map.gz emd_33373.map.gz | 473.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33373-v30.xml emd-33373-v30.xml emd-33373.xml emd-33373.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33373.png emd_33373.png | 138.5 KB | ||
| Filedesc metadata |  emd-33373.cif.gz emd-33373.cif.gz | 5.8 KB | ||
| その他 |  emd_33373_half_map_1.map.gz emd_33373_half_map_1.map.gz emd_33373_half_map_2.map.gz emd_33373_half_map_2.map.gz | 370.7 MB 370.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33373 http://ftp.pdbj.org/pub/emdb/structures/EMD-33373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33373 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33373_validation.pdf.gz emd_33373_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33373_full_validation.pdf.gz emd_33373_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_33373_validation.xml.gz emd_33373_validation.xml.gz | 19.2 KB | 表示 | |
| CIF形式データ |  emd_33373_validation.cif.gz emd_33373_validation.cif.gz | 22.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33373 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33373 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33373 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33373 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33373.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33373.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0537 Å | ||||||||||||||||||||||||||||||||||||
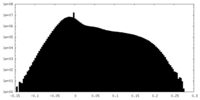
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_33373_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
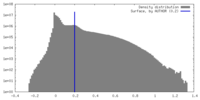
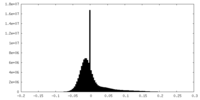
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33373_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
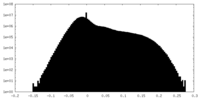
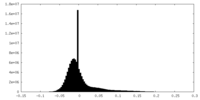
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lake Sinai virus 2
| 全体 | 名称:  Lake Sinai virus 2 (ウイルス) Lake Sinai virus 2 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Lake Sinai virus 2
| 超分子 | 名称: Lake Sinai virus 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 / NCBI-ID: 1041831 / 生物種: Lake Sinai virus 2 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|
-分子 #1: Capsid protein alpha
| 分子 | 名称: Capsid protein alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lake Sinai virus 1 (ウイルス) Lake Sinai virus 1 (ウイルス) |
| 分子量 | 理論値: 62.979355 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNSICSLYCL FRRVMPYSSE ILSSTVYRPP FLATSGLFFT RDHYKFIIMN QQQINPAQRS LRPRAQSAPS RSARRRRNRR RRNPSTPAG TVALQPSRIT RRVVSNLARR PLTITTAGLA WLRQYLNPMG PSTSSVSGFP DGSAVTTCIA DYTNTFNISF P PREAIYCT ...文字列: MNSICSLYCL FRRVMPYSSE ILSSTVYRPP FLATSGLFFT RDHYKFIIMN QQQINPAQRS LRPRAQSAPS RSARRRRNRR RRNPSTPAG TVALQPSRIT RRVVSNLARR PLTITTAGLA WLRQYLNPMG PSTSSVSGFP DGSAVTTCIA DYTNTFNISF P PREAIYCT GSNSDEKPVM LDAATYAKID AWTKSDITLC ILALPMLRNV VMIRLYASTP TAFTLSEGVP NFVQRFPNWS AF TTEGKVL NNGDSPGYIQ SFVYLPNVDK HLSAARGFRL LSRGLTGIYT APSLETQGFV TACQYLAEGA LQTQTVGNDF VQS VEVNAD KTVKNVNGKR LHYSGPPKFV FPLEGDNCAP SSLVETYHQA YQARAVDGFY MPILSSSRDN PFISPKPQPI AVFN RWYYR GCLDPVPASK VADGPSQYYY DLNVADDVAP LYNTGVVWME GISSKFSLKL KTRTVIQYIP TSGSVLANFT RHEPT YDQV ALDAADRIRN MMPHAYPAAY NDWGWLGDLL DSTLSMLPGI GTAYRFAKPL IKPAWNWLGG KVSDFFGNPV SRDGDI YFD AK UniProtKB: Capsid protein alpha |
-分子 #2: RNA (5'-R(P*UP*G)-3')
| 分子 | 名称: RNA (5'-R(P*UP*G)-3') / タイプ: rna / ID: 2 / コピー数: 3 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 606.414 Da |
| 配列 | 文字列: UG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 46.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.72 µm / 最小 デフォーカス(公称値): 0.25 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)