+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of R-loop monoclonal antibody S9.6 in recognizing RNA:DNA hybrids | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Li Q / Lin C / Luo Z / Li H / Li X / Sun Q | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Genet Genomics / 年: 2022 ジャーナル: J Genet Genomics / 年: 2022タイトル: Cryo-EM structure of R-loop monoclonal antibody S9.6 in recognizing RNA:DNA hybrids. 著者: Qin Li / Chao Lin / Zhipu Luo / Haitao Li / Xueming Li / Qianwen Sun /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33286.map.gz emd_33286.map.gz | 20.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33286-v30.xml emd-33286-v30.xml emd-33286.xml emd-33286.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33286.png emd_33286.png | 30.5 KB | ||
| その他 |  emd_33286_half_map_1.map.gz emd_33286_half_map_1.map.gz emd_33286_half_map_2.map.gz emd_33286_half_map_2.map.gz | 20.6 MB 20.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33286 http://ftp.pdbj.org/pub/emdb/structures/EMD-33286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33286 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33286_validation.pdf.gz emd_33286_validation.pdf.gz | 625 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33286_full_validation.pdf.gz emd_33286_full_validation.pdf.gz | 624.6 KB | 表示 | |
| XML形式データ |  emd_33286_validation.xml.gz emd_33286_validation.xml.gz | 10.1 KB | 表示 | |
| CIF形式データ |  emd_33286_validation.cif.gz emd_33286_validation.cif.gz | 11.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33286 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33286 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33286.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33286.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.091 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33286_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
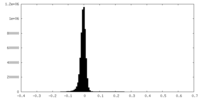
| 投影像・断面図 |
| ||||||||||||
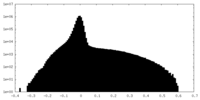
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33286_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
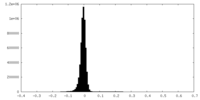
| 投影像・断面図 |
| ||||||||||||
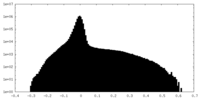
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM Structure of R-loop monoclonal antibody S9.6 in recognizi...
| 全体 | 名称: Cryo-EM Structure of R-loop monoclonal antibody S9.6 in recognizing RNA:DNA hybrids |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM Structure of R-loop monoclonal antibody S9.6 in recognizi...
| 超分子 | 名称: Cryo-EM Structure of R-loop monoclonal antibody S9.6 in recognizing RNA:DNA hybrids タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: S9.6 Fab HC
| 分子 | 名称: S9.6 Fab HC / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.85675 KDa |
| 配列 | 文字列: EVQLQQSGPE LVKPGASVKM SCKASGYTFT SYVMHWVKQK PGQGLEWIGF INLYNDGTKY NEKFKGKATL TSDKSSSTAY MELSSLTSK DSAVYYCARD YYGSRWFDYW GQGTTLTVSS AKTTAPSVYP LAPVCGDTTG SSVTLGCLVK GYFPEPVTLT W NSGSLSSG ...文字列: EVQLQQSGPE LVKPGASVKM SCKASGYTFT SYVMHWVKQK PGQGLEWIGF INLYNDGTKY NEKFKGKATL TSDKSSSTAY MELSSLTSK DSAVYYCARD YYGSRWFDYW GQGTTLTVSS AKTTAPSVYP LAPVCGDTTG SSVTLGCLVK GYFPEPVTLT W NSGSLSSG VHTFPAVLQS DLYTLSSSVT VTSSTWPSQS ITCNVAHPAS STKVDKKIEP RV |
-分子 #2: S9.6 Fab LC
| 分子 | 名称: S9.6 Fab LC / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.182803 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DVLMTQTPLS LPVSLGDQAS ISCRSSQSIV HSNGNTYLEW YLQKPGQSPK LLIYKVSNRF SGVPDRFSGS GSGTDFTLKI SRVEAEDLG VYYCFQGSHV PYTFGGGTKL EIKRADAAPT VSIFPPSSEQ LTSGGASVVC FLNNFYPKDI NVKWKIDGSE R QNGVLNSW ...文字列: DVLMTQTPLS LPVSLGDQAS ISCRSSQSIV HSNGNTYLEW YLQKPGQSPK LLIYKVSNRF SGVPDRFSGS GSGTDFTLKI SRVEAEDLG VYYCFQGSHV PYTFGGGTKL EIKRADAAPT VSIFPPSSEQ LTSGGASVVC FLNNFYPKDI NVKWKIDGSE R QNGVLNSW TDQDSKDSTY SMSSTLTLTK DEYERHNSYT CEATHKTSTS PIVKSFNRNE C |
-分子 #3: DNA
| 分子 | 名称: DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12.142727 KDa |
| 配列 | 文字列: (DT)(DC)(DT)(DG)(DT)(DT)(DC)(DT)(DT)(DC) (DG)(DT)(DG)(DT)(DT)(DC)(DC)(DT)(DC)(DG) (DT)(DC)(DT)(DG)(DT)(DT)(DC)(DT)(DT) (DC)(DG)(DT)(DG)(DT)(DT)(DC)(DC)(DT)(DC) (DG) |
-分子 #4: RNA
| 分子 | 名称: RNA / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.12308 KDa |
| 配列 | 文字列: CGAGGAACAC GAAGAACAGA CGAGGAACAC GAAGAACAGA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 64305 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X