+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
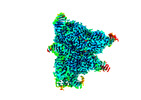
| タイトル | Structure of the OgeuIscB-omega RNA-target DNA complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas9 / RNP / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 生物種 | metagenome (メタゲノム) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.55 Å | |||||||||
 データ登録者 データ登録者 | Kato K / Okazaki O / Isayama Y / Ishikawa J / Nishizawa T / Nishimasu H | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of the IscB-ωRNA ribonucleoprotein complex, the likely ancestor of CRISPR-Cas9. 著者: Kazuki Kato / Sae Okazaki / Soumya Kannan / Han Altae-Tran / F Esra Demircioglu / Yukari Isayama / Junichiro Ishikawa / Masahiro Fukuda / Rhiannon K Macrae / Tomohiro Nishizawa / Kira S ...著者: Kazuki Kato / Sae Okazaki / Soumya Kannan / Han Altae-Tran / F Esra Demircioglu / Yukari Isayama / Junichiro Ishikawa / Masahiro Fukuda / Rhiannon K Macrae / Tomohiro Nishizawa / Kira S Makarova / Eugene V Koonin / Feng Zhang / Hiroshi Nishimasu /   要旨: Transposon-encoded IscB family proteins are RNA-guided nucleases in the OMEGA (obligate mobile element-guided activity) system, and likely ancestors of the RNA-guided nuclease Cas9 in the type II ...Transposon-encoded IscB family proteins are RNA-guided nucleases in the OMEGA (obligate mobile element-guided activity) system, and likely ancestors of the RNA-guided nuclease Cas9 in the type II CRISPR-Cas adaptive immune system. IscB associates with its cognate ωRNA to form a ribonucleoprotein complex that cleaves double-stranded DNA targets complementary to an ωRNA guide segment. Although IscB shares the RuvC and HNH endonuclease domains with Cas9, it is much smaller than Cas9, mainly due to the lack of the α-helical nucleic-acid recognition lobe. Here, we report the cryo-electron microscopy structure of an IscB protein from the human gut metagenome (OgeuIscB) in complex with its cognate ωRNA and a target DNA, at 2.6-Å resolution. This high-resolution structure reveals the detailed architecture of the IscB-ωRNA ribonucleoprotein complex, and shows how the small IscB protein assembles with the ωRNA and mediates RNA-guided DNA cleavage. The large ωRNA scaffold structurally and functionally compensates for the recognition lobe of Cas9, and participates in the recognition of the guide RNA-target DNA heteroduplex. These findings provide insights into the mechanism of the programmable DNA cleavage by the IscB-ωRNA complex and the evolution of the type II CRISPR-Cas9 effector complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33198.map.gz emd_33198.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33198-v30.xml emd-33198-v30.xml emd-33198.xml emd-33198.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33198.png emd_33198.png | 63.1 KB | ||
| Filedesc metadata |  emd-33198.cif.gz emd-33198.cif.gz | 6.3 KB | ||
| その他 |  emd_33198_half_map_1.map.gz emd_33198_half_map_1.map.gz emd_33198_half_map_2.map.gz emd_33198_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33198 http://ftp.pdbj.org/pub/emdb/structures/EMD-33198 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33198 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33198 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33198_validation.pdf.gz emd_33198_validation.pdf.gz | 793.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33198_full_validation.pdf.gz emd_33198_full_validation.pdf.gz | 792.9 KB | 表示 | |
| XML形式データ |  emd_33198_validation.xml.gz emd_33198_validation.xml.gz | 14.1 KB | 表示 | |
| CIF形式データ |  emd_33198_validation.cif.gz emd_33198_validation.cif.gz | 16.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33198 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33198 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33198 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33198 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33198.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33198.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33198_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
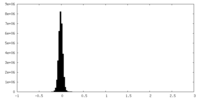
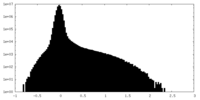
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33198_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
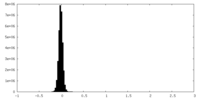
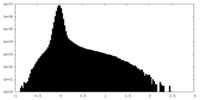
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of the IscB-omegaRNA ribonucleoprotein complex
| 全体 | 名称: Structure of the IscB-omegaRNA ribonucleoprotein complex |
|---|---|
| 要素 |
|
-超分子 #1: Structure of the IscB-omegaRNA ribonucleoprotein complex
| 超分子 | 名称: Structure of the IscB-omegaRNA ribonucleoprotein complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #4 |
|---|
-超分子 #2: IscB-omegaRNA
| 超分子 | 名称: IscB-omegaRNA / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|
-分子 #1: RNA (228-MER)
| 分子 | 名称: RNA (228-MER) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 73.640742 KDa |
| 配列 | 文字列: UGUGAGCGGA UAACAAUUCC CCGGCUCUUC CAACUUUAUG GUUGCGACCG UAGGUUGAAA GAGCACAGGC UGAGACAUUC GUAAGGCCG AAAGACCGGA CGCACCCUGG GAUUUCCCCA GUCCCCGGAA CUGCAUAGCG GAUGCCAGUU GAUGGAGCAA U CUAUCAGA ...文字列: UGUGAGCGGA UAACAAUUCC CCGGCUCUUC CAACUUUAUG GUUGCGACCG UAGGUUGAAA GAGCACAGGC UGAGACAUUC GUAAGGCCG AAAGACCGGA CGCACCCUGG GAUUUCCCCA GUCCCCGGAA CUGCAUAGCG GAUGCCAGUU GAUGGAGCAA U CUAUCAGA UAAGCCAGGG GGAACAAUCA CCUCUCUGUA UCAGAGAGAG UUUUACAAAA GGAGGAACGG |
-分子 #2: DNA (49-MER)
| 分子 | 名称: DNA (49-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.092702 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DT)(DG)(DG)(DT)(DT)(DT)(DT) (DC)(DT)(DT)(DC)(DG)(DG)(DG)(DG)(DA)(DA) (DT)(DT)(DG)(DT)(DT)(DA)(DT)(DC)(DC) (DG)(DC)(DT)(DC)(DA)(DC)(DA)(DA)(DT)(DT) (DC) (DC)(DT)(DT)(DA)(DG)(DA)(DA)(DA) (DA) |
-分子 #3: DNA (5'-D(P*GP*AP*AP*GP*AP*AP*AP*AP*CP*CP*AP*T)-3')
| 分子 | 名称: DNA (5'-D(P*GP*AP*AP*GP*AP*AP*AP*AP*CP*CP*AP*T)-3') / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 4.281832 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DG)(DA)(DA)(DA)(DA)(DC)(DC) (DA)(DT)(DT)(DC) |
-分子 #4: OgeuIscB
| 分子 | 名称: OgeuIscB / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 56.81968 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMAVVYVISK SGKPLMPTTR CGHVRILLKE GKARVVERKP FTIQLTYESA EETQPLVLGI DPGRTNIGMS VVTESGESVF NAQIETRNK DVPKLMKDRK QYRMAHRRLK RRCKRRRRAK AAGTAFEEGE KQRLLPGCFK PITCKSIRNK EARFNNRKRP V GWLTPTAN ...文字列: MMAVVYVISK SGKPLMPTTR CGHVRILLKE GKARVVERKP FTIQLTYESA EETQPLVLGI DPGRTNIGMS VVTESGESVF NAQIETRNK DVPKLMKDRK QYRMAHRRLK RRCKRRRRAK AAGTAFEEGE KQRLLPGCFK PITCKSIRNK EARFNNRKRP V GWLTPTAN HLLVTHLNVV KKVQKILPVA KVVLELNRFS FMAMNNPKVQ RWQYQRGPLY GKGSVEEAVS MQQDGHCLFC KH GIDHYHH VVPRRKNGSE TLENRVGLCE EHHRLVHTDK EWEANLASKK SGMNKKYHAL SVLNQIIPYL ADQLADMFPG NFC VTSGQD TYLFREEHGI PKDHYLDAYC IACSALTDAK KVSSPKGRPY MVHQFRRHDR QACHKANLNR SYYMGGKLVA TNRH KAMDQ KTDSLEEYRA AHSAADVSKL TVKHPSAQYK DMSRIMPGSI LVSGEGKLFT LSRSEGRNKG QVNYFVSTEG IKYWA RKCQ YLRNNGGLQI YV |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: LAURYL DIMETHYLAMINE-N-OXIDE
| 分子 | 名称: LAURYL DIMETHYLAMINE-N-OXIDE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: LDA |
|---|---|
| 分子量 | 理論値: 229.402 Da |
| Chemical component information |  ChemComp-LDA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 47.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.55 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 792405 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)