+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full length human CLC-2 channel in apo state | |||||||||
 マップデータ マップデータ | full lenth human CLC-2 channel in apo state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | homo-dimer / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of aldosterone biosynthetic process / cell differentiation involved in salivary gland development / regulation of membrane depolarization during action potential / volume-sensitive chloride channel activity / astrocyte end-foot / stabilization of membrane potential / acinar cell differentiation / cellular hypotonic response / regulation of resting membrane potential / voltage-gated chloride channel activity ...regulation of aldosterone biosynthetic process / cell differentiation involved in salivary gland development / regulation of membrane depolarization during action potential / volume-sensitive chloride channel activity / astrocyte end-foot / stabilization of membrane potential / acinar cell differentiation / cellular hypotonic response / regulation of resting membrane potential / voltage-gated chloride channel activity / axon initial segment / chloride channel regulator activity / dendritic spine membrane / chloride transport / phagocytosis, engulfment / positive regulation of oligodendrocyte differentiation / chloride channel complex / lung development / Stimuli-sensing channels / myelin sheath / retina development in camera-type eye / basolateral plasma membrane / perikaryon / postsynaptic membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
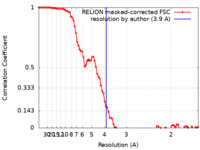
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Wang L | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structures of ClC-2 chloride channel reveal the blocking mechanism of its specific inhibitor AK-42 著者: Ma T / Wang L / Chai A / Liu C / Cui W / Yuan S / Wing Ngor Au S / Sun L / Zhang X / Zhang Z / Lu J / Gao Y / Wang P / Li Z / Liang Y / Vogel H / Wang YT / Wang D / Yan K / Zhang H | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33169.map.gz emd_33169.map.gz | 6.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33169-v30.xml emd-33169-v30.xml emd-33169.xml emd-33169.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33169_fsc.xml emd_33169_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33169.png emd_33169.png | 50.7 KB | ||
| Filedesc metadata |  emd-33169.cif.gz emd-33169.cif.gz | 6 KB | ||
| その他 |  emd_33169_half_map_1.map.gz emd_33169_half_map_1.map.gz emd_33169_half_map_2.map.gz emd_33169_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33169 http://ftp.pdbj.org/pub/emdb/structures/EMD-33169 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33169 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33169 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33169_validation.pdf.gz emd_33169_validation.pdf.gz | 793.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33169_full_validation.pdf.gz emd_33169_full_validation.pdf.gz | 793.4 KB | 表示 | |
| XML形式データ |  emd_33169_validation.xml.gz emd_33169_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_33169_validation.cif.gz emd_33169_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33169 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33169 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33169 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33169 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xf5MC  7xjaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33169.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33169.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full lenth human CLC-2 channel in apo state | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.829 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_33169_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
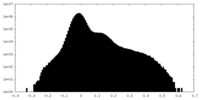
| 投影像・断面図 |
| ||||||||||||
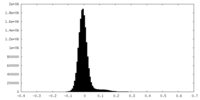
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33169_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
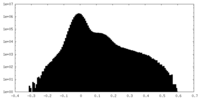
| 投影像・断面図 |
| ||||||||||||
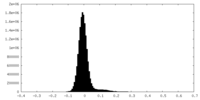
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Full length human CLC-2 homo dimer
| 全体 | 名称: Full length human CLC-2 homo dimer |
|---|---|
| 要素 |
|
-超分子 #1: Full length human CLC-2 homo dimer
| 超分子 | 名称: Full length human CLC-2 homo dimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 180 kDa/nm |
-分子 #1: Chloride channel protein 2
| 分子 | 名称: Chloride channel protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 98.642352 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAAAAAEEGM EPRALQYEQT LMYGRYTQDL GAFAKEEAAR IRLGGPEPWK GPPSSRAAPE LLEYGRSRCA RCRVCSVRCH KFLVSRVGE DWIFLVLLGL LMALVSWVMD YAIAACLQAQ QWMSRGLNTS ILLQYLAWVT YPVVLITFSA GFTQILAPQA V GSGIPEMK ...文字列: MAAAAAEEGM EPRALQYEQT LMYGRYTQDL GAFAKEEAAR IRLGGPEPWK GPPSSRAAPE LLEYGRSRCA RCRVCSVRCH KFLVSRVGE DWIFLVLLGL LMALVSWVMD YAIAACLQAQ QWMSRGLNTS ILLQYLAWVT YPVVLITFSA GFTQILAPQA V GSGIPEMK TILRGVVLKE YLTLKTFIAK VIGLTCALGS GMPLGKEGPF VHIASMCAAL LSKFLSLFGG IYENESRNTE ML AAACAVG VGCCFAAPIG GVLFSIEVTS TFFAVRNYWR GFFAATFSAF IFRVLAVWNR DEETITALFK TRFRLDFPFD LQE LPAFAV IGIASGFGGA LFVYLNRKIV QVMRKQKTIN RFLMRKRLLF PALVTLLIST LTFPPGFGQF MAGQLSQKET LVTL FDNRT WVRQGLVEEL EPPSTSQAWN PPRANVFLTL VIFILMKFWM SALATTIPVP CGAFMPVFVI GAAFGRLVGE SMAAW FPDG IHTDSSTYRI VPGGYAVVGA AALAGAVTHT VSTAVIVFEL TGQIAHILPV MIAVILANAV AQSLQPSLYD SIIRIK KLP YLPELGWGRH QQYRVRVEDI MVRDVPHVAL SCTFRDLRLA LHRTKGRMLA LVESPESMIL LGSIERSQVV ALLGAQL SP ARRRQHMQER RATQTSPLSD QEGPPTPEAS VCFQVNTEDS AFPAARGETH KPLKPALKRG PSVTRNLGES PTGSAESA G IALRSLFCGS PPPEAASEKL ESCEKRKLKR VRISLASDAD LEGEMSPEEI LEWEEQQLDE PVNFSDCKID PAPFQLVER TSLHKTHTIF SLLGVDHAYV TSIGRLIGIV TLKELRKAIE GSVTAQGVKV RPPLASFRDS ATSSSDTETT EVHALWGPHS RHGLPREGS PSDSDDKCQ UniProtKB: Chloride channel protein 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 300 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7xf5: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)