+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure and mechanism of a mitochondrial AAA+ disaggregase CLPB | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CLPB / AAA-ATPase / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報granulocyte differentiation / RIG-I signaling pathway / ATP-dependent protein disaggregase activity / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / antiviral innate immune response / mitochondrial intermembrane space / cellular response to heat / ATP hydrolysis activity / mitochondrion / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Wu D / Liu Y / Dai Y / Wang G / Lu G / Chen Y / Li N / Lin J / Gao N | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Biol / 年: 2023 ジャーナル: PLoS Biol / 年: 2023タイトル: Comprehensive structural characterization of the human AAA+ disaggregase CLPB in the apo- and substrate-bound states reveals a unique mode of action driven by oligomerization. 著者: Damu Wu / Yan Liu / Yuhao Dai / Guopeng Wang / Guoliang Lu / Yan Chen / Ningning Li / Jinzhong Lin / Ning Gao /  要旨: The human AAA+ ATPase CLPB (SKD3) is a protein disaggregase in the mitochondrial intermembrane space (IMS) and functions to promote the solubilization of various mitochondrial proteins. Loss-of- ...The human AAA+ ATPase CLPB (SKD3) is a protein disaggregase in the mitochondrial intermembrane space (IMS) and functions to promote the solubilization of various mitochondrial proteins. Loss-of-function CLPB mutations are associated with a few human diseases with neutropenia and neurological disorders. Unlike canonical AAA+ proteins, CLPB contains a unique ankyrin repeat domain (ANK) at its N-terminus. How CLPB functions as a disaggregase and the role of its ANK domain are currently unclear. Herein, we report a comprehensive structural characterization of human CLPB in both the apo- and substrate-bound states. CLPB assembles into homo-tetradecamers in apo-state and is remodeled into homo-dodecamers upon substrate binding. Conserved pore-loops (PLs) on the ATPase domains form a spiral staircase to grip and translocate the substrate in a step-size of 2 amino acid residues. The ANK domain is not only responsible for maintaining the higher-order assembly but also essential for the disaggregase activity. Interactome analysis suggests that the ANK domain may directly interact with a variety of mitochondrial substrates. These results reveal unique properties of CLPB as a general disaggregase in mitochondria and highlight its potential as a target for the treatment of various mitochondria-related diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33104.map.gz emd_33104.map.gz | 59.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33104-v30.xml emd-33104-v30.xml emd-33104.xml emd-33104.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33104.png emd_33104.png | 55.7 KB | ||
| Filedesc metadata |  emd-33104.cif.gz emd-33104.cif.gz | 5.9 KB | ||
| その他 |  emd_33104_half_map_1.map.gz emd_33104_half_map_1.map.gz emd_33104_half_map_2.map.gz emd_33104_half_map_2.map.gz | 52.1 MB 52.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33104 http://ftp.pdbj.org/pub/emdb/structures/EMD-33104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33104 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xbkMC  7xc5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33104.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33104.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_33104_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
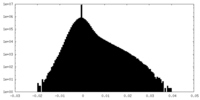
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33104_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CLPB
| 全体 | 名称: CLPB |
|---|---|
| 要素 |
|
-超分子 #1: CLPB
| 超分子 | 名称: CLPB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Isoform 2 of Caseinolytic peptidase B protein homolog
| 分子 | 名称: Isoform 2 of Caseinolytic peptidase B protein homolog タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 75.566336 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLGSLVLRRK ALAPRLLLRL LRSPTLRGHG GASGRNVTTG SLGEPQWLRV ATGGRPGTSP ALFSGRGAAT GGRQGGRFDT KCLAAATWG RLPGPEETLP GQDSWNGVPS RAGLGMCALA AALVVHCYSK SPSNKDAALL EAARANNMQE VSRLLSEGAD V NAKHRLGW ...文字列: MLGSLVLRRK ALAPRLLLRL LRSPTLRGHG GASGRNVTTG SLGEPQWLRV ATGGRPGTSP ALFSGRGAAT GGRQGGRFDT KCLAAATWG RLPGPEETLP GQDSWNGVPS RAGLGMCALA AALVVHCYSK SPSNKDAALL EAARANNMQE VSRLLSEGAD V NAKHRLGW TALMVAAINR NNSVVQVLLA AGADPNLGDD FSSVYKTAKE QGIHSLEVLI TREDDFNNRL NNRASFKGCT AL HYAVLAD DYRTVKELLD GGANPLQRNE MGHTPLDYAR EGEVMKLLRT SEAKYQEKQR KREAEERRRF PLEQRLKEHI IGQ ESAIAT VGAAIRRKEN GWYDEEHPLV FLFLGSSGIG KTELAKQTAK YMHKDAKKGF IRLDMSEFQE RHEVAKFIGS PPGY VGHEE GGQLTKKLKQ CPNAVVLFDQ VDKAHPDVLT IMLQLFDEGR LTDGKGKTID CKDAIFIMTS NVASDEIAQH ALQLR QEAL EMSRNRIAEN LGDVQISDKI TISKNFKENV IRPILKAHFR RDEFLGRINE IVYFLPFCHS ELIQLVNKEL NFWAKR AKQ RHNITLLWDR EVADVLVDGY NVHYGARSIK HEVERRVVNQ LAAAYEQDLL PGGCTLRITV EDSDKQLLKS PELPSPQ AE KRLPKLRLEI IDKDSKTRRL DIRAPLHPEK VCNTI UniProtKB: Mitochondrial disaggregase |
-分子 #2: Unknown peptide
| 分子 | 名称: Unknown peptide / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.464797 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 8 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetare |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: DARK FIELD 最大 デフォーカス(公称値): 1.4000000000000001 µm 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 45907 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X