+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of engineered hepatitis C virus E1E2 ectodomain in complex with antibodies AR4A, HEPC74, and IGH520 | |||||||||
 マップデータ マップデータ | Primary map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HCV / E1E2 / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell mitochondrial membrane / host cell lipid droplet / host cell mitochondrion / lipid droplet / viral nucleocapsid / clathrin-dependent endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / ribonucleoprotein complex / virus-mediated perturbation of host defense response / viral envelope ...host cell mitochondrial membrane / host cell lipid droplet / host cell mitochondrion / lipid droplet / viral nucleocapsid / clathrin-dependent endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / ribonucleoprotein complex / virus-mediated perturbation of host defense response / viral envelope / host cell nucleus / structural molecule activity / virion membrane / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Hepacivirus C (ウイルス) / synthetic construct (人工物) / Hepacivirus C (ウイルス) / synthetic construct (人工物) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.65 Å | |||||||||
 データ登録者 データ登録者 | Metcalf MC / Ofek G | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of engineered hepatitis C virus E1E2 ectodomain in complex with neutralizing antibodies. 著者: Matthew C Metcalf / Benjamin M Janus / Rui Yin / Ruixue Wang / Johnathan D Guest / Edwin Pozharski / Mansun Law / Roy A Mariuzza / Eric A Toth / Brian G Pierce / Thomas R Fuerst / Gilad Ofek /  要旨: Hepatitis C virus (HCV) is a major global health burden as the leading causative agent of chronic liver disease and hepatocellular carcinoma. While the main antigenic target for HCV-neutralizing ...Hepatitis C virus (HCV) is a major global health burden as the leading causative agent of chronic liver disease and hepatocellular carcinoma. While the main antigenic target for HCV-neutralizing antibodies is the membrane-associated E1E2 surface glycoprotein, the development of effective vaccines has been hindered by complications in the biochemical preparation of soluble E1E2 ectodomains. Here, we present a cryo-EM structure of an engineered, secreted E1E2 ectodomain of genotype 1b in complex with neutralizing antibodies AR4A, HEPC74, and IGH520. Structural characterization of the E1 subunit and C-terminal regions of E2 reveal an overall architecture of E1E2 that concurs with that observed for non-engineered full-length E1E2. Analysis of the AR4A epitope within a region of E2 that bridges between the E2 core and E1 defines the structural basis for its broad neutralization. Our study presents the structure of an E1E2 complex liberated from membrane via a designed scaffold, one that maintains all essential structural features of native E1E2. The study advances the understanding of the E1E2 heterodimer structure, crucial for the rational design of secreted E1E2 antigens in vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29419.map.gz emd_29419.map.gz | 167.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29419-v30.xml emd-29419-v30.xml emd-29419.xml emd-29419.xml | 33.5 KB 33.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29419.png emd_29419.png | 46.3 KB | ||
| その他 |  emd_29419_additional_1.map.gz emd_29419_additional_1.map.gz emd_29419_additional_2.map.gz emd_29419_additional_2.map.gz emd_29419_additional_3.map.gz emd_29419_additional_3.map.gz emd_29419_additional_4.map.gz emd_29419_additional_4.map.gz emd_29419_additional_5.map.gz emd_29419_additional_5.map.gz emd_29419_additional_6.map.gz emd_29419_additional_6.map.gz emd_29419_half_map_1.map.gz emd_29419_half_map_1.map.gz emd_29419_half_map_2.map.gz emd_29419_half_map_2.map.gz | 164.9 MB 167.8 MB 164.9 MB 88.7 MB 164.8 MB 88.7 MB 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29419 http://ftp.pdbj.org/pub/emdb/structures/EMD-29419 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29419 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29419 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29419_validation.pdf.gz emd_29419_validation.pdf.gz | 907 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29419_full_validation.pdf.gz emd_29419_full_validation.pdf.gz | 906.6 KB | 表示 | |
| XML形式データ |  emd_29419_validation.xml.gz emd_29419_validation.xml.gz | 14.7 KB | 表示 | |
| CIF形式データ |  emd_29419_validation.cif.gz emd_29419_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29419 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29419 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29419 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29419 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8fsjMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29419.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29419.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.889 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Half map A for local refinement map
| ファイル | emd_29419_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
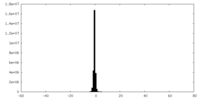
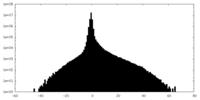
| 注釈 | Half map A for local refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
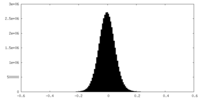
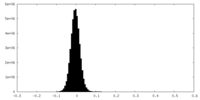
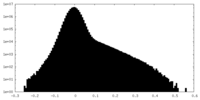
| 密度ヒストグラム |
-追加マップ: Sharpened local refinement map
| ファイル | emd_29419_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened local refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
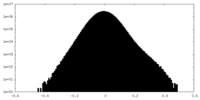
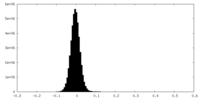
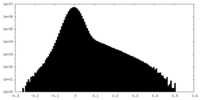
| 密度ヒストグラム |
-追加マップ: Half map B for local refinement map
| ファイル | emd_29419_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
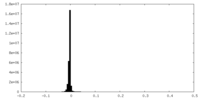
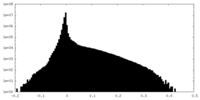
| 注釈 | Half map B for local refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
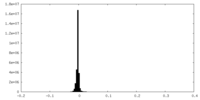
| 密度ヒストグラム |
-追加マップ: Non-sharpened local refinement map
| ファイル | emd_29419_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-sharpened local refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
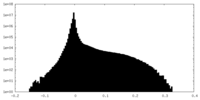
| 密度ヒストグラム |
-追加マップ: Merged primary and local refinement map
| ファイル | emd_29419_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Merged primary and local refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Non-sharpened primary map
| ファイル | emd_29419_additional_6.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-sharpened primary map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B for primary map
| ファイル | emd_29419_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B for primary map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A for primary map
| ファイル | emd_29419_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A for primary map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HCV E1E2 ectodomain in complex with AR4A, HEPC74, and IGH520
| 全体 | 名称: HCV E1E2 ectodomain in complex with AR4A, HEPC74, and IGH520 |
|---|---|
| 要素 |
|
-超分子 #1: HCV E1E2 ectodomain in complex with AR4A, HEPC74, and IGH520
| 超分子 | 名称: HCV E1E2 ectodomain in complex with AR4A, HEPC74, and IGH520 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Hepacivirus C (ウイルス) Hepacivirus C (ウイルス) |
| 分子量 | 理論値: 250 KDa |
-分子 #1: HCV E2 ectodomain, SYNZIP2 scaffold fusion
| 分子 | 名称: HCV E2 ectodomain, SYNZIP2 scaffold fusion / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 42.975371 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: STHVTGGTAS HTTRHFASLF SSGASQRVQL INTNGSWHIN RTALNCNDSL HTGFLAALFY THKFNASGCP ERMAHCRPID EFAQGWGPI TYAEGHGSDQ RPYCWHYAPR QCGTIPASQV CGPVYCFTPS PVVVGTTDRF GAPTYTWGEN ETDVLILNNT R PPQGNWFG ...文字列: STHVTGGTAS HTTRHFASLF SSGASQRVQL INTNGSWHIN RTALNCNDSL HTGFLAALFY THKFNASGCP ERMAHCRPID EFAQGWGPI TYAEGHGSDQ RPYCWHYAPR QCGTIPASQV CGPVYCFTPS PVVVGTTDRF GAPTYTWGEN ETDVLILNNT R PPQGNWFG CTWMNSTGFT KTCGGPPCNI GGVGNNTLTC PTDCFRKHPE ATYTKCGSGP WLTPRCLVDY PYRLWHYPCT VN FTIFKVR MYVGGVEHRL NAACNWTRGE RCDLQDRDRS ELSPLLLSTT EWQILPCSFT TLPALSTGLI HLHQNIVDVQ YLY GIGSAV VSFAIPGGLV AQLENEVASL ENENETLKKK NLHKKDLIAY LEKEIANLRK KIEHHHHHH UniProtKB: Genome polyprotein |
-分子 #2: HEPC74 heavy chain
| 分子 | 名称: HEPC74 heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.644807 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGAE VKKPGSSVKV SCTTSGGTYI NYAISWVRQA PGQGLEWVGG MSPISNTPKY AQKFQGRVTI TADESTSTTY MELSSLRPE DTAVYYCARD LLKYCGGGNC HSLLVDPWGQ GTLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW ...文字列: QVQLVQSGAE VKKPGSSVKV SCTTSGGTYI NYAISWVRQA PGQGLEWVGG MSPISNTPKY AQKFQGRVTI TADESTSTTY MELSSLRPE DTAVYYCARD LLKYCGGGNC HSLLVDPWGQ GTLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW NSGALTSGVH TFPAVLQSSG LYSLSSVVTV PSSSLGTQTY ICNVNHKPSN TKVDKRVEPK SCDKTAGWSH PQ FEKGGGS GGGSGGSSAW SHPQFEK |
-分子 #3: HEPC74 light chain
| 分子 | 名称: HEPC74 light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.470111 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIVMTQSPST LSASVGDRVT ISCRASQSIS SWLAWYQQKP GRAPKLLIYK ASSLETGVPS RFSGSGSGTE FTLTISSLQP DDFATYYCQ HYNTYLFTFG PGTKVDLKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIVMTQSPST LSASVGDRVT ISCRASQSIS SWLAWYQQKP GRAPKLLIYK ASSLETGVPS RFSGSGSGTE FTLTISSLQP DDFATYYCQ HYNTYLFTFG PGTKVDLKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #4: HCV E1 ectodomain, SYNZIP1 scaffold fusion
| 分子 | 名称: HCV E1 ectodomain, SYNZIP1 scaffold fusion / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 22.951379 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: YEVRNASGLY HVTNDCSNAS IVYETTDMIM HTPGCVPCVR EDNSSRCWVA LTPTLAARNA SVPTVAIRRH VDLLVGAAAF CSAMYVGDL CGSVFLVSQL FTFSPRRHET VQDCNCSIYP GHVSGHRMAW DMMMNWSPTA ALVVSQLLRI PQAVVDMVAP G GRNAYLRK ...文字列: YEVRNASGLY HVTNDCSNAS IVYETTDMIM HTPGCVPCVR EDNSSRCWVA LTPTLAARNA SVPTVAIRRH VDLLVGAAAF CSAMYVGDL CGSVFLVSQL FTFSPRRHET VQDCNCSIYP GHVSGHRMAW DMMMNWSPTA ALVVSQLLRI PQAVVDMVAP G GRNAYLRK KIARLKKDNL QLERDEQNLE KIIANLRDEI ARLENEVA UniProtKB: Genome polyprotein |
-分子 #5: AR4A light chain
| 分子 | 名称: AR4A light chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.743781 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGVPTQVLGL LLLWLTDARC EIELTLTQSP GTLSLSPGER ATLSCRASQS VSNNYLAWYQ QKPGQAPRLL IYGASSRATG IPDRFSGSG SGTGFTLIIS RLEPEDFAVY YCQQYGSSSI TFGQGTRLEI KRTVAAPSVF IFPPSDEQLK SGTASVVCLL N NFYREAKV ...文字列: MGVPTQVLGL LLLWLTDARC EIELTLTQSP GTLSLSPGER ATLSCRASQS VSNNYLAWYQ QKPGQAPRLL IYGASSRATG IPDRFSGSG SGTGFTLIIS RLEPEDFAVY YCQQYGSSSI TFGQGTRLEI KRTVAAPSVF IFPPSDEQLK SGTASVVCLL N NFYREAKV QWKVDNALQS GNSQESVTEQ DSKDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #6: AR4A heavy chain
| 分子 | 名称: AR4A heavy chain / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31.862697 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MYRMQLLSCI ALSLALVTNS EVQLLEQSGP EVKKPGDSLR ISCKMSGDSL VTTWIGWVRQ KPGQGLEWMG IINPGDSSTN IYPGDSATR YGPSFQGQVT ISIDKSTSTA YLQWNNVKAS DTGIYYCARH VPVPISGTFL WREREMHDFG YFDDWGQGTL V IVSSASTK ...文字列: MYRMQLLSCI ALSLALVTNS EVQLLEQSGP EVKKPGDSLR ISCKMSGDSL VTTWIGWVRQ KPGQGLEWMG IINPGDSSTN IYPGDSATR YGPSFQGQVT ISIDKSTSTA YLQWNNVKAS DTGIYYCARH VPVPISGTFL WREREMHDFG YFDDWGQGTL V IVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP EPVTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SL GTQTYIC NVNHKPSNTK VDKKVEPKSC AGWSHPQFEK GGGSGGGSGG SSAWSHPQFE K |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 47.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X