+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Duplex-G-quadruplex-duplex (DGD) class_1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | G-quadruplex / promoter / duplex / DNA | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.4 Å | |||||||||
 データ登録者 データ登録者 | Monsen RC / Chua EYD / Hopkins JB / Chaires JB / Trent JO | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Structure of a 28.5 kDa duplex-embedded G-quadruplex system resolved to 7.4 Å resolution with cryo-EM. 著者: Robert C Monsen / Eugene Y D Chua / Jesse B Hopkins / Jonathan B Chaires / John O Trent /  要旨: Genomic regions with high guanine content can fold into non-B form DNA four-stranded structures known as G-quadruplexes (G4s). Extensive in vivo investigations have revealed that promoter G4s are ...Genomic regions with high guanine content can fold into non-B form DNA four-stranded structures known as G-quadruplexes (G4s). Extensive in vivo investigations have revealed that promoter G4s are transcriptional regulators. Little structural information exists for these G4s embedded within duplexes, their presumed genomic environment. Here, we report the 7.4 Å resolution structure and dynamics of a 28.5 kDa duplex-G4-duplex (DGD) model system using cryo-EM, molecular dynamics, and small-angle X-ray scattering (SAXS) studies. The DGD cryo-EM refined model features a 53° bend induced by a stacked duplex-G4 interaction at the 5' G-tetrad interface with a persistently unstacked 3' duplex. The surrogate complement poly dT loop preferably stacks onto the 3' G-tetrad interface resulting in occlusion of both 5' and 3' tetrad interfaces. Structural analysis shows that the DGD model is quantifiably more druggable than the monomeric G4 structure alone and represents a new structural drug target. Our results illustrate how the integration of cryo-EM, MD, and SAXS can reveal complementary detailed static and dynamic structural information on DNA G4 systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27726.map.gz emd_27726.map.gz | 32.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27726-v30.xml emd-27726-v30.xml emd-27726.xml emd-27726.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
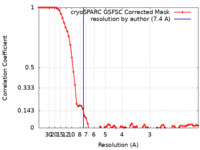
| FSC (解像度算出) |  emd_27726_fsc.xml emd_27726_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27726.png emd_27726.png | 25.2 KB | ||
| Filedesc metadata |  emd-27726.cif.gz emd-27726.cif.gz | 4.9 KB | ||
| その他 |  emd_27726_half_map_1.map.gz emd_27726_half_map_1.map.gz emd_27726_half_map_2.map.gz emd_27726_half_map_2.map.gz | 31.8 MB 31.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27726 http://ftp.pdbj.org/pub/emdb/structures/EMD-27726 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27726 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27726 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27726_validation.pdf.gz emd_27726_validation.pdf.gz | 652 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27726_full_validation.pdf.gz emd_27726_full_validation.pdf.gz | 651.6 KB | 表示 | |
| XML形式データ |  emd_27726_validation.xml.gz emd_27726_validation.xml.gz | 14.6 KB | 表示 | |
| CIF形式データ |  emd_27726_validation.cif.gz emd_27726_validation.cif.gz | 18.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27726 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27726 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27726 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27726 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27726.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27726.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.0252 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_27726_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
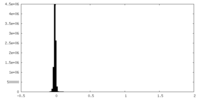
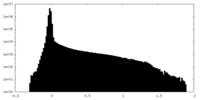
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27726_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
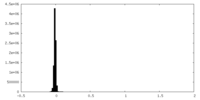
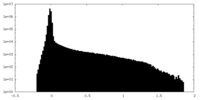
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : synthetic duplex-G-quadruplex-duplex construct mimicking a promoter G4
| 全体 | 名称: synthetic duplex-G-quadruplex-duplex construct mimicking a promoter G4 |
|---|---|
| 要素 |
|
-超分子 #1: synthetic duplex-G-quadruplex-duplex construct mimicking a promoter G4
| 超分子 | 名称: synthetic duplex-G-quadruplex-duplex construct mimicking a promoter G4 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Duplex-G-quadruplex-duplex (46-MER)
| 分子 | 名称: Duplex-G-quadruplex-duplex (46-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.086031 KDa |
| 配列 | 文字列: (DG)(DC)(DG)(DT)(DG)(DC)(DC)(DG)(DC)(DA) (DT)(DT)(DA)(DA)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DG)(DT)(DA) (DT) (DA)(DC)(DA)(DT)(DA)(DG) |
-分子 #2: Duplex-G-quadruplex-duplex (46-MER)
| 分子 | 名称: Duplex-G-quadruplex-duplex (46-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.407232 KDa |
| 配列 | 文字列: (DC)(DT)(DA)(DT)(DG)(DT)(DA)(DT)(DA)(DC) (DA)(DA)(DA)(DG)(DA)(DG)(DG)(DG)(DT)(DG) (DG)(DG)(DT)(DA)(DG)(DG)(DG)(DT)(DG) (DG)(DG)(DT)(DT)(DT)(DA)(DA)(DT)(DG)(DC) (DG) (DG)(DC)(DA)(DC)(DG)(DC) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 7 sec. / 前処理 - 雰囲気: OTHER | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | Sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 12175 / 平均露光時間: 0.4 sec. / 平均電子線量: 68.54 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: HELIUM |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 557.2 / 当てはまり具合の基準: map correlation |
|---|---|
| 得られたモデル |  PDB-8dut: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X