+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
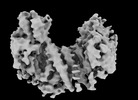
| タイトル | Structure of the PEAK3 pseudokinase homodimer | ||||||||||||
 マップデータ マップデータ | Reconstruction of the PEAK3 pseudokinase homodimer filtered to 4.9A and sharpened with -105 bFactor. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | complex / pseudokinase / kinase / adapter / SIGNALING PROTEIN | ||||||||||||
| 機能・相同性 | regulation of actin cytoskeleton organization / : / actin cytoskeleton / regulation of cell shape / protein kinase activity / focal adhesion / Protein PEAK3 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
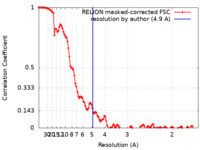
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.9 Å | ||||||||||||
 データ登録者 データ登録者 | Torosyan H / Paul M / Jura N / Verba KA | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural insights into regulation of the PEAK3 pseudokinase scaffold by 14-3-3. 著者: Hayarpi Torosyan / Michael D Paul / Antoine Forget / Megan Lo / Devan Diwanji / Krzysztof Pawłowski / Nevan J Krogan / Natalia Jura / Kliment A Verba /   要旨: PEAK pseudokinases are molecular scaffolds which dimerize to regulate cell migration, morphology, and proliferation, as well as cancer progression. The mechanistic role dimerization plays in PEAK ...PEAK pseudokinases are molecular scaffolds which dimerize to regulate cell migration, morphology, and proliferation, as well as cancer progression. The mechanistic role dimerization plays in PEAK scaffolding remains unclear, as there are no structures of PEAKs in complex with their interactors. Here, we report the cryo-EM structure of dimeric PEAK3 in complex with an endogenous 14-3-3 heterodimer. Our structure reveals an asymmetric binding mode between PEAK3 and 14-3-3 stabilized by one pseudokinase domain and the SHED domain of the PEAK3 dimer. The binding interface contains a canonical phosphosite-dependent primary interaction and a unique secondary interaction not observed in previous structures of 14-3-3/client complexes. Additionally, we show that PKD regulates PEAK3/14-3-3 binding, which when prevented leads to PEAK3 nuclear enrichment and distinct protein-protein interactions. Altogether, our data demonstrate that PEAK3 dimerization forms an unusual secondary interface for 14-3-3 binding, facilitating 14-3-3 regulation of PEAK3 localization and interactome diversity. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27684.map.gz emd_27684.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27684-v30.xml emd-27684-v30.xml emd-27684.xml emd-27684.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27684_fsc.xml emd_27684_fsc.xml | 11.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27684.png emd_27684.png | 60.1 KB | ||
| マスクデータ |  emd_27684_msk_1.map emd_27684_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27684.cif.gz emd-27684.cif.gz | 6.2 KB | ||
| その他 |  emd_27684_half_map_1.map.gz emd_27684_half_map_1.map.gz emd_27684_half_map_2.map.gz emd_27684_half_map_2.map.gz | 98.3 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27684 http://ftp.pdbj.org/pub/emdb/structures/EMD-27684 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27684 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27684 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27684_validation.pdf.gz emd_27684_validation.pdf.gz | 826.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27684_full_validation.pdf.gz emd_27684_full_validation.pdf.gz | 826.1 KB | 表示 | |
| XML形式データ |  emd_27684_validation.xml.gz emd_27684_validation.xml.gz | 18.5 KB | 表示 | |
| CIF形式データ |  emd_27684_validation.cif.gz emd_27684_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27684 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27684 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27684 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27684 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ds6MC  8dp5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27684.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27684.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the PEAK3 pseudokinase homodimer filtered to 4.9A and sharpened with -105 bFactor. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||
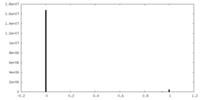
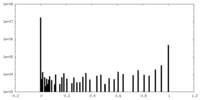
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27684_msk_1.map emd_27684_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
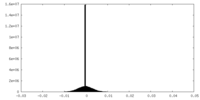
| 密度ヒストグラム |
-ハーフマップ: unfiltered, unsharpened half maps
| ファイル | emd_27684_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered, unsharpened half maps | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
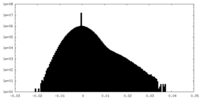
| 密度ヒストグラム |
-ハーフマップ: unfiltered, unsharpened half maps
| ファイル | emd_27684_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered, unsharpened half maps | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PEAK3 pseudokinase homodimer
| 全体 | 名称: PEAK3 pseudokinase homodimer |
|---|---|
| 要素 |
|
-超分子 #1: PEAK3 pseudokinase homodimer
| 超分子 | 名称: PEAK3 pseudokinase homodimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 104.58 KDa |
-分子 #1: Protein PEAK3
| 分子 | 名称: Protein PEAK3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 52.357031 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSSPEPPTEP PEPDNPTWST QPTYSNLGQI RAHLLPSKAC RLRTPGSLST NPEPLPPPLP KKILTRTQSL PTRRTLHPSS IQVQPPRRP FLGSHSVDKS QAAVGPACLP AELTFGPADA PLGLSLRDLH SPEAVHTALA ARQLQGLRTI YARLRARLMG G HPGPCHPG ...文字列: MSSPEPPTEP PEPDNPTWST QPTYSNLGQI RAHLLPSKAC RLRTPGSLST NPEPLPPPLP KKILTRTQSL PTRRTLHPSS IQVQPPRRP FLGSHSVDKS QAAVGPACLP AELTFGPADA PLGLSLRDLH SPEAVHTALA ARQLQGLRTI YARLRARLMG G HPGPCHPG HSFRLLDSSP CAESGDALYY RVVRAHEDAW HILVAKVPKP GADVPHPWGL ELQASLSPHF NLQGLCGLVP EG TLPGAPW RGAVALAAEV PERTVAQWLA EACTQPPEEF VWAVALLLLQ LSAALKFLEA WGAALVELRP ENLLLVAPRG CAT TGPPRL LLTDFGRVCL QPPGPPGSPG PHAPQLGSLL RALLSLAAPS TTPLAAGLEL LAAQLTRLRP SASRTRGALQ ALLW GPGPE LRGRGAPLGP WLRALGPWLR VRRGLLVLRL AERAAGGEAP SLEDWLCCEY LAEATESSMG QALALLWDLE GGGGA DYKD DDDKGPV UniProtKB: Protein PEAK3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: A final concentration of 0.1% of Octyl-beta-Glucoside (C14H28O6) was added to the sample before freezing. | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278.15 K / 装置: FEI VITROBOT MARK IV / 詳細: blot time = 7s blot force = 4. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均電子線量: 69.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X