+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of EBOV GP lacking the mucin-like domain with 9.20.1A2 Fab and 6D6 scFv bound | |||||||||
 マップデータ マップデータ | Structure of EBOV GP lacking the mucin-like domain with 9.20.1A2 Fab and 6D6 scFv bound | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Antibody / Viral protein / glycoprotein / immune system / Ebola virus / VIRAL PROTEIN-Immune System complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endoplasmic reticulum / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / membrane raft / fusion of virus membrane with host endosome membrane / viral envelope ...host cell endoplasmic reticulum / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / membrane raft / fusion of virus membrane with host endosome membrane / viral envelope / lipid binding / host cell plasma membrane / virion membrane / extracellular region / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Yu X / Saphire EO | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2023 ジャーナル: Cell Rep / 年: 2023タイトル: The evolution and determinants of neutralization of potent head-binding antibodies against Ebola virus. 著者: Xiaoying Yu / Kathryn M Hastie / Carl W Davis / Ruben Diaz Avalos / Dewight Williams / Diptiben Parekh / Sean Hui / Colin Mann / Chitra Hariharan / Ayato Takada / Rafi Ahmed / Erica Ollmann Saphire /   要旨: Monoclonal antibodies against the Ebola virus (EBOV) surface glycoprotein are effective treatments for EBOV disease. Antibodies targeting the EBOV glycoprotein (GP) head epitope have potent ...Monoclonal antibodies against the Ebola virus (EBOV) surface glycoprotein are effective treatments for EBOV disease. Antibodies targeting the EBOV glycoprotein (GP) head epitope have potent neutralization and Fc effector function activity and thus are of high interest as therapeutics and for vaccine design. Here we focus on the head-binding antibodies 1A2 and 1D5, which have been identified previously in a longitudinal study of survivors of EBOV infection. 1A2 and 1D5 have the same heavy- and light-chain germlines despite being isolated from different individuals and at different time points after recovery from infection. Cryoelectron microscopy analysis of each antibody in complex with the EBOV surface GP reveals key amino acid substitutions in 1A2 that contribute to greater affinity, improved neutralization potency, and enhanced breadth as well as two strategies for antibody evolution from a common site. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27638.map.gz emd_27638.map.gz | 170.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27638-v30.xml emd-27638-v30.xml emd-27638.xml emd-27638.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27638.png emd_27638.png | 49.1 KB | ||
| Filedesc metadata |  emd-27638.cif.gz emd-27638.cif.gz | 6.3 KB | ||
| その他 |  emd_27638_half_map_1.map.gz emd_27638_half_map_1.map.gz emd_27638_half_map_2.map.gz emd_27638_half_map_2.map.gz | 171.1 MB 171.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27638 http://ftp.pdbj.org/pub/emdb/structures/EMD-27638 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27638 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27638 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27638_validation.pdf.gz emd_27638_validation.pdf.gz | 901.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27638_full_validation.pdf.gz emd_27638_full_validation.pdf.gz | 900.9 KB | 表示 | |
| XML形式データ |  emd_27638_validation.xml.gz emd_27638_validation.xml.gz | 15.3 KB | 表示 | |
| CIF形式データ |  emd_27638_validation.cif.gz emd_27638_validation.cif.gz | 18.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27638 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27638 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27638 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27638 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dpmMC  8dplC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27638.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27638.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of EBOV GP lacking the mucin-like domain with 9.20.1A2 Fab and 6D6 scFv bound | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||
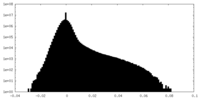
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_27638_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
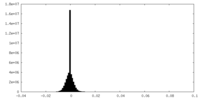
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_27638_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
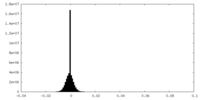
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Ebola virus glycoprotein trimer with 1A2 antibody Fab ...
| 全体 | 名称: Complex of Ebola virus glycoprotein trimer with 1A2 antibody Fab and 6D6 antibody scFv |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Ebola virus glycoprotein trimer with 1A2 antibody Fab ...
| 超分子 | 名称: Complex of Ebola virus glycoprotein trimer with 1A2 antibody Fab and 6D6 antibody scFv タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: Antibody 9.20.1A2 Fab
| 超分子 | 名称: Antibody 9.20.1A2 Fab / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #4-#5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glycoprotein GP1
| 分子 | 名称: Glycoprotein GP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: Mayinga-76 |
| 分子量 | 理論値: 31.297158 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: IPLGVIHNST LQVSDVDKLV CRDKLSSTNQ LRSVGLNLEG NGVATDVPSA TKRWGFRSGV PPKVVNYEAG EWAENCYNLE IKKPDGSEC LPAAPDGIRG FPRCRYVHKV SGTGPCAGDF AFHKEGAFFL YDRLASTVIY RGTTFAEGVV AFLILPQAKK D FFSSHPLR ...文字列: IPLGVIHNST LQVSDVDKLV CRDKLSSTNQ LRSVGLNLEG NGVATDVPSA TKRWGFRSGV PPKVVNYEAG EWAENCYNLE IKKPDGSEC LPAAPDGIRG FPRCRYVHKV SGTGPCAGDF AFHKEGAFFL YDRLASTVIY RGTTFAEGVV AFLILPQAKK D FFSSHPLR EPVNATEDPS SGYYSTTIRY QATGFGTNET EYLFEVDNLT YVQLESRFTP QFLLQLNETI YTSGKRSNTT GK LIWKVNP EIDTTIGEWA FWETKKNLTR KIRSEELSFT VVS UniProtKB: Envelope glycoprotein |
-分子 #2: Glycoprotein GP2
| 分子 | 名称: Glycoprotein GP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.335362 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EAIVNAQPKC NPNLHYWTTQ DEGAAIGLAW IPYFGPAAEG IYTEGLMHNQ DGLICGLRQL ANETTQALQL FLRATTELRT FSILNRKAI DFLLQRWGGT CHILGPDCCI EPHDWTKNIT DKIDQIIHDF VDKTLPD UniProtKB: Envelope glycoprotein |
-分子 #3: Antibody 6D6 scFv
| 分子 | 名称: Antibody 6D6 scFv / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.853459 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQQSGTE LVKPGASVKL SCKASGYTFT SYWMHWVKQR PGQGLEWIGE INPRNGRTDF SEKFKSKATL TVDTSSSTAF IQLSSLTSE DSAVYYCARW GYYGSSDYWG QGTALTVSSG TGGSGGGGSG GGGSGGGASD IVVTQSHKFM STSVGDRVSI T CKASQDVS ...文字列: QVQLQQSGTE LVKPGASVKL SCKASGYTFT SYWMHWVKQR PGQGLEWIGE INPRNGRTDF SEKFKSKATL TVDTSSSTAF IQLSSLTSE DSAVYYCARW GYYGSSDYWG QGTALTVSSG TGGSGGGGSG GGGSGGGASD IVVTQSHKFM STSVGDRVSI T CKASQDVS VAVAWYQQKT GQSPKLLIYS ASYRITGVPD RFTGSGSGTD FTFTISSVQA EDMAVYYCQQ HYSTPPWTFG GG TKL |
-分子 #4: Antibody 9.20.1A2 Fab heavy chain
| 分子 | 名称: Antibody 9.20.1A2 Fab heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.340016 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGGD LVQPGGSLRL SCAASGITLS GVWMNWVRQA PGKGLEWIGR IKSTSDGGRA DFAAPARGRF TMSRDESKNK LFLQMNNLG IEDTGMYYCF TRVQRDGTKD DFWGRGTLVT VSS |
-分子 #5: Antibody 9.20.1A2 Fab light chain
| 分子 | 名称: Antibody 9.20.1A2 Fab light chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.985138 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QSVLTQPPSV SGAPGQTVTI SCTGSYSNIG AGYDVQWYQH LPGTAPKLLI YDNVHRPSGV PDRFSGSKSG TSASLAITGL QTEDEADYY CQSYDSRLRD QWVFGGGTKL TVL |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 81175 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X