+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
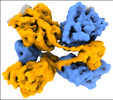
| タイトル | CYP102A1 in Closed Conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cytochrome P450 electron transfer protein dynamic CYP102A1 / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報aromatase activity / NADPH-hemoprotein reductase / NADPH-hemoprotein reductase activity / oxidoreductase activity, acting on paired donors, with incorporation or reduction of molecular oxygen, reduced flavin or flavoprotein as one donor, and incorporation of one atom of oxygen / unspecific monooxygenase / FMN binding / flavin adenine dinucleotide binding / iron ion binding / heme binding / identical protein binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Priestia megaterium NBRC 15308 = ATCC 14581 (バクテリア) Priestia megaterium NBRC 15308 = ATCC 14581 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Su M / Xu H | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Insight into the conformational dynamics of cytochrome P450 CYP102A1 enzyme using Cryo-EM 著者: Su M / Xu H | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27536.map.gz emd_27536.map.gz | 21 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27536-v30.xml emd-27536-v30.xml emd-27536.xml emd-27536.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27536.png emd_27536.png | 115.7 KB | ||
| Filedesc metadata |  emd-27536.cif.gz emd-27536.cif.gz | 6.4 KB | ||
| その他 |  emd_27536_half_map_1.map.gz emd_27536_half_map_1.map.gz emd_27536_half_map_2.map.gz emd_27536_half_map_2.map.gz | 20.6 MB 20.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27536 http://ftp.pdbj.org/pub/emdb/structures/EMD-27536 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27536 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27536 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27536_validation.pdf.gz emd_27536_validation.pdf.gz | 731.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27536_full_validation.pdf.gz emd_27536_full_validation.pdf.gz | 731.1 KB | 表示 | |
| XML形式データ |  emd_27536_validation.xml.gz emd_27536_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  emd_27536_validation.cif.gz emd_27536_validation.cif.gz | 11.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27536 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27536 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27536 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27536 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dmgMC  8dmeC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27536.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27536.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.02 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_27536_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27536_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CYP102A1
| 全体 | 名称: CYP102A1 |
|---|---|
| 要素 |
|
-超分子 #1: CYP102A1
| 超分子 | 名称: CYP102A1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: BM3 CYP102A |
|---|---|
| 由来(天然) | 生物種:  Priestia megaterium NBRC 15308 = ATCC 14581 (バクテリア) Priestia megaterium NBRC 15308 = ATCC 14581 (バクテリア) |
| 分子量 | 理論値: 240 KDa |
-分子 #1: Bifunctional cytochrome P450/NADPH--P450 reductase
| 分子 | 名称: Bifunctional cytochrome P450/NADPH--P450 reductase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Priestia megaterium NBRC 15308 = ATCC 14581 (バクテリア) Priestia megaterium NBRC 15308 = ATCC 14581 (バクテリア)株: ATCC 14581 / DSM 32 / CCUG 1817 / JCM 2506 / NBRC 15308 / NCIMB 9376 / NCTC 10342 / NRRL B-14308 / VKM B-512 / Ford 19 |
| 分子量 | 理論値: 116.887188 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTIKEMPQPK TFGELKNLPL LNTDKPVQAL MKIADELGEI FKFEAPGRVT RYLSSQRLIK EACDESRFDK NLSQALKFVR DFFGDGLFT SWTHEKNWKK AHNILLPSFS QQAMKGYHAM MVDIAVQLVQ KWERLNADEH IEVPEDMTRL TLDTIGLCGF N YRFNSFYR ...文字列: MTIKEMPQPK TFGELKNLPL LNTDKPVQAL MKIADELGEI FKFEAPGRVT RYLSSQRLIK EACDESRFDK NLSQALKFVR DFFGDGLFT SWTHEKNWKK AHNILLPSFS QQAMKGYHAM MVDIAVQLVQ KWERLNADEH IEVPEDMTRL TLDTIGLCGF N YRFNSFYR DQPHPFITSM VRALDEAMNK LQRANPDDPA YDENKRQFQE DIKVMNDLVD KIIADRKASG EQSDDLLTHM LN GKDPETG EPLDDENIRY QIITFLIAGH ETTSGLLSFA LYFLVKNPHV LQKAAEEAAR VLVDPVPSYK QVKQLKYVGM VLN EALRLW PTAPAFSLYA KEDTVLGGEY PLEKGDELMV LIPQLHRDKT IWGDDVEEFR PERFENPSAI PQHAFKPFGN GQRA CIGQQ FALHEATLVL GMMLKHFDFE DHTNYELDIK ETLTLKPEGF VVKAKSKKIP LKKVRKKAEN AHNTPLLVLY GSNMG TAEG TARDLADIAM SKGFAPQVAT LDSHAGNLPR EGAVLIVTAS YNGHPPDNAK QFVDWLDQAS ADEVKGVRYS VFGCGD KNW ATTYQKVPAF IDETLAAKGA ENIADRGEAD ASDDFEGTYE EWREHMWSDV AAYFNLDIEN SEDNKSTLSL QFVDSAA DM PLAKMHGAFS TNVVASKELQ QPGSARSTRH LEIELPKEAS YQEGDHLGVI PRNYEGIVNR VTARFGLDAS QQIRLEAE E EKLAHLPLAK TVSVEELLQY VELQDPVTRT QLRAMAAKTV CPPHKVELEA LLEKQAYKEQ VLAKRLTMLE LLEKYPACE MKFSEFIALL PSIRPRYYSI SSSPRVDEKQ ASITVSVVSG EAWSGYGEYK GIASNYLAEL QEGDTITCFI STPQSEFTLP KDPETPLIM VGPGTGVAPF RGFVQARKQL KEQGQSLGEA HLYFGCRSPH EDYLYQEELE NAQSEGIITL HTAFSRMPNQ P KTYVQHVM EQDGKKLIEL LDQGAHFYIC GDGSQMAPAV EATLMKSYAD VHQVSEADAR LWLQQLEEKG RYAKDVWAG UniProtKB: Bifunctional cytochrome P450/NADPH--P450 reductase |
-分子 #2: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #3: 6-methoxy-2-{[(4-methoxy-3,5-dimethylpyridin-2-yl)methyl]sulfanyl...
| 分子 | 名称: 6-methoxy-2-{[(4-methoxy-3,5-dimethylpyridin-2-yl)methyl]sulfanyl}-1H-benzimidazole タイプ: ligand / ID: 3 / コピー数: 2 / 式: 1C6 |
|---|---|
| 分子量 | 理論値: 329.417 Da |
| Chemical component information |  ChemComp-1C6: |
-分子 #4: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-分子 #5: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #6: SULFATE ION
| 分子 | 名称: SULFATE ION / タイプ: ligand / ID: 6 / コピー数: 6 / 式: SO4 |
|---|---|
| 分子量 | 理論値: 96.063 Da |
| Chemical component information |  ChemComp-SO4: |
-分子 #7: TETRAETHYLENE GLYCOL
| 分子 | 名称: TETRAETHYLENE GLYCOL / タイプ: ligand / ID: 7 / コピー数: 4 / 式: PG4 |
|---|---|
| 分子量 | 理論値: 194.226 Da |
| Chemical component information |  ChemComp-PG4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 50.0 mM / 構成要素 - 式: K2HPO4-KH2PO4-KCL 構成要素 - 名称: sodium phosphate + potassium chloride 詳細: Fresh prepare sodium phosphate (50mM) + potassium chloride (150mM), pH7.4 and filter with 0.22um filters. |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
| 詳細 | The CYP102A1 sample purify and concentrated to 5mg/ml. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 15.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 249374 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)