+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a Xenopus Nucleosome with Widom 601 DNA | |||||||||
 マップデータ マップデータ | Nucleosome | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | meiosis / recombination / chromosome axis / nucleosome / PHD / winged helix / DNA BINDING PROTEIN-DNA complex | |||||||||
| 生物種 |   | |||||||||
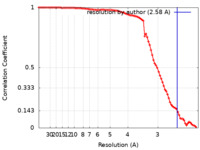
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.58 Å | |||||||||
 データ登録者 データ登録者 | Gu Y / Ur SN / Milano CR / Tromer EC / Vale-Silva LA / Hochwagen A / Corbett KD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2024 ジャーナル: EMBO J / 年: 2024タイトル: Chromatin binding by HORMAD proteins regulates meiotic recombination initiation. 著者: Carolyn R Milano / Sarah N Ur / Yajie Gu / Jessie Zhang / Rachal Allison / George Brown / Matthew J Neale / Eelco C Tromer / Kevin D Corbett / Andreas Hochwagen /    要旨: The meiotic chromosome axis coordinates chromosome organization and interhomolog recombination in meiotic prophase and is essential for fertility. In S. cerevisiae, the HORMAD protein Hop1 mediates ...The meiotic chromosome axis coordinates chromosome organization and interhomolog recombination in meiotic prophase and is essential for fertility. In S. cerevisiae, the HORMAD protein Hop1 mediates the enrichment of axis proteins at nucleosome-rich islands through a central chromatin-binding region (CBR). Here, we use cryoelectron microscopy to show that the Hop1 CBR directly recognizes bent nucleosomal DNA through a composite interface in its PHD and winged helix-turn-helix domains. Targeted disruption of the Hop1 CBR-nucleosome interface causes a localized reduction of axis protein binding and meiotic DNA double-strand breaks (DSBs) in axis islands and leads to defects in chromosome synapsis. Synthetic effects with mutants of the Hop1 regulator Pch2 suggest that nucleosome binding delays a conformational switch in Hop1 from a DSB-promoting, Pch2-inaccessible state to a DSB-inactive, Pch2-accessible state to regulate the extent of meiotic DSB formation. Phylogenetic analyses of meiotic HORMADs reveal an ancient origin of the CBR, suggesting that the mechanisms we uncover are broadly conserved. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27096.map.gz emd_27096.map.gz | 32.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27096-v30.xml emd-27096-v30.xml emd-27096.xml emd-27096.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27096_fsc.xml emd_27096_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27096.png emd_27096.png | 158.7 KB | ||
| Filedesc metadata |  emd-27096.cif.gz emd-27096.cif.gz | 6.3 KB | ||
| その他 |  emd_27096_half_map_1.map.gz emd_27096_half_map_1.map.gz emd_27096_half_map_2.map.gz emd_27096_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27096 http://ftp.pdbj.org/pub/emdb/structures/EMD-27096 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27096 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27096 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27096_validation.pdf.gz emd_27096_validation.pdf.gz | 905.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27096_full_validation.pdf.gz emd_27096_full_validation.pdf.gz | 904.6 KB | 表示 | |
| XML形式データ |  emd_27096_validation.xml.gz emd_27096_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_27096_validation.cif.gz emd_27096_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27096 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27096 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27096 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27096 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27096.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27096.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nucleosome | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_27096_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
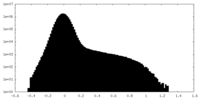
| 投影像・断面図 |
| ||||||||||||
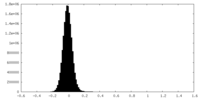
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27096_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
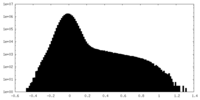
| 投影像・断面図 |
| ||||||||||||
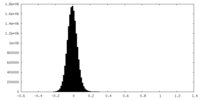
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome
| 全体 | 名称: Nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome
| 超分子 | 名称: Nucleosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 200 KDa |
-分子 #1: Histone H3
| 分子 | 名称: Histone H3 / タイプ: protein_or_peptide / ID: 1 / 詳細: GenBank:CAD89679.1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.30393 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVALFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / 詳細: GenBank:NP_001087926.1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.263231 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG |
-分子 #3: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 3 / 詳細: GenBank:CAD89676.1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.978241 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKT RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAVRN DEELNKLLGR VTIAQGGVLP NIQSVLLPKK TESSKSAKSK |
-分子 #4: Histone H2B
| 分子 | 名称: Histone H2B / タイプ: protein_or_peptide / ID: 4 / 詳細: GenBank:CAD89678.1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.524752 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AKSAPAPKKG SKKAVTKTQK KDGKKRRKTR KESYAIYVYK VLKQVHPDTG ISSKAMSIMN SFVNDVFERI AGEASRLAHY NKRSTITSR EIQTAVRLLL PGELAKHAVS EGTKAVTKYT SAK |
-分子 #5: Widom 601 DNA (146-MER)
| 分子 | 名称: Widom 601 DNA (146-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.27484 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT) (DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC)(DT) (DC)(DC)(DA) |
-分子 #6: Widom 601 DNA (146-MER)
| 分子 | 名称: Widom 601 DNA (146-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 44.85657 KDa |
| 配列 | 文字列: (DT)(DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT) ...文字列: (DT)(DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT)(DC)(DT)(DA) (DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT)(DA) (DA)(DA) (DC)(DG)(DC)(DA)(DC)(DG)(DT) (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT)(DC) (DC)(DC)(DC) (DC)(DG)(DC)(DG)(DT)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DA) (DA)(DG)(DG)(DG) (DG)(DA)(DT)(DT)(DA) (DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT)(DC) (DT)(DC)(DC)(DA)(DG) (DG)(DC)(DA)(DC) (DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT)(DA) (DT)(DA)(DT)(DA)(DC)(DA) (DT)(DC)(DC) (DT)(DG)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20mM Tris 7.5, 50mM NaCl, 1mM DTT, 1mM EDTA |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均露光時間: 10.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8cze: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)