+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structures and computational analysis for enhanced potency in MTA-synergic inhibition of human protein arginine methyltransferase 5 | |||||||||
 マップデータ マップデータ | human protein arginine methyltransferase 5 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PRMT5 / protein arginine methyl transferase / MTA-inhibitor synergy / cryo-EM structure-based drug design / computational analysis / catalytic mechanism / drug discovery / docking analysis / ONCOPROTEIN / ONCOPROTEIN-Transferase complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of adenylate cyclase-inhibiting dopamine receptor signaling pathway / peptidyl-arginine N-methylation / oocyte axis specification / type II protein arginine methyltransferase / protein-arginine omega-N symmetric methyltransferase activity / peptidyl-arginine methylation / Golgi ribbon formation / negative regulation of epithelial cell proliferation involved in prostate gland development / histone H4R3 methyltransferase activity / secretory columnal luminar epithelial cell differentiation involved in prostate glandular acinus development ...positive regulation of adenylate cyclase-inhibiting dopamine receptor signaling pathway / peptidyl-arginine N-methylation / oocyte axis specification / type II protein arginine methyltransferase / protein-arginine omega-N symmetric methyltransferase activity / peptidyl-arginine methylation / Golgi ribbon formation / negative regulation of epithelial cell proliferation involved in prostate gland development / histone H4R3 methyltransferase activity / secretory columnal luminar epithelial cell differentiation involved in prostate glandular acinus development / histone arginine N-methyltransferase activity / epithelial cell proliferation involved in prostate gland development / protein-arginine N-methyltransferase activity / methylosome / methyl-CpG binding / endothelial cell activation / histone H3 methyltransferase activity / positive regulation of mRNA splicing, via spliceosome / negative regulation of gene expression via chromosomal CpG island methylation / Cul4B-RING E3 ubiquitin ligase complex / regulation of mitotic nuclear division / histone methyltransferase complex / positive regulation of oligodendrocyte differentiation / histone methyltransferase activity / E-box binding / negative regulation of cell differentiation / ubiquitin-like ligase-substrate adaptor activity / ribonucleoprotein complex binding / spliceosomal snRNP assembly / regulation of ERK1 and ERK2 cascade / nuclear receptor coactivator activity / regulation of signal transduction by p53 class mediator / methyltransferase activity / liver regeneration / DNA-templated transcription termination / circadian regulation of gene expression / Regulation of TP53 Activity through Methylation / RMTs methylate histone arginines / protein polyubiquitination / transcription corepressor activity / p53 binding / snRNP Assembly / ubiquitin-dependent protein catabolic process / chromatin remodeling / protein heterodimerization activity / positive regulation of cell population proliferation / chromatin / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / Golgi apparatus / nucleoplasm / identical protein binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||
 データ登録者 データ登録者 | Yadav GP / Wei Z / Xiaozhi Y / Chenglong L / Jiang Q | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2022 ジャーナル: Commun Biol / 年: 2022タイトル: Cryo-EM structure-based selection of computed ligand poses enables design of MTA-synergic PRMT5 inhibitors of better potency. 著者: Wei Zhou / Gaya P Yadav / Xiaozhi Yang / Feng Qin / Chenglong Li / Qiu-Xing Jiang /  要旨: Projected potential of 2.5-4.0 Å cryo-EM structures for structure-based drug design is not well realized yet. Here we show that a 3.1 Å structure of PRMT5 is suitable for selecting computed ...Projected potential of 2.5-4.0 Å cryo-EM structures for structure-based drug design is not well realized yet. Here we show that a 3.1 Å structure of PRMT5 is suitable for selecting computed poses of a chemical inhibitor and its analogs for enhanced potency. PRMT5, an oncogenic target for various cancer types, has many inhibitors manifesting little cooperativity with MTA, a co-factor analog accumulated in MTAP-/- cells. To achieve MTA-synergic inhibition, a pharmacophore from virtual screen leads to a specific inhibitor (11-2 F). Cryo-EM structures of 11-2 F / MTA-bound human PRMT5/MEP50 complex and its apo form resolved at 3.1 and 3.2 Å respectively show that 11-2 F in the catalytic pocket shifts the cofactor-binding pocket away by ~2.0 Å, contributing to positive cooperativity. Computational analysis predicts subtype specificity of 11-2 F among PRMTs. Structural analysis of ligands in the binding pockets is performed to compare poses of 11-2 F and its redesigned analogs and identifies three new analogs predicted to have significantly better potency. One of them, after synthesis, is ~4 fold more efficient in inhibiting PRMT5 catalysis than 11-2 F, with strong MTA-synergy. These data suggest the feasibility of employing near-atomic resolution cryo-EM structures and computational analysis of ligand poses for small molecule therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27078.map.gz emd_27078.map.gz | 59.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27078-v30.xml emd-27078-v30.xml emd-27078.xml emd-27078.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27078.png emd_27078.png | 267.1 KB | ||
| Filedesc metadata |  emd-27078.cif.gz emd-27078.cif.gz | 6.6 KB | ||
| その他 |  emd_27078_half_map_1.map.gz emd_27078_half_map_1.map.gz emd_27078_half_map_2.map.gz emd_27078_half_map_2.map.gz | 12.8 MB 12.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27078 http://ftp.pdbj.org/pub/emdb/structures/EMD-27078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27078 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27078_validation.pdf.gz emd_27078_validation.pdf.gz | 808.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27078_full_validation.pdf.gz emd_27078_full_validation.pdf.gz | 807.9 KB | 表示 | |
| XML形式データ |  emd_27078_validation.xml.gz emd_27078_validation.xml.gz | 12.6 KB | 表示 | |
| CIF形式データ |  emd_27078_validation.cif.gz emd_27078_validation.cif.gz | 14.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27078 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27078 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cyiMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27078.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27078.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | human protein arginine methyltransferase 5 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_27078_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
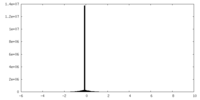
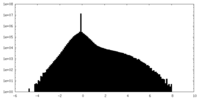
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_27078_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
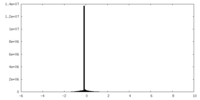
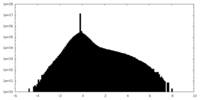
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of PRMT5 and MEP50 in the presence of MTA and a novel inh...
| 全体 | 名称: Complex of PRMT5 and MEP50 in the presence of MTA and a novel inhibitor (11-2F) |
|---|---|
| 要素 |
|
-超分子 #1: Complex of PRMT5 and MEP50 in the presence of MTA and a novel inh...
| 超分子 | 名称: Complex of PRMT5 and MEP50 in the presence of MTA and a novel inhibitor (11-2F) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 440 KDa |
-分子 #1: Protein arginine N-methyltransferase 5
| 分子 | 名称: Protein arginine N-methyltransferase 5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: type II protein arginine methyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 72.766664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAMAVGGAG GSRVSSGRDL NCVPEIADTL GAVAKQGFDF LCMPVFHPRF KREFIQEPAK NRPGPQTRSD LLLSGRDWNT LIVGKLSPW IRPDSKVEKI RRNSEAAMLQ ELNFGAYLGL PAFLLPLNQE DNTNLARVLT NHIHTGHHSS MFWMRVPLVA P EDLRDDII ...文字列: MAAMAVGGAG GSRVSSGRDL NCVPEIADTL GAVAKQGFDF LCMPVFHPRF KREFIQEPAK NRPGPQTRSD LLLSGRDWNT LIVGKLSPW IRPDSKVEKI RRNSEAAMLQ ELNFGAYLGL PAFLLPLNQE DNTNLARVLT NHIHTGHHSS MFWMRVPLVA P EDLRDDII ENAPTTHTEE YSGEEKTWMW WHNFRTLCDY SKRIAVALEI GADLPSNHVI DRWLGEPIKA AILPTSIFLT NK KGFPVLS KMHQRLIFRL LKLEVQFIIT GTNHHSEKEF CSYLQYLEYL SQNRPPPNAY ELFAKGYEDY LQSPLQPLMD NLE SQTYEV FEKDPIKYSQ YQQAIYKCLL DRVPEEEKDT NVQVLMVLGA GRGPLVNASL RAAKQADRRI KLYAVEKNPN AVVT LENWQ FEEWGSQVTV VSSDMREWVA PEKADIIVSE LLGSFADNEL SPECLDGAQH FLKDDGVSIP GEYTSFLAPI SSSKL YNEV RACREKDRDP EAQFEMPYVV RLHNFHQLSA PQPCFTFSHP NRDPMIDNNR YCTLEFPVEV NTVLHGFAGY FETVLY QDI TLSIRPETHS PGMFSWFPIL FPIKQPITVR EGQTICVRFW RCSNSKKVWY EWAVTAPVCS AIHNPTGRSY TIGL UniProtKB: Protein arginine N-methyltransferase 5 |
-分子 #2: Methylosome protein 50
| 分子 | 名称: Methylosome protein 50 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 33.226211 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PNAPACMERQ LEAARYRSDG ALLLGASSLS GRCWAGSLWL FKDPCAAPNE GFCSAGVQTE AGVADLTWVG ERGILVASDS GAVELWELD ENETLIVSKF CKYEHDDIVS TVSVLSSGTQ AVSGSKDICI KVWDLAQQVV LSSYRAHAAQ VTCVAASPHK D SVFLSCSE ...文字列: PNAPACMERQ LEAARYRSDG ALLLGASSLS GRCWAGSLWL FKDPCAAPNE GFCSAGVQTE AGVADLTWVG ERGILVASDS GAVELWELD ENETLIVSKF CKYEHDDIVS TVSVLSSGTQ AVSGSKDICI KVWDLAQQVV LSSYRAHAAQ VTCVAASPHK D SVFLSCSE DNRILLWDTR CPKPASQIGC SAPGYLPTSL AWHPQQSEVF VFGDENGTVS LVDTKSTSCV LSSAVHSQCV TG LVFSPHS VPFLASLSED CSLAVLDSSL SELFRSQAHR DFVRDATWSP LNHSLLTTVG WDHQVVHHVV PT UniProtKB: Methylosome protein WDR77 |
-分子 #3: N-[(2-aminoquinolin-7-yl)methyl]-9-(2-hydroxyethyl)-2,3,4,9-tetra...
| 分子 | 名称: N-[(2-aminoquinolin-7-yl)methyl]-9-(2-hydroxyethyl)-2,3,4,9-tetrahydro-1H-carbazole-6-carboxamide タイプ: ligand / ID: 3 / コピー数: 1 / 式: P2R |
|---|---|
| 分子量 | 理論値: 414.5 Da |
| Chemical component information | 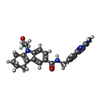 ChemComp-P2R: |
-分子 #4: 5'-DEOXY-5'-METHYLTHIOADENOSINE
| 分子 | 名称: 5'-DEOXY-5'-METHYLTHIOADENOSINE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MTA |
|---|---|
| 分子量 | 理論値: 297.334 Da |
| Chemical component information | 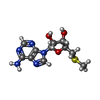 ChemComp-MTA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: OTHER / 詳細: Preclean plasma cleaner |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 4115 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: DARK FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 68 |
|---|---|
| 得られたモデル |  PDB-8cyi: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X