+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 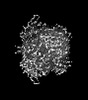 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Mtb Lpd bound to inhibitor complex with 2-((2-cyano-N,5-dimethyl-1H-indole)-7-sulfonamido)-N-(4-(oxetan-3-yl)-3,4-dihydro-2H-benzo[b] [1,4]oxazin-7-yl)acetamide | ||||||||||||
 マップデータ マップデータ | Density-modified map generated using phenix.cryoem.resolve | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | flavoprotein / glycolysis / redox-active center / OXIDOREDUCTASE-INHIBITOR complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Cell redox homeostasis / dihydrolipoyl dehydrogenase / dihydrolipoyl dehydrogenase (NADH) activity / NADH binding / disulfide oxidoreductase activity / pyruvate dehydrogenase complex / 2-oxoglutarate metabolic process / zymogen binding / pyruvate metabolic process / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor ...Cell redox homeostasis / dihydrolipoyl dehydrogenase / dihydrolipoyl dehydrogenase (NADH) activity / NADH binding / disulfide oxidoreductase activity / pyruvate dehydrogenase complex / 2-oxoglutarate metabolic process / zymogen binding / pyruvate metabolic process / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor / antioxidant activity / Prevention of phagosomal-lysosomal fusion / cell redox homeostasis / flavin adenine dinucleotide binding / extracellular region / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
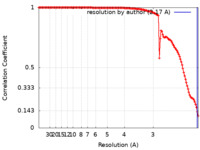
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.17 Å | ||||||||||||
 データ登録者 データ登録者 | Kochanczyk T / Arango N / Lima CD | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Not published ジャーナル: Not publishedタイトル: Cryo-EM structure of Mtb Lpd bound to the inhibitor 2-((2-cyano-N,5-dimethyl-1H-indole)-7-sulfonamido)-N-(4-(oxetan-3-yl)-3,4-dihydro-2H-benzo[b] [1,4]oxazin-7-yl)acetamide at 2.17 Angstrom resolution 著者: Kochanczyk T / Arango N / Michino M / Sun S / Ginn J / Bryk R / Nathan C / Lima CD | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
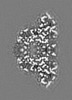
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26981.map.gz emd_26981.map.gz | 3.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26981-v30.xml emd-26981-v30.xml emd-26981.xml emd-26981.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26981_fsc.xml emd_26981_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26981.png emd_26981.png | 76.3 KB | ||
| Filedesc metadata |  emd-26981.cif.gz emd-26981.cif.gz | 6.5 KB | ||
| その他 |  emd_26981_additional_1.map.gz emd_26981_additional_1.map.gz emd_26981_half_map_1.map.gz emd_26981_half_map_1.map.gz emd_26981_half_map_2.map.gz emd_26981_half_map_2.map.gz | 109.6 MB 200.3 MB 200.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26981 http://ftp.pdbj.org/pub/emdb/structures/EMD-26981 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26981 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26981 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26981_validation.pdf.gz emd_26981_validation.pdf.gz | 751.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26981_full_validation.pdf.gz emd_26981_full_validation.pdf.gz | 751.2 KB | 表示 | |
| XML形式データ |  emd_26981_validation.xml.gz emd_26981_validation.xml.gz | 19.3 KB | 表示 | |
| CIF形式データ |  emd_26981_validation.cif.gz emd_26981_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26981 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26981 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26981 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26981 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ct4MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26981.map.gz / 形式: CCP4 / 大きさ: 4.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26981.map.gz / 形式: CCP4 / 大きさ: 4.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density-modified map generated using phenix.cryoem.resolve | ||||||||||||||||||||||||||||||||||||
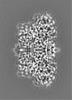
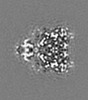
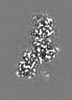
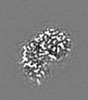
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.064 Å | ||||||||||||||||||||||||||||||||||||
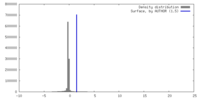
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Raw map
| ファイル | emd_26981_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map | ||||||||||||
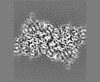
| 投影像・断面図 |
| ||||||||||||
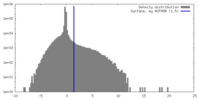
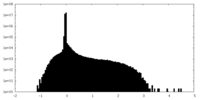
| 密度ヒストグラム |
-ハーフマップ: half-map 1
| ファイル | emd_26981_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 1 | ||||||||||||
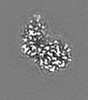
| 投影像・断面図 |
| ||||||||||||
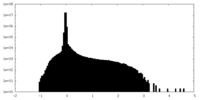
| 密度ヒストグラム |
-ハーフマップ: half-map 2
| ファイル | emd_26981_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 2 | ||||||||||||
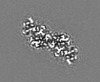
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dihydrolipoyl dehydrogenase in complex with 2-((2-cyano-N,5-dimet...
| 全体 | 名称: Dihydrolipoyl dehydrogenase in complex with 2-((2-cyano-N,5-dimethyl-1H-indole)-7-sulfonamido)-N-(4-(oxetan-3-yl)-3,4-dihydro-2H-benzo[b] [1,4]oxazin-7-yl)acetamide |
|---|---|
| 要素 |
|
-超分子 #1: Dihydrolipoyl dehydrogenase in complex with 2-((2-cyano-N,5-dimet...
| 超分子 | 名称: Dihydrolipoyl dehydrogenase in complex with 2-((2-cyano-N,5-dimethyl-1H-indole)-7-sulfonamido)-N-(4-(oxetan-3-yl)-3,4-dihydro-2H-benzo[b] [1,4]oxazin-7-yl)acetamide タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: 2-((2-cyano-N,5-dimethyl-1H-indole)-7-sulfonamido)-N-(4-(oxetan-3-yl)-3,4-dihydro-2H-benzo[b] [1,4]oxazin-7-yl)acetamide provided by the Tri-Institutional Therapeutics Discovery Institute ...詳細: 2-((2-cyano-N,5-dimethyl-1H-indole)-7-sulfonamido)-N-(4-(oxetan-3-yl)-3,4-dihydro-2H-benzo[b] [1,4]oxazin-7-yl)acetamide provided by the Tri-Institutional Therapeutics Discovery Institute (tritdi.org) contact: John Ginn (jginn@tritdi.org) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 99 KDa |
-分子 #1: Dihydrolipoyl dehydrogenase
| 分子 | 名称: Dihydrolipoyl dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: dihydrolipoyl dehydrogenase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.437035 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMTHYDVVV LGAGPGGYVA AIRAAQLGLS TAIVEPKYWG GVCLNVGCIP SKALLRNAEL VHIFTKDAKA FGISGEVTFD YGIAYDRSR KVAEGRVAGV HFLMKKNKIT EIHGYGTFAD ANTLLVDLND GGTESVTFDN AIIATGSSTR LVPGTSLSAN V VTYEEQIL ...文字列: GSMTHYDVVV LGAGPGGYVA AIRAAQLGLS TAIVEPKYWG GVCLNVGCIP SKALLRNAEL VHIFTKDAKA FGISGEVTFD YGIAYDRSR KVAEGRVAGV HFLMKKNKIT EIHGYGTFAD ANTLLVDLND GGTESVTFDN AIIATGSSTR LVPGTSLSAN V VTYEEQIL SRELPKSIII AGAGAIGMEF GYVLKNYGVD VTIVEFLPRA LPNEDADVSK EIEKQFKKLG VTILTATKVE SI ADGGSQV TVTVTKDGVA QELKAEKVLQ AIGFAPNVEG YGLDKAGVAL TDRKAIGVDD YMRTNVGHIY AIGDVNGLLQ LAH VAEAQG VVAAETIAGA ETLTLGDHRM LPRATFCQPN VASFGLTEQQ ARNEGYDVVV AKFPFTANAK AHGVGDPSGF VKLV ADAKH GELLGGHLVG HDVAELLPEL TLAQRWDLTA SELARNVHTH PTMSEALQEC FHGLVGHMIN F UniProtKB: Dihydrolipoyl dehydrogenase |
-分子 #2: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #3: N~2~-(2-cyano-5-methyl-1H-indole-7-sulfonyl)-N~2~-methyl-N-[4-(ox...
| 分子 | 名称: N~2~-(2-cyano-5-methyl-1H-indole-7-sulfonyl)-N~2~-methyl-N-[4-(oxetan-3-yl)-3,4-dihydro-2H-1,4-benzoxazin-7-yl]glycinamide タイプ: ligand / ID: 3 / コピー数: 2 / 式: OU6 |
|---|---|
| 分子量 | 理論値: 495.551 Da |
| Chemical component information | 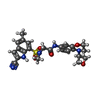 ChemComp-OU6: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 665 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20 mM Tris-HCl, pH 8.0, 87.5 mM NaCl, 0.05% IGEPAL |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV 詳細: Temperature 22 C, humidity 100%, Wait time 8s, Blot time: 3.5s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 10748 / 平均露光時間: 4.0 sec. / 平均電子線量: 70.66 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: AB / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| ソフトウェア | 名称: PHENIX (ver. 1.20.1-44487) |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-8ct4: |
 ムービー
ムービー コントローラー
コントローラー









 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)