+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 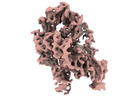 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Kinetically trapped misfolded state of the Tetrahymena ribozyme | |||||||||
 マップデータ マップデータ | Map of misfolded state (M state) of Tetrahymena ribozyme sharpened with a B factor of 167. Refinement was performed in cryoSPARC using a 'non-uniform refinement' job. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribozyme / catalytic RNA / folding intermediate / misfolded / kinetic trap / M state / Tetrahymena / RNA | |||||||||
| 生物種 |  | |||||||||
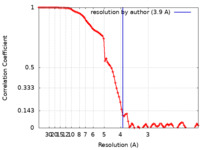
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Bonilla SL / Vicens Q / Kieft JS | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Cryo-EM reveals an entangled kinetic trap in the folding of a catalytic RNA. 著者: Steve L Bonilla / Quentin Vicens / Jeffrey S Kieft /  要旨: Functional RNAs fold through complex pathways that can contain misfolded "kinetic traps." A complete model of RNA folding requires understanding the formation of these misfolded states, but they are ...Functional RNAs fold through complex pathways that can contain misfolded "kinetic traps." A complete model of RNA folding requires understanding the formation of these misfolded states, but they are difficult to characterize because of their transient and potentially conformationally dynamic nature. We used cryo-electron microscopy (cryo-EM) to visualize a long-lived misfolded state in the folding pathway of the group I intron, a paradigmatic RNA structure-function model system. The structure revealed how this state forms native-like secondary structure and tertiary contacts but contains two incorrectly crossed strands, consistent with a previous model. This incorrect topology mispositions a critical catalytic domain and cannot be resolved locally as extensive refolding is required. This work provides a structural framework for interpreting decades of biochemical and functional studies and demonstrates the power of cryo-EM for the exploration of RNA folding pathways. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26816.map.gz emd_26816.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26816-v30.xml emd-26816-v30.xml emd-26816.xml emd-26816.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26816_fsc.xml emd_26816_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26816.png emd_26816.png | 69.5 KB | ||
| Filedesc metadata |  emd-26816.cif.gz emd-26816.cif.gz | 4.6 KB | ||
| その他 |  emd_26816_additional_1.map.gz emd_26816_additional_1.map.gz emd_26816_half_map_1.map.gz emd_26816_half_map_1.map.gz emd_26816_half_map_2.map.gz emd_26816_half_map_2.map.gz | 32 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26816 http://ftp.pdbj.org/pub/emdb/structures/EMD-26816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26816 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26816_validation.pdf.gz emd_26816_validation.pdf.gz | 861.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26816_full_validation.pdf.gz emd_26816_full_validation.pdf.gz | 861.4 KB | 表示 | |
| XML形式データ |  emd_26816_validation.xml.gz emd_26816_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_26816_validation.cif.gz emd_26816_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26816 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26816 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26816 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26816 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26816.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26816.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of misfolded state (M state) of Tetrahymena ribozyme sharpened with a B factor of 167. Refinement was performed in cryoSPARC using a 'non-uniform refinement' job. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.01156 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map of misfolded state (M state) of...
| ファイル | emd_26816_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map of misfolded state (M state) of Tetrahymena ribozyme. Refinement was performed in cryoSPARC using a 'non-uniform refinement' job. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B of misfolded state (M state)...
| ファイル | emd_26816_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of misfolded state (M state) of Tetrahymena ribozyme sharpened with a B factor of 167. Refinement was performed in cryoSPARC using a 'non-uniform refinement' job. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A of misfolded state (M state)...
| ファイル | emd_26816_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of misfolded state (M state) of Tetrahymena ribozyme sharpened with a B factor of 167. Refinement was performed in cryoSPARC using a 'non-uniform refinement' job. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : apo L-21 ScaI Tetrahymena Ribozyme RNA
| 全体 | 名称: apo L-21 ScaI Tetrahymena Ribozyme RNA |
|---|---|
| 要素 |
|
-超分子 #1: apo L-21 ScaI Tetrahymena Ribozyme RNA
| 超分子 | 名称: apo L-21 ScaI Tetrahymena Ribozyme RNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Misfolded state. |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: RNA (386-MER)
| 分子 | 名称: RNA (386-MER) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 125.096773 KDa |
| 配列 | 文字列: GGAGGGAAAA GUUAUCAGGC AUGCACCUGG UAGCUAGUCU UUAAACCAAU AGAUUGCAUC GGUUUAAAAG GCAAGACCGU CAAAUUGCG GGAAAGGGGU CAACAGCCGU UCAGUACCAA GUCUCAGGGG AAACUUUGAG AUGGCCUUGC AAAGGGUAUG G UAAUAAGC ...文字列: GGAGGGAAAA GUUAUCAGGC AUGCACCUGG UAGCUAGUCU UUAAACCAAU AGAUUGCAUC GGUUUAAAAG GCAAGACCGU CAAAUUGCG GGAAAGGGGU CAACAGCCGU UCAGUACCAA GUCUCAGGGG AAACUUUGAG AUGGCCUUGC AAAGGGUAUG G UAAUAAGC UGACGGACAU GGUCCUAACC ACGCAGCCAA GUCCUAAGUC AACAGAUCUU CUGUUGAUAU GGAUGCAGUU CA CAGACUA AAUGUCGGUC GGGGAAGAUG UAUUCUUCUC AUAAGAUAUA GUCGGACCUC UCCUUAAUGG GAGCUAGCGG AUG AAGUGA UGCAACACUG GAGCCGCUGG GAACUAAUUU GUAUGCGAAA GUAUAUUGAU UAGUUUUGGA G GENBANK: GENBANK: X54512.1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 詳細: Buffer contained 50 mM NaMOPS, pH 7.0 and 10 mM MgCl2 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 32.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X