+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Shiga toxin 2 in complex with the native ribosomal P-stalk | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Shiga toxin 2 / Ribosomal P-stalk / Complex / TOXIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報rRNA N-glycosylase / rRNA N-glycosylase activity / toxin activity / negative regulation of translation 類似検索 - 分子機能 | |||||||||
| 生物種 |  Shigella dysenteriae (志賀赤痢菌) / Shigella dysenteriae (志賀赤痢菌) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Kulczyk AW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2023 ジャーナル: J Biol Chem / 年: 2023タイトル: Cryo-EM structure of Shiga toxin 2 in complex with the native ribosomal P-stalk reveals residues involved in the binding interaction. 著者: Arkadiusz W Kulczyk / Carlos Oscar S Sorzano / Przemysław Grela / Marek Tchórzewski / Nilgun E Tumer / Xiao-Ping Li /    要旨: Shiga toxin 2a (Stx2a) is the virulence factor of enterohemorrhagic Escherichia coli. The catalytic A1 subunit of Stx2a (Stx2A1) interacts with the ribosomal P-stalk for loading onto the ribosome and ...Shiga toxin 2a (Stx2a) is the virulence factor of enterohemorrhagic Escherichia coli. The catalytic A1 subunit of Stx2a (Stx2A1) interacts with the ribosomal P-stalk for loading onto the ribosome and depurination of the sarcin-ricin loop, which halts protein synthesis. Because of the intrinsic flexibility of the P-stalk, a structure of the Stx2a-P-stalk complex is currently unknown. We demonstrated that the native P-stalk pentamer binds to Stx2a with nanomolar affinity, and we employed cryo-EM to determine a structure of the 72 kDa Stx2a complexed with the P-stalk. The structure identifies Stx2A1 residues involved in binding and reveals that Stx2a is anchored to the P-stalk via only the last six amino acids from the C-terminal domain of a single P-protein. For the first time, the cryo-EM structure shows the loop connecting Stx2A1 and Stx2A2, which is critical for activation of the toxin. Our principal component analysis of the cryo-EM data reveals the intrinsic dynamics of the Stx2a-P-stalk interaction, including conformational changes in the P-stalk binding site occurring upon complex formation. Our computational analysis unveils the propensity for structural rearrangements within the C-terminal domain, with its C-terminal six amino acids transitioning from a random coil to an α-helix upon binding to Stx2a. In conclusion, our cryo-EM structure sheds new light into the dynamics of the Stx2a-P-stalk interaction and indicates that the binding interface between Stx2a and the P-stalk is the potential target for drug discovery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26381.map.gz emd_26381.map.gz | 6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26381-v30.xml emd-26381-v30.xml emd-26381.xml emd-26381.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26381_fsc.xml emd_26381_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26381.png emd_26381.png | 61.7 KB | ||
| Filedesc metadata |  emd-26381.cif.gz emd-26381.cif.gz | 5.7 KB | ||
| その他 |  emd_26381_half_map_1.map.gz emd_26381_half_map_1.map.gz emd_26381_half_map_2.map.gz emd_26381_half_map_2.map.gz | 80.7 MB 80.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26381 http://ftp.pdbj.org/pub/emdb/structures/EMD-26381 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26381 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26381 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26381_validation.pdf.gz emd_26381_validation.pdf.gz | 636.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26381_full_validation.pdf.gz emd_26381_full_validation.pdf.gz | 636.3 KB | 表示 | |
| XML形式データ |  emd_26381_validation.xml.gz emd_26381_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_26381_validation.cif.gz emd_26381_validation.cif.gz | 23.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26381 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26381 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26381 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26381 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7u6vMC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26381.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26381.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8248 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_26381_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
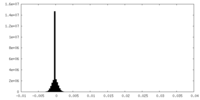
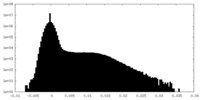
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26381_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
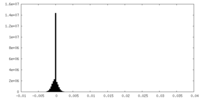
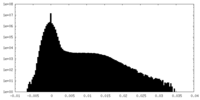
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Shiga toxin 2a in complex with the native ribosomal P-stalk
| 全体 | 名称: Shiga toxin 2a in complex with the native ribosomal P-stalk |
|---|---|
| 要素 |
|
-超分子 #1: Shiga toxin 2a in complex with the native ribosomal P-stalk
| 超分子 | 名称: Shiga toxin 2a in complex with the native ribosomal P-stalk タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 100 KDa |
-超分子 #2: Shiga toxin 2a
| 超分子 | 名称: Shiga toxin 2a / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 / 詳細: 72 KDa Stx2a holotoxin |
|---|---|
| 由来(天然) | 生物種:  Shigella dysenteriae (志賀赤痢菌) Shigella dysenteriae (志賀赤痢菌) |
-超分子 #3: Native ribosomal P-stalk
| 超分子 | 名称: Native ribosomal P-stalk / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 / 詳細: 56 kDa native ribosomal P-stalk |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Shiga toxin 2a subunit A (Stx2A)
| 分子 | 名称: Shiga toxin 2a subunit A (Stx2A) / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: rRNA N-glycosylase |
|---|---|
| 由来(天然) | 生物種:  Shigella dysenteriae (志賀赤痢菌) Shigella dysenteriae (志賀赤痢菌) |
| 分子量 | 理論値: 33.214188 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: REFTIDFSTQ QSYVSSLNSI RTEISTPLEH ISQGTTSVSV INHTPPGSYF AVDIRGLDVY QARFDHLRLI IEQNNLYVAG FVNTATNTF YRFSDFTHIS VPGVTTVSMT TDSSYTTLQR VAALERSGMQ ISRHSLVSSY LALMEFSGNT MTRDASRAVL R FVTVTAEA ...文字列: REFTIDFSTQ QSYVSSLNSI RTEISTPLEH ISQGTTSVSV INHTPPGSYF AVDIRGLDVY QARFDHLRLI IEQNNLYVAG FVNTATNTF YRFSDFTHIS VPGVTTVSMT TDSSYTTLQR VAALERSGMQ ISRHSLVSSY LALMEFSGNT MTRDASRAVL R FVTVTAEA LRFRQIQREF RQALSETAPV YTMTPGDVDL TLNWGRISNV LPEYRGEDGV RVGRISFNNI SAILGTVAVI LN CHHQGAR SVRAVNEDSQ PECQITGDRP VIKINNTLWE SNTAAAFLNR KSQFLYTTGK UniProtKB: rRNA N-glycosylase |
-分子 #2: Shiga toxin 2a subunit B (Stx2B)
| 分子 | 名称: Shiga toxin 2a subunit B (Stx2B) / タイプ: protein_or_peptide / ID: 2 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Shigella dysenteriae (志賀赤痢菌) Shigella dysenteriae (志賀赤痢菌) |
| 分子量 | 理論値: 7.82459 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ADCAKGKIEF SKYNEDDTFT VKVDGKEYWT SRWNLQPLLQ SAQLTGMTVT IKSSTCESGS GFAEVQFNND |
-分子 #3: C-terminal domain (CTD) from the Ribosomal P-stalk
| 分子 | 名称: C-terminal domain (CTD) from the Ribosomal P-stalk / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 654.712 Da |
| 配列 | 文字列: GFGLFD |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 23 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 1.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7u6v: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)