+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
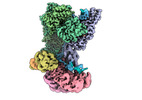
| タイトル | I-F3b Cascade-TniQ full R-loop complex | |||||||||
 マップデータ マップデータ | combined map of local-resolution filtered maps from full refinement and focused refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / Transposon / CAST / Cascade / I-F / I-F3 / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 生物種 |  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) | |||||||||
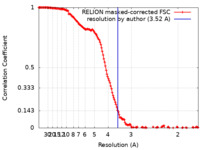
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.52 Å | |||||||||
 データ登録者 データ登録者 | Park JU / Mehrotra E / Kellogg EH | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Multiple adaptations underly co-option of a CRISPR surveillance complex for RNA-guided DNA transposition. 著者: Jung-Un Park / Michael T Petassi / Shan-Chi Hsieh / Eshan Mehrotra / Gabriel Schuler / Jagat Budhathoki / Vinh H Truong / Summer B Thyme / Ailong Ke / Elizabeth H Kellogg / Joseph E Peters /  要旨: CRISPR-associated transposons (CASTs) are natural RNA-directed transposition systems. We demonstrate that transposon protein TniQ plays a central role in promoting R-loop formation by RNA-guided DNA- ...CRISPR-associated transposons (CASTs) are natural RNA-directed transposition systems. We demonstrate that transposon protein TniQ plays a central role in promoting R-loop formation by RNA-guided DNA-targeting modules. TniQ residues, proximal to CRISPR RNA (crRNA), are required for recognizing different crRNA categories, revealing an unappreciated role of TniQ to direct transposition into different classes of crRNA targets. To investigate adaptations allowing CAST elements to utilize attachment sites inaccessible to CRISPR-Cas surveillance complexes, we compared and contrasted PAM sequence requirements in both I-F3b CAST and I-F1 CRISPR-Cas systems. We identify specific amino acids that enable a wider range of PAM sequences to be accommodated in I-F3b CAST elements compared with I-F1 CRISPR-Cas, enabling CAST elements to access attachment sites as sequences drift and evade host surveillance. Together, this evidence points to the central role of TniQ in facilitating the acquisition of CRISPR effector complexes for RNA-guided DNA transposition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26348.map.gz emd_26348.map.gz | 7.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26348-v30.xml emd-26348-v30.xml emd-26348.xml emd-26348.xml | 31.7 KB 31.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26348_fsc.xml emd_26348_fsc.xml | 15 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26348.png emd_26348.png | 81.8 KB | ||
| Filedesc metadata |  emd-26348.cif.gz emd-26348.cif.gz | 7.7 KB | ||
| その他 |  emd_26348_additional_1.map.gz emd_26348_additional_1.map.gz emd_26348_additional_2.map.gz emd_26348_additional_2.map.gz emd_26348_additional_3.map.gz emd_26348_additional_3.map.gz emd_26348_additional_4.map.gz emd_26348_additional_4.map.gz emd_26348_half_map_1.map.gz emd_26348_half_map_1.map.gz emd_26348_half_map_2.map.gz emd_26348_half_map_2.map.gz | 225.1 MB 51.6 MB 51.9 MB 51.9 MB 225.5 MB 225.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26348 http://ftp.pdbj.org/pub/emdb/structures/EMD-26348 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26348 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26348 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26348_validation.pdf.gz emd_26348_validation.pdf.gz | 881.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26348_full_validation.pdf.gz emd_26348_full_validation.pdf.gz | 880.8 KB | 表示 | |
| XML形式データ |  emd_26348_validation.xml.gz emd_26348_validation.xml.gz | 21.9 KB | 表示 | |
| CIF形式データ |  emd_26348_validation.cif.gz emd_26348_validation.cif.gz | 28.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26348 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26348 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26348 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26348 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26348.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26348.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | combined map of local-resolution filtered maps from full refinement and focused refinement | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.873 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: unsharpened map of the full structure
| ファイル | emd_26348_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
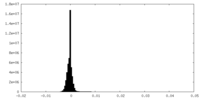
| 注釈 | unsharpened map of the full structure | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map from focused refinement
| ファイル | emd_26348_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
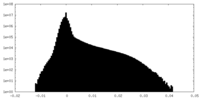
| 注釈 | Unsharpened map from focused refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: halfmap 1 from focused refinement
| ファイル | emd_26348_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
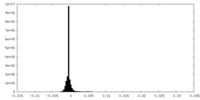
| 注釈 | halfmap 1 from focused refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: halfmap 2 from focused refinement
| ファイル | emd_26348_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
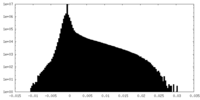
| 注釈 | halfmap 2 from focused refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 2 of the full structure
| ファイル | emd_26348_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 2 of the full structure | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Halfmap 1 of the full structure
| ファイル | emd_26348_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap 1 of the full structure | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : I-F3b Cascade-TniQ Full R-loop complex
| 全体 | 名称: I-F3b Cascade-TniQ Full R-loop complex |
|---|---|
| 要素 |
|
-超分子 #1: I-F3b Cascade-TniQ Full R-loop complex
| 超分子 | 名称: I-F3b Cascade-TniQ Full R-loop complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 450 KDa |
-分子 #1: crRNA
| 分子 | 名称: crRNA / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 19.399604 KDa |
| 配列 | 文字列: CCAAGAAAAG GACUGGAAGA AAUCAUCCAA GUUGGGGACU AUUUUCUGCC GUAUAGGCAG |
-分子 #2: Target strand DNA
| 分子 | 名称: Target strand DNA / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 35.61368 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(DG)(DC)(DT)(DG)(DG)(DC)(DG) (DA)(DA)(DC)(DG)(DA)(DG)(DC)(DG)(DC)(DA) (DA)(DG)(DG)(DT)(DG)(DG)(DT)(DG)(DG) (DC)(DC)(DC)(DC)(DA)(DT)(DC)(DA)(DG)(DC) (DC) (DA)(DC)(DA)(DT)(DC) ...文字列: (DC)(DT)(DG)(DG)(DC)(DT)(DG)(DG)(DC)(DG) (DA)(DA)(DC)(DG)(DA)(DG)(DC)(DG)(DC)(DA) (DA)(DG)(DG)(DT)(DG)(DG)(DT)(DG)(DG) (DC)(DC)(DC)(DC)(DA)(DT)(DC)(DA)(DG)(DC) (DC) (DA)(DC)(DA)(DT)(DC)(DC)(DC)(DG) (DG)(DC)(DA)(DC)(DT)(DC)(DG)(DA)(DA)(DG) (DT)(DC) (DC)(DC)(DC)(DA)(DA)(DC)(DT) (DT)(DG)(DG)(DA)(DT)(DG)(DA)(DT)(DT)(DT) (DC)(DT)(DT) (DC)(DC)(DA)(DG)(DT)(DC) (DC)(DT)(DG)(DG)(DT)(DA)(DA)(DG)(DC)(DA) (DC)(DC)(DC)(DG) (DA)(DA)(DT)(DC)(DA) (DT)(DC)(DC)(DT)(DC)(DT)(DT)(DG)(DC)(DG) (DG) |
-分子 #3: Non-target strand DNA
| 分子 | 名称: Non-target strand DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 35.737723 KDa |
| 配列 | 文字列: (DC)(DC)(DG)(DC)(DA)(DA)(DG)(DA)(DG)(DG) (DA)(DT)(DG)(DA)(DT)(DT)(DC)(DG)(DG)(DG) (DT)(DG)(DC)(DT)(DT)(DA)(DC)(DC)(DT) (DC)(DC)(DT)(DG)(DA)(DC)(DC)(DT)(DT)(DC) (DT) (DT)(DT)(DA)(DG)(DT) ...文字列: (DC)(DC)(DG)(DC)(DA)(DA)(DG)(DA)(DG)(DG) (DA)(DT)(DG)(DA)(DT)(DT)(DC)(DG)(DG)(DG) (DT)(DG)(DC)(DT)(DT)(DA)(DC)(DC)(DT) (DC)(DC)(DT)(DG)(DA)(DC)(DC)(DT)(DT)(DC) (DT) (DT)(DT)(DA)(DG)(DT)(DA)(DG)(DG) (DT)(DT)(DC)(DA)(DA)(DC)(DC)(DC)(DC)(DT) (DG)(DA) (DT)(DC)(DG)(DA)(DG)(DT)(DG) (DC)(DC)(DG)(DG)(DG)(DA)(DT)(DG)(DT)(DG) (DG)(DC)(DT) (DG)(DA)(DT)(DG)(DG)(DG) (DG)(DC)(DC)(DA)(DC)(DC)(DA)(DC)(DC)(DT) (DT)(DG)(DC)(DG) (DC)(DT)(DC)(DG)(DT) (DT)(DC)(DG)(DC)(DC)(DA)(DG)(DC)(DC)(DA) (DG) |
-分子 #4: Cas8/5
| 分子 | 名称: Cas8/5 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 78.728367 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVTIMHIEEL LDIEDHGERD RQLRRYLAPY SAEIGVDGAE KMALVVLLNL TLKRDRVESL CDEGLARQLL SDEGHITNCL HTVRWLHTH NLKYPDARVS GERLIINAPP LIPGVISSAG LPMRMGWAHD SSDINLAKLF GTSFRYRDDS TNLALQLVAR S KTWEQALI ...文字列: MVTIMHIEEL LDIEDHGERD RQLRRYLAPY SAEIGVDGAE KMALVVLLNL TLKRDRVESL CDEGLARQLL SDEGHITNCL HTVRWLHTH NLKYPDARVS GERLIINAPP LIPGVISSAG LPMRMGWAHD SSDINLAKLF GTSFRYRDDS TNLALQLVAR S KTWEQALI GLGLTQQQLD IWCQLLASNL ENNTFPTVVS PFSKQVRFLY QGNYCVVTPV VSHALLAQLQ NVVHEKKLQC TY IHHDHPA SVGSLVGALG GKVAVLDYPP PVSPDKARSF SQARKHRLAN GQSLFDRSVF NDHVFIDALK HVISRPGLTR KQQ RQLRLS ALRYLRRQLA IWLGPIIEWR DEIVSSGRGE PGNLPSGGLE LELITQPKKM LPELMLQVAG RFHLELQNHS AGRR FAFHP ALMAPIKSQI LWLLRQLADD EEKDEPHPPT SCYYLHLSGL TVYDASALAN PYLCGIPSLS ALAGFCHDYE RRLQS LIGQ SVYFRGLAWY LGRYSLVTGK HLPEPSKSAD PKSVSAIRRP GLLDGRYCDL GMDLIIEVHI PTGGSLPFTT CLDLLR VAL PARFAGGCLH PPSLYEEYNW CTVYQDKSTL FTVLSRLPRY GCWIYPSDAD LRSFEELSEA LALDRRLRPV ATGFVFL EE PVERAGSIEG QHVYAESAIG TALCINPVEM RLAGKKRFFG AGFWQLNDAK GAILMNGSAN TG |
-分子 #5: Cas7
| 分子 | 名称: Cas7 / タイプ: protein_or_peptide / ID: 5 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 39.105172 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MELCTHLSYS RSLSPGKAVF FYKTAESDFV PLRIEVAKIS GQKCGYTEGF DANLKPKNIE RYELAYSNPQ TIEACYVPPN VDELYCRFS LRVEANSMRP YVCSNPDVLR VMIGLAQAYQ RLGGYNELAR RYSANVLRGI WLWRNQYTQG TKIEIKTSLG S TYHIPDAR ...文字列: MELCTHLSYS RSLSPGKAVF FYKTAESDFV PLRIEVAKIS GQKCGYTEGF DANLKPKNIE RYELAYSNPQ TIEACYVPPN VDELYCRFS LRVEANSMRP YVCSNPDVLR VMIGLAQAYQ RLGGYNELAR RYSANVLRGI WLWRNQYTQG TKIEIKTSLG S TYHIPDAR RLSWSGDWPE LEQKQLEQLT SEMAKALSQP DIFWFADVTA SLKTGFCQEI FPSQKFTERP DDHSVASRQL AT VECSDGQ LAACINPQKI GAALQKIDDW WANDADLPLR VHEYGANHEA LTALRHPATG QDFYHLLTKA EQFVTVLESS EGG GVELPG EVHYLMAVLV KGGLFQKGKG R |
-分子 #6: Cas6
| 分子 | 名称: Cas6 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 23.640832 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTENRYFFAI RYLSDDVDCG LLAGRCISIL HGFRQAHPGI QIGVAFPEWS DRDLGRSIAF VSTNKSLLER FRERSYFQVM QADNFFALS LVLEVPDTCQ NVRFIRNQNL AKLFVGERRR RLARAKRRAK ARGEAFQPHM PDETKVVGVF HSVFMQSASS G QSYILHIQ ...文字列: MTENRYFFAI RYLSDDVDCG LLAGRCISIL HGFRQAHPGI QIGVAFPEWS DRDLGRSIAF VSTNKSLLER FRERSYFQVM QADNFFALS LVLEVPDTCQ NVRFIRNQNL AKLFVGERRR RLARAKRRAK ARGEAFQPHM PDETKVVGVF HSVFMQSASS G QSYILHIQ KHRYERSEDS GYSSYGLASN DLYTGYVPDL GAIFSTLF |
-分子 #7: TniQ
| 分子 | 名称: TniQ / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aeromonas salmonicida (バクテリア) Aeromonas salmonicida (バクテリア) |
| 分子量 | 理論値: 46.366637 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHLLVRPEPF ADEALESYFL RLSQENGFER YRIFSGSVQD WLHTTDHAAA GAFPLELSRL NIFHASRSSG LRVRALQLVD RLTDGAPFR LLQLALCHSA ISFGNHYKAV HRSGVDIPLS FIRVHQIPCC PDCLRESAYV RQCWHFKPYV GCHRHGGRLI Y SCPACGES ...文字列: MHLLVRPEPF ADEALESYFL RLSQENGFER YRIFSGSVQD WLHTTDHAAA GAFPLELSRL NIFHASRSSG LRVRALQLVD RLTDGAPFR LLQLALCHSA ISFGNHYKAV HRSGVDIPLS FIRVHQIPCC PDCLRESAYV RQCWHFKPYV GCHRHGGRLI Y SCPACGES LNYLASESIN HCQCGFDLRT ASTVPAQPDE IQLSALAYGC SFESSNPLLA IGSLSARFGA LYWYQQRYLS DH EAVRDDR ALTKAIGHFT AWPDAFWREL QQMVDDALVR QTKPLNHTDF VDVFGSVVAD CRQIPMRNTG QNFILKNLIG FLT DLVARH PQCRVANVGD LLLSAVDAAT LLSTSVEQVR RLHHEGFLPL SIRPASRNTV SPHRAVFHLR HVVELRQARM QSHH DHSST YLPAW |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)