+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SthK closed state, cAMP-bound in the presence of POPA | ||||||||||||
 マップデータ マップデータ | unsharpened reconstruction | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | cyclic nucleotide-gated channel / lipid modulation / pacemaker channel / TRANSPORT PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intracellular cyclic nucleotide activated cation channel complex / intracellularly cGMP-activated cation channel activity / intracellularly cAMP-activated cation channel activity / cGMP binding / protein-containing complex binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Spirochaeta thermophila (strain ATCC 700085 / DSM 6578 / Z-1203) (バクテリア) / Spirochaeta thermophila (strain ATCC 700085 / DSM 6578 / Z-1203) (バクテリア) /   Spirochaeta thermophila DSM 6578 (バクテリア) Spirochaeta thermophila DSM 6578 (バクテリア) | ||||||||||||
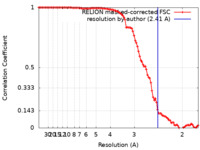
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.41 Å | ||||||||||||
 データ登録者 データ登録者 | Schmidpeter PA / Nimigean CM | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Anionic lipids unlock the gates of select ion channels in the pacemaker family. 著者: Philipp A M Schmidpeter / Di Wu / Jan Rheinberger / Paul M Riegelhaupt / Haiping Tang / Carol V Robinson / Crina M Nimigean /    要旨: Lipids play important roles in regulating membrane protein function, but the molecular mechanisms used are elusive. Here we investigated how anionic lipids modulate SthK, a bacterial pacemaker ...Lipids play important roles in regulating membrane protein function, but the molecular mechanisms used are elusive. Here we investigated how anionic lipids modulate SthK, a bacterial pacemaker channel homolog, and HCN2, whose activity contributes to pacemaking in the heart and brain. Using SthK allowed the reconstitution of purified channels in controlled lipid compositions for functional and structural assays that are not available for the eukaryotic channels. We identified anionic lipids bound tightly to SthK and their exact binding locations and determined that they potentiate channel activity. Cryo-EM structures in the most potentiating lipids revealed an open state and identified a nonannular lipid bound with its headgroup near an intersubunit salt bridge that clamps the intracellular channel gate shut. Breaking this conserved salt bridge abolished lipid modulation in SthK and eukaryotic HCN2 channels, indicating that anionic membrane lipids facilitate channel opening by destabilizing these interactions. Our findings underline the importance of state-dependent protein-lipid interactions. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25916.map.gz emd_25916.map.gz | 62.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25916-v30.xml emd-25916-v30.xml emd-25916.xml emd-25916.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25916_fsc.xml emd_25916_fsc.xml | 10.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25916.png emd_25916.png | 137.6 KB | ||
| マスクデータ |  emd_25916_msk_1.map emd_25916_msk_1.map | 87.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25916.cif.gz emd-25916.cif.gz | 6.8 KB | ||
| その他 |  emd_25916_half_map_1.map.gz emd_25916_half_map_1.map.gz emd_25916_half_map_2.map.gz emd_25916_half_map_2.map.gz | 63.8 MB 63.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25916 http://ftp.pdbj.org/pub/emdb/structures/EMD-25916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25916 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25916_validation.pdf.gz emd_25916_validation.pdf.gz | 735.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25916_full_validation.pdf.gz emd_25916_full_validation.pdf.gz | 734.9 KB | 表示 | |
| XML形式データ |  emd_25916_validation.xml.gz emd_25916_validation.xml.gz | 16.7 KB | 表示 | |
| CIF形式データ |  emd_25916_validation.cif.gz emd_25916_validation.cif.gz | 22.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25916 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25916 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25916 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25916 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25916.map.gz / 形式: CCP4 / 大きさ: 87.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25916.map.gz / 形式: CCP4 / 大きさ: 87.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened reconstruction | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25916_msk_1.map emd_25916_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
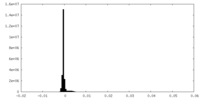
| 投影像・断面図 |
| ||||||||||||
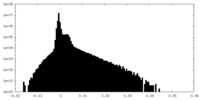
| 密度ヒストグラム |
-ハーフマップ: half map 1, unfiltered
| ファイル | emd_25916_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1, unfiltered | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2, unfiltered
| ファイル | emd_25916_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2, unfiltered | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : closed conformation of SthK bound to cAMP
| 全体 | 名称: closed conformation of SthK bound to cAMP |
|---|---|
| 要素 |
|
-超分子 #1: closed conformation of SthK bound to cAMP
| 超分子 | 名称: closed conformation of SthK bound to cAMP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: SthK was reconstituted into nanodiscs containing POPA lipids. |
|---|---|
| 由来(天然) | 生物種:   Spirochaeta thermophila (strain ATCC 700085 / DSM 6578 / Z-1203) (バクテリア) Spirochaeta thermophila (strain ATCC 700085 / DSM 6578 / Z-1203) (バクテリア) |
-分子 #1: Putative transcriptional regulator, Crp/Fnr family
| 分子 | 名称: Putative transcriptional regulator, Crp/Fnr family / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Spirochaeta thermophila DSM 6578 (バクテリア) Spirochaeta thermophila DSM 6578 (バクテリア)株: ATCC 700085 / DSM 6578 / Z-1203 |
| 分子量 | 理論値: 51.118574 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKDIGINSD PNSSSVDKLM KSSGVSNPTY TLVWKVWILA VTLYYAIRIP LTLVFPSLFS PLLPLDILAS LALIADIPLD LAFESRRTS GRKPTLLAPS RLPDLLAALP LDLLVFALHL PSPLSLLSLV RLLKLISVQR SATRILSYRI NPALLRLLSL V GFILLAAH ...文字列: MAKDIGINSD PNSSSVDKLM KSSGVSNPTY TLVWKVWILA VTLYYAIRIP LTLVFPSLFS PLLPLDILAS LALIADIPLD LAFESRRTS GRKPTLLAPS RLPDLLAALP LDLLVFALHL PSPLSLLSLV RLLKLISVQR SATRILSYRI NPALLRLLSL V GFILLAAH GIACGWMSLQ PPSENPAGTR YLSAFYWTIT TLTTIGYGDI TPSTPTQTVY TIVIELLGAA MYGLVIGNIA SL VSKLDAA KLLHRERVER VTAFLSYKRI SPELQRRIIE YFDYLWETRR GYEEREVLKE LPHPLRLAVA MEIHGDVIEK VPL FKGAGE EFIRDIILHL EPVIYGPGEY IIRAGEMGSD VYFINRGSVE VLSADEKTRY AILSEGQFFG EMALILRAPR TATV RARAF CDLYRLDKET FDRILSRYPE IAAQIQELAV RRKELESSGL VPRGSVKHHH H UniProtKB: Transcriptional regulator, Crp/Fnr family |
-分子 #2: ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE
| 分子 | 名称: ADENOSINE-3',5'-CYCLIC-MONOPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: CMP |
|---|---|
| 分子量 | 理論値: 329.206 Da |
| Chemical component information |  ChemComp-CMP: |
-分子 #3: (2R)-1-(hexadecanoyloxy)-3-(phosphonooxy)propan-2-yl (9Z)-octadec...
| 分子 | 名称: (2R)-1-(hexadecanoyloxy)-3-(phosphonooxy)propan-2-yl (9Z)-octadec-9-enoate タイプ: ligand / ID: 3 / コピー数: 28 / 式: D21 |
|---|---|
| 分子量 | 理論値: 674.929 Da |
| Chemical component information |  ChemComp-D21: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 80 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298.15 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | WT SthK in MSP1E3 nanodiscs composed of 3:1 DOPC:POPA |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 5684 / 平均露光時間: 2.6 sec. / 平均電子線量: 50.812 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X