+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the HCV IRES bound to the 40S ribosomal subunit, head opening. Structure 8(delta dII) | |||||||||||||||
 マップデータ マップデータ | Post processed map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | HCV / IRES / 40S / RIBOSOME | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribosomal subunit / laminin receptor activity / ubiquitin ligase inhibitor activity / positive regulation of signal transduction by p53 class mediator / 90S preribosome / phagocytic cup / ribosomal small subunit export from nucleus / rough endoplasmic reticulum / laminin binding / translation regulator activity ...ribosomal subunit / laminin receptor activity / ubiquitin ligase inhibitor activity / positive regulation of signal transduction by p53 class mediator / 90S preribosome / phagocytic cup / ribosomal small subunit export from nucleus / rough endoplasmic reticulum / laminin binding / translation regulator activity / gastrulation / MDM2/MDM4 family protein binding / cytosolic ribosome / class I DNA-(apurinic or apyrimidinic site) endonuclease activity / DNA-(apurinic or apyrimidinic site) lyase / maturation of LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / positive regulation of apoptotic signaling pathway / maturation of SSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / maturation of SSU-rRNA / small-subunit processome / spindle / rRNA processing / rhythmic process / positive regulation of canonical Wnt signaling pathway / regulation of translation / ribosome binding / virus receptor activity / ribosomal small subunit biogenesis / ribosomal small subunit assembly / small ribosomal subunit / small ribosomal subunit rRNA binding / cytosolic small ribosomal subunit / perikaryon / cytosolic large ribosomal subunit / cytoplasmic translation / cell differentiation / mitochondrial inner membrane / postsynaptic density / rRNA binding / structural constituent of ribosome / ribosome / translation / ribonucleoprotein complex / cell division / DNA repair / mRNA binding / apoptotic process / synapse / dendrite / centrosome / nucleolus / perinuclear region of cytoplasm / Golgi apparatus / DNA binding / RNA binding / zinc ion binding / nucleus / membrane / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Hepatitis C virus (isolate 1) (C型肝炎ウイルス) Hepatitis C virus (isolate 1) (C型肝炎ウイルス) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Brown ZP / Abaeva IS / De S / Hellen CUT / Pestova TV / Frank J | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Molecular architecture of 40S initiation complexes on the Hepatitis C virus IRES: from ribosomal attachment to eIF5B-mediated reorientation of initiator tRNA 著者: Brown ZP / Abaeva IS / De S / Hellen CUT / Pestova TV / Frank J | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25534.map.gz emd_25534.map.gz | 228.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25534-v30.xml emd-25534-v30.xml emd-25534.xml emd-25534.xml | 59.8 KB 59.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25534_fsc.xml emd_25534_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25534.png emd_25534.png | 37.1 KB | ||
| マスクデータ |  emd_25534_msk_1.map emd_25534_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25534.cif.gz emd-25534.cif.gz | 12.7 KB | ||
| その他 |  emd_25534_additional_1.map.gz emd_25534_additional_1.map.gz emd_25534_additional_2.map.gz emd_25534_additional_2.map.gz emd_25534_additional_3.map.gz emd_25534_additional_3.map.gz emd_25534_half_map_1.map.gz emd_25534_half_map_1.map.gz emd_25534_half_map_2.map.gz emd_25534_half_map_2.map.gz | 122.9 MB 143.3 MB 192.4 MB 193.7 MB 193.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25534 http://ftp.pdbj.org/pub/emdb/structures/EMD-25534 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25534 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25534 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25534_validation.pdf.gz emd_25534_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25534_full_validation.pdf.gz emd_25534_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_25534_validation.xml.gz emd_25534_validation.xml.gz | 20.8 KB | 表示 | |
| CIF形式データ |  emd_25534_validation.cif.gz emd_25534_validation.cif.gz | 27.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25534 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25534 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25534 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25534 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7synMC  7sygC 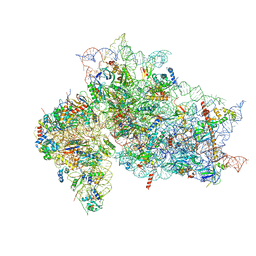 7syhC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25534.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25534.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post processed map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : 40S ribosomal small subunit with HCV IRES
+超分子 #1: 40S ribosomal small subunit with HCV IRES
+分子 #1: 18S rRNA
+分子 #36: HCV IRES
+分子 #2: uS2 (SA)
+分子 #3: eS1
+分子 #4: 40S ribosomal protein S2
+分子 #5: uS3
+分子 #6: eS4 (S4 X isoform)
+分子 #7: uS7
+分子 #8: eS6
+分子 #9: eS7
+分子 #10: eS8
+分子 #11: uS4
+分子 #12: eS10
+分子 #13: uS17
+分子 #14: eS12
+分子 #15: uS15
+分子 #16: uS11
+分子 #17: uS19
+分子 #18: uS9
+分子 #19: eS17
+分子 #20: uS13
+分子 #21: eS19
+分子 #22: uS10
+分子 #23: eS21
+分子 #24: uS8
+分子 #25: uS12
+分子 #26: eS24
+分子 #27: eS25
+分子 #28: eS26
+分子 #29: eS27
+分子 #30: eS28
+分子 #31: uS14
+分子 #32: eS30
+分子 #33: eS31
+分子 #34: Receptor for Activated C Kinase 1 (RACK1)
+分子 #35: eL41
+分子 #37: ZINC ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.000075 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: OTHER / 詳細: H2/O2 mixture for 25 seconds at 25W power |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: 4 second blot time, force 3. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 4.0 sec. / 平均電子線量: 70.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 144252 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) / 詳細: Relion 3 |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー















