+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human Kv1.3 with Fab-ShK fusion | |||||||||
 マップデータ マップデータ | Human Kv1.3 with Fab-ShK | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報voltage-gated monoatomic ion channel activity / corpus callosum development / delayed rectifier potassium channel activity / Voltage gated Potassium channels / outward rectifier potassium channel activity / optic nerve development / action potential / calyx of Held / voltage-gated potassium channel activity / voltage-gated potassium channel complex ...voltage-gated monoatomic ion channel activity / corpus callosum development / delayed rectifier potassium channel activity / Voltage gated Potassium channels / outward rectifier potassium channel activity / optic nerve development / action potential / calyx of Held / voltage-gated potassium channel activity / voltage-gated potassium channel complex / potassium ion transmembrane transport / bioluminescence / generation of precursor metabolites and energy / potassium ion transport / protein homooligomerization / presynaptic membrane / postsynaptic membrane / membrane raft / axon / glutamatergic synapse / perinuclear region of cytoplasm / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
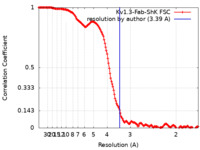
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.39 Å | |||||||||
 データ登録者 データ登録者 | Meyerson JR / Selvakumar P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structures of the T cell potassium channel Kv1.3 with immunoglobulin modulators. 著者: Purushotham Selvakumar / Ana I Fernández-Mariño / Nandish Khanra / Changhao He / Alice J Paquette / Bing Wang / Ruiqi Huang / Vaughn V Smider / William J Rice / Kenton J Swartz / Joel R Meyerson /  要旨: The Kv1.3 potassium channel is expressed abundantly on activated T cells and mediates the cellular immune response. This role has made the channel a target for therapeutic immunomodulation to block ...The Kv1.3 potassium channel is expressed abundantly on activated T cells and mediates the cellular immune response. This role has made the channel a target for therapeutic immunomodulation to block its activity and suppress T cell activation. Here, we report structures of human Kv1.3 alone, with a nanobody inhibitor, and with an antibody-toxin fusion blocker. Rather than block the channel directly, four copies of the nanobody bind the tetramer's voltage sensing domains and the pore domain to induce an inactive pore conformation. In contrast, the antibody-toxin fusion docks its toxin domain at the extracellular mouth of the channel to insert a critical lysine into the pore. The lysine stabilizes an active conformation of the pore yet blocks ion permeation. This study visualizes Kv1.3 pore dynamics, defines two distinct mechanisms to suppress Kv1.3 channel activity with exogenous inhibitors, and provides a framework to aid development of emerging T cell immunotherapies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25414.map.gz emd_25414.map.gz | 307.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25414-v30.xml emd-25414-v30.xml emd-25414.xml emd-25414.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25414_fsc.xml emd_25414_fsc.xml | 15.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25414.png emd_25414.png | 94.5 KB | ||
| Filedesc metadata |  emd-25414.cif.gz emd-25414.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25414 http://ftp.pdbj.org/pub/emdb/structures/EMD-25414 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25414 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25414 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25414_validation.pdf.gz emd_25414_validation.pdf.gz | 520.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25414_full_validation.pdf.gz emd_25414_full_validation.pdf.gz | 519.6 KB | 表示 | |
| XML形式データ |  emd_25414_validation.xml.gz emd_25414_validation.xml.gz | 14.6 KB | 表示 | |
| CIF形式データ |  emd_25414_validation.cif.gz emd_25414_validation.cif.gz | 20 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25414 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25414 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25414 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25414 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ssvMC  7ssxC  7ssyC  7sszC  8dflC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-11081 (タイトル: Human Kv1.3 with a Fab-ShK fusion / Data size: 2.0 TB EMPIAR-11081 (タイトル: Human Kv1.3 with a Fab-ShK fusion / Data size: 2.0 TBData #1: Human Kv1.3 ShK-Fab dataset [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25414.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25414.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human Kv1.3 with Fab-ShK | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.852 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Kv1.3 with Fab-ShK
| 全体 | 名称: Kv1.3 with Fab-ShK |
|---|---|
| 要素 |
|
-超分子 #1: Kv1.3 with Fab-ShK
| 超分子 | 名称: Kv1.3 with Fab-ShK / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Potassium voltage-gated channel subfamily A member 3,Green fluore...
| 分子 | 名称: Potassium voltage-gated channel subfamily A member 3,Green fluorescent protein fusion タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 95.0185 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDERLSLLRS PPPPSARHRA HPPQRPASSG GAHTLVNHGY AEPAAGRELP PDMTVVPGDH LLEPEVADGG GAPPQGGCGG GGCDRYEPL PPSLPAAGEQ DCCGERVVIN ISGLRFETQL KTLCQFPETL LGDPKRRMRY FDPLRNEYFF DRNRPSFDAI L YYYQSGGR ...文字列: MDERLSLLRS PPPPSARHRA HPPQRPASSG GAHTLVNHGY AEPAAGRELP PDMTVVPGDH LLEPEVADGG GAPPQGGCGG GGCDRYEPL PPSLPAAGEQ DCCGERVVIN ISGLRFETQL KTLCQFPETL LGDPKRRMRY FDPLRNEYFF DRNRPSFDAI L YYYQSGGR IRRPVNVPID IFSEEIRFYQ LGEEAMEKFR EDEGFLREEE RPLPRRDFQR QVWLLFEYPE SSGPARGIAI VS VLVILIS IVIFCLETLP EFRDEKDYPA STSQDSFEAA GNSTSGSRAG ASSFSDPFFV VETLCIIWFS FELLVRFFAC PSK ATFSRN IMNLIDIVAI IPYFITLGTE LAERQGNGQQ AMSLAILRVI RLVRVFRIFK LSRHSKGLQI LGQTLKASMR ELGL LIFFL FIGVILFSSA VYFAEADDPT SGFSSIPDAF WWAVVTMTTV GYGDMHPVTI GGKIVGSLCA IAGVLTIALP VPVIV SNFN YFYHRETEGE EQSQYMHVGS CQHLSSSAEE LRKARSNSTL SKSEYMVIEE GGMNHSAFPQ TPFKTGNSTA TCTTNN NPN SCVNIKKIFT DVSLEVLFQG PAAAMVSKGE ELFTGVVPIL VELDGDVNGH KFSVSGEGEG DATYGKLTLK LICTTGK LP VPWPTLVTTL GYGLQCFARY PDHMKQHDFF KSAMPEGYVQ ERTIFFKDDG NYKTRAEVKF EGDTLVNRIE LKGIDFKE D GNILGHKLEY NYNSHNVYIT ADKQKNGIKA NFKIRHNIED GGVQLADHYQ QNTPIGDGPV LLPDNHYLSY QSKLSKDPN EKRDHMVLLE FVTAAGITLG MDELYKSAWS HPQFEKGGGS GGGSGGGSWS HPQFEK UniProtKB: Potassium voltage-gated channel subfamily A member 3, Green fluorescent protein |
-分子 #2: Fab-ShK fusion, heavy chain
| 分子 | 名称: Fab-ShK fusion, heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 29.417229 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLREWGAG LLKPSETLSL TCAVYGGSFS DKYWSWIRQP PGKGLEWIGS INHSGSTNYN PSLKSRVTIS VDTSKNQFSL KLSSVTAAD TAVYYCTSVH QETKKYQSRS CIDTIPKSRC TAFQCKHSMK YRLSFCRKTC GTCSYTYNYE WHVDVWGQGL L VTVSSAST ...文字列: QVQLREWGAG LLKPSETLSL TCAVYGGSFS DKYWSWIRQP PGKGLEWIGS INHSGSTNYN PSLKSRVTIS VDTSKNQFSL KLSSVTAAD TAVYYCTSVH QETKKYQSRS CIDTIPKSRC TAFQCKHSMK YRLSFCRKTC GTCSYTYNYE WHVDVWGQGL L VTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SS LGTQTYI CNVNHKPSNT KVDKKVEPKS CDK |
-分子 #3: Fab-ShK fusion, light chain
| 分子 | 名称: Fab-ShK fusion, light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 22.524752 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QAVLNQPSSV SGSLGQKVTI SCSGSSSNIG NNYVSWYQQL PGTAPKLLIY GDTKRPSGIP DRFSGSKSGT SATLGITGLQ TGDEADYYC ASAEDSSSNA VFGSGTTLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP ...文字列: QAVLNQPSSV SGSLGQKVTI SCSGSSSNIG NNYVSWYQQL PGTAPKLLIY GDTKRPSGIP DRFSGSKSGT SATLGITGLQ TGDEADYYC ASAEDSSSNA VFGSGTTLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS |
-分子 #4: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 4 / コピー数: 3 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)