+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cas9:sgRNA:DNA (S. pyogenes) with 0 RNA:DNA base pairs, open-protein/linear-DNA conformation | |||||||||
 マップデータ マップデータ | LocSpiral-sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / 3'-5' exonuclease activity / DNA endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus pyogenes serotype M1 (化膿レンサ球菌) / synthetic construct (人工物) / Streptococcus pyogenes serotype M1 (化膿レンサ球菌) / synthetic construct (人工物) /  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) | |||||||||
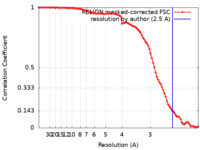
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Cofsky JC / Soczek KM / Knott GJ / Nogales E / Doudna JA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: CRISPR-Cas9 bends and twists DNA to read its sequence. 著者: Joshua C Cofsky / Katarzyna M Soczek / Gavin J Knott / Eva Nogales / Jennifer A Doudna /   要旨: In bacterial defense and genome editing applications, the CRISPR-associated protein Cas9 searches millions of DNA base pairs to locate a 20-nucleotide, guide RNA-complementary target sequence that ...In bacterial defense and genome editing applications, the CRISPR-associated protein Cas9 searches millions of DNA base pairs to locate a 20-nucleotide, guide RNA-complementary target sequence that abuts a protospacer-adjacent motif (PAM). Target capture requires Cas9 to unwind DNA at candidate sequences using an unknown ATP-independent mechanism. Here we show that Cas9 sharply bends and undertwists DNA on PAM binding, thereby flipping DNA nucleotides out of the duplex and toward the guide RNA for sequence interrogation. Cryogenic-electron microscopy (cryo-EM) structures of Cas9-RNA-DNA complexes trapped at different states of the interrogation pathway, together with solution conformational probing, reveal that global protein rearrangement accompanies formation of an unstacked DNA hinge. Bend-induced base flipping explains how Cas9 'reads' snippets of DNA to locate target sites within a vast excess of nontarget DNA, a process crucial to both bacterial antiviral immunity and genome editing. This mechanism establishes a physical solution to the problem of complementarity-guided DNA search and shows how interrogation speed and local DNA geometry may influence genome editing efficiency. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24823.map.gz emd_24823.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24823-v30.xml emd-24823-v30.xml emd-24823.xml emd-24823.xml | 25.2 KB 25.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24823_fsc.xml emd_24823_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24823.png emd_24823.png | 106.3 KB | ||
| マスクデータ |  emd_24823_msk_1.map emd_24823_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_24823_additional_1.map.gz emd_24823_additional_1.map.gz emd_24823_additional_2.map.gz emd_24823_additional_2.map.gz emd_24823_half_map_1.map.gz emd_24823_half_map_1.map.gz emd_24823_half_map_2.map.gz emd_24823_half_map_2.map.gz | 30.7 MB 4.1 MB 59.1 MB 59.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24823 http://ftp.pdbj.org/pub/emdb/structures/EMD-24823 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24823 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24823 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24823_validation.pdf.gz emd_24823_validation.pdf.gz | 656.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24823_full_validation.pdf.gz emd_24823_full_validation.pdf.gz | 655.8 KB | 表示 | |
| XML形式データ |  emd_24823_validation.xml.gz emd_24823_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_24823_validation.cif.gz emd_24823_validation.cif.gz | 21.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24823 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24823 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24823 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24823 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24823.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24823.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocSpiral-sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_24823_msk_1.map emd_24823_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: unsharpened map from cryoSPARC
| ファイル | emd_24823_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map from cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: post-processed map from RELION
| ファイル | emd_24823_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post-processed map from RELION | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A from cryoSPARC
| ファイル | emd_24823_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A from cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B from cryoSPARC
| ファイル | emd_24823_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B from cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas9 bound to sgRNA and DNA with 0bp complementarity between RNA ...
| 全体 | 名称: Cas9 bound to sgRNA and DNA with 0bp complementarity between RNA and DNA |
|---|---|
| 要素 |
|
-超分子 #1: Cas9 bound to sgRNA and DNA with 0bp complementarity between RNA ...
| 超分子 | 名称: Cas9 bound to sgRNA and DNA with 0bp complementarity between RNA and DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes serotype M1 (化膿レンサ球菌) Streptococcus pyogenes serotype M1 (化膿レンサ球菌) |
-分子 #1: Non-target DNA strand
| 分子 | 名称: Non-target DNA strand / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 9.314044 KDa |
| 配列 | 文字列: (DG)(DA)(DC)(DG)(DC)(DA)(DT)(DA)(DA)(DA) (DG)(DA)(DT)(DG)(DA)(DG)(DA)(DC)(DA)(DA) (DT)(DG)(DG)(DC)(DG)(DA)(DT)(DT)(DA) (DC) |
-分子 #4: Target DNA strand
| 分子 | 名称: Target DNA strand / タイプ: dna / ID: 4 詳細: The 5'-most deoxycytidine is actually N4-cystamine 2'-deoxycytidine コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 9.190992 KDa |
| 配列 | 文字列: (DG)(DT)(DA)(DA)(DT)(2YR)(DG)(DC)(DC)(DA) (DT)(DT)(DG)(DT)(DC)(DT)(DC)(DA)(DT) (DC)(DT)(DT)(DT)(DA)(DT)(DG)(DC)(DG)(DT) (DC) |
-分子 #2: CRISPR-associated endonuclease Cas9/Csn1
| 分子 | 名称: CRISPR-associated endonuclease Cas9/Csn1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes serotype M1 (化膿レンサ球菌) Streptococcus pyogenes serotype M1 (化膿レンサ球菌) |
| 分子量 | 理論値: 158.974125 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMDKKYSI GLDIGTNSVG WAVITDEYKV PSKKFKVLGN TDRHSIKKNL IGALLFDSGE TAEATRLKRT ARRRYTRRKN RICYLQEIF SNEMAKVDDS FFHRLEESFL VEEDKKHERH PIFGNIVDEV AYHEKYPTIY HLRKKLVDST DKADLRLIYL A LAHMIKFR ...文字列: SNAMDKKYSI GLDIGTNSVG WAVITDEYKV PSKKFKVLGN TDRHSIKKNL IGALLFDSGE TAEATRLKRT ARRRYTRRKN RICYLQEIF SNEMAKVDDS FFHRLEESFL VEEDKKHERH PIFGNIVDEV AYHEKYPTIY HLRKKLVDST DKADLRLIYL A LAHMIKFR GHFLIEGDLN PDNSDVDKLF IQLVQTYNQL FEENPINASG VDAKAILSAR LSKSRRLENL IAQLPGEKKN GL FGNLIAL SLGLTPNFKS NFDLAEDAKL QLSKDTYDDD LDNLLAQIGD QYADLFLAAK NLSDAILLSD ILRVNTEITK APL SASMIK RYDEHHQDLT LLKALVRQQL PEKYKEIFFD QSKNGYAGYI DGGASQEEFY KFIKPILEKM DGTEELLVKL NRED LLRKQ RTFDNGSIPH QIHLGELHAI LRRQEDFYPF LKDNREKIEK ILTFRIPYYV GPLARGNSRF AWMTRKSEET ITPWN FEEV VDKGASAQSF IERMTNFDKN LPNEKVLPKH SLLYEYFTVY NELTKVKYVT EGMRKPAFLS GEQKKAIVDL LFKTNR KVT VKQLKEDYFK KIECFDSVEI SGVEDRFNAS LGTYHDLLKI IKDKDFLDNE ENEDILEDIV LTLTLFEDRE MIEERLK TY AHLFDDKVMK QLKRRRYTGW GRLSRKLING IRDKQSGKTI LDFLKSDGFA NRNFMQLIHD DSLTFKEDIQ KAQVSGQG D SLHEHIANLA GSPAIKKGIL QTVKVVDELV KVMGRHKPEN IVIEMARENQ TTQKGQKNSR ERMKRIEEGI KELGSQILK EHPVENTQLQ NEKLYLYYLQ NGRDMYVDQE LDINRLSDYD VDHIVPQSFL KDDSIDNKVL TRSDKNRGKS DNVPSEEVVK KMKNYWRQL LNAKLITQRK FDNLTKAERG GLSELDKAGF IKRQLVETRQ ITKHVAQILD SRMNTKYDEN DKLIREVKVI T LKSKLVSD FRKDFQFYKV REINNYHHAH DAYLNAVVGT ALIKKYPKLE SEFVYGDYKV YDVRKMIAKS EQEIGKATAK YF FYSNIMN FFKTEITLAN GEIRKRPLIE TNGETGEIVW DKGRDFATVR KVLSMPQVNI VKKTEVQTGG FSKESILPKR NSD KLIARK KDWDPKKYGG FDSPTVAYSV LVVAKVEKGK SKKLKSVKEL LGITIMERSS FEKNPIDFLE AKGYKEVKKD LIIK LPKYS LFELENGRKR MLASAGELQK GNELALPSKY VNFLYLASHY EKLKGSPEDN EQKQLFVEQH KHYLDEIIEQ ISEFS KRVI LADANLDKVL SAYNKHRDKP IREQAENIIH LFTLTNLGAP AAFKYFDTTI DRKRYCSTKE VLDATLIHQS ITGLYE TRI DLSQLGGD |
-分子 #3: Single-guide RNA
| 分子 | 名称: Single-guide RNA / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 32.787355 KDa |
| 配列 | 文字列: GGCUGCGUAU UUCUACUCUG UUGUUUUAGA GCUAGAAAUA GCAAGUUAAA AUAAGGCUAG UCCGUUAUCA ACUUGAAAAA GUGGCACCG AGUCGGUGCU UCG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: 25mA | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X